Филогенетические деревья.
1. Укоренение в среднюю точку.
Дерево, построенное в задании 4 предыдущего практикума с помощью Jalview методом "Neighbor Joining Using % Identity", необходимо было укоренить в среднюю точку.
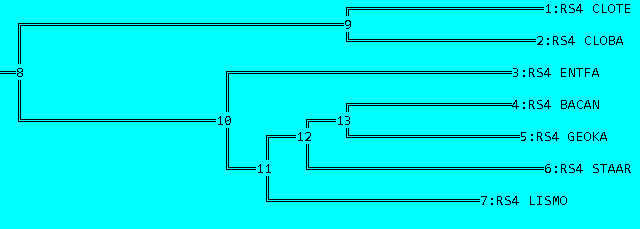
Так как программа MEGA отказывается переукоренить дерево, если оно было построено Jalview, был использован пакет retree программы PHYLIP (команда "Midpoint root the tree"). Метод укоренения основан на поиске максимального расстояния между двумя листьями и размещении корня в середине данного пути.
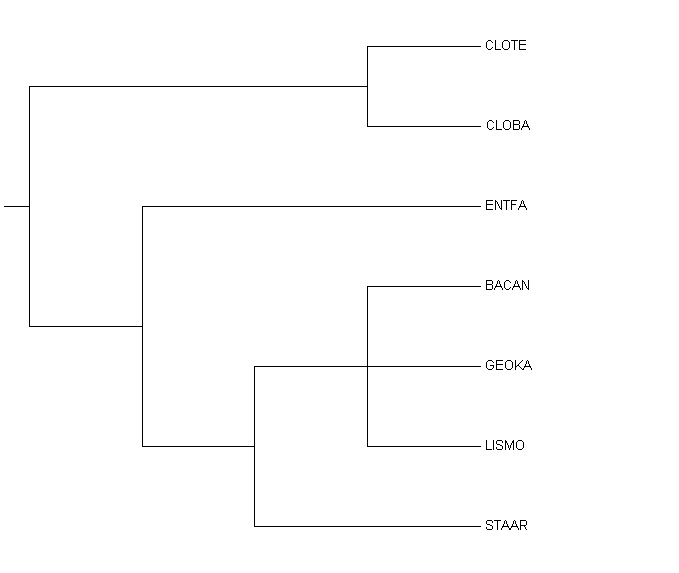
Рис. 1а. Эталонное дерево.
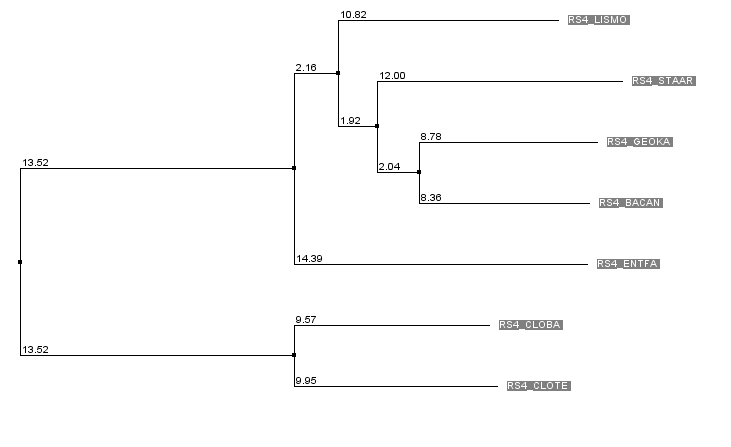
Рис. 1б. Дерево, построенное в Jalview.
Рис. 1в. Дерево, укорененное PHYLIP.
Сравнение реального дерева (рис. 1а) и деревьев, построенного Jalview (рис. 1б) и укорененного PHYLIP (рис. 1в).
2. Использование внешней группы.
Деревья, построенные методом максимальной экономии ("Maximum parsimony") невозможно укоренить в среднюю точку, так как этот метод не реконструирует расстояния (ветви не имеют длины). Однако можно воспользоваться укоренением с помощью внешней группы.
В качестве внешней группы был взят белок того же семейства (RS4) из кишечной палочки (Escherichia coli, мнемоника ECOLI в Uniprot).
По полученному новому выравниванию методом максимальной экономии ("Maximum parsimony") построили дерево. Укоренение произошло по тривиальной ветви {ECOLI} против нетривиальной {CLOTE,CLOBA,ENTFA,LISMO,STAAR,BACAN,GEOKA}.
После удаления аутгруппы получили дерево, представленное ниже.
Укоренение произошло в ветвь {CLOBA,CLOTE} vs {ENTFA,LISMO,STAAR,BACAN,GEOKA}, что согласуется с эталонным деревом. Однако появляются и новые ветви:
{LISMO,ENTFA} vs {CLOBA,CLOTE,STAAR,BACAN,GEOKA} (этой ветви нет также в дереве, построенном с помощью Jalview NJ, и в дереве, переукорененном в среднюю точку)
{STAAR,BACAN,GEOKA} vs {LISMO,ENTFA,CLOBA,CLOTE}
{BACAN,GEOKA} vs {STAAR,LISMO,ENTFA,CLOBA,CLOTE}Очевидно, что анализ взаимосвязи между организмами некорректно проводить по одному белку.
3. Bootstrap.
Смысл анализа бутстрэп заключается в том, чтобы построить дерево по половине данных и затем сравнить результаты от по-разному выбранных половин.
Для этого из входного выравнивания делается много (например, 100) так наз. «бутстрэп-реплик». Каждая бутстрэп-реплика получается в результате случайного удаления половины столбцов из выравнивания с заменой их копиями других (тоже случайно выбранных) столбцов.
Для выполнения задания в программе MEGA было построено дерево с применением бустрэпа (Test of Phylogeny → Bootstrap method(100)) методом UPGMA. Результаты представлены на рисунке.
Топология оригинального и консенсусного деревьев одинакова и практически совпадает с эталонным деревом (и полностью совпадает с деревом, полученным построением в JV NJ).
Нетривиальные ветви:
{CLOBA,CLOTE} vs {ENTFA,LISMO,STAAR,BACAN,GEOKA}
{ENTFA,CLOBA,CLOTE}vs {LISMO,STAAR,GEOKA,BACAN}
{STAAR,GEOKA,BACAN} vs {LISMO,ENTFA,CLOBA,CLOTE}
{GEOKA,BACAN} vs { STAAR,LISMO,ENTFA,CLOBA,CLOTE}
Поддержка бутстрэпа, для нетривиальных ветвей, сходных с ветвями эталонного дерева, достаточна высокая (100 и 92).
.png)