Evolutionary trees.
1.Построение дерева по нуклеотидным последовательностям.
Для каждой из бактерий, отобранных в практикуме 1,были получены последовательность 16S рибосомальной РНК.Из файлов .frn из базы полных геномов NCBI для каждой бактерии было отобрано по одной последовательности, кодирующей субъединицу 16S, и был составлен sequence16S.fasta-файл.
Отобранные последовательности были выровнены в Jalview алгоритмом Muscle (выравнивание в формате fasta), а затем по ним было построено дерево в программе MEGA методом Neighbour Joining. Изображения выравнивания и дерева представлены ниже.
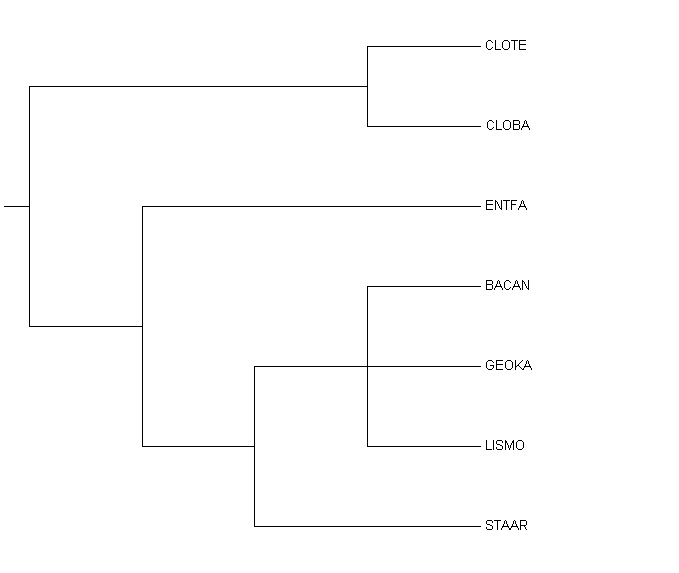
Рис.1а. Дерево, построенное в Jalview по последовательностям рибосомальной РНК.
Рис.1б. Эталонное дерево.
Топология дерева, построенного по рибосомальной РНК (4 нетривиальных ветви):
{CLOBA,CLOTE} vs {GEOKA,BACAN,STAAR,ENTFA,LISMO}
{BACAN,STAAR} vs {LISMO,ENTFA,GEOKA,CLOTE,CLOBA}
{LISMO,ENTFA} vs {BACAN,STAAR,GEOKA,CLOTE,CLOBA}
{LISMO,ENTFA,BACAN,STAAR} vs {GEOKA,CLOBA,CLOTE}Сравнивания данное дерево с эталонным, отмечаем, что совпадает лишь одна нетривиальная ветвь (выделена курсивом), укоренение тем не менее происходит верно. С одной стороны, гены, кодирующие рибосомальные РНК, отличаются повышенной консервативностью, и поэтому реконструкция в данном случае должна быть более точна. Однако же этого не происходит. Построение дерева другими методами также не приближает его к эталону.
2. Построение и анализ дерева, содержащего паралоги.
В выбранных бактериях были найдены гомологи белка CLPX_BACSU. Для этого был создан единый файл proteomes.fasta, содержащий протеомы отобранных бактерий.
Далее на основе протеомов была создана база данных,
makeblastdb –in proteomes.fasta –dbtype prot –out db.fastaпо которой производился поиск гомологов с порогом evalue = 0.001.
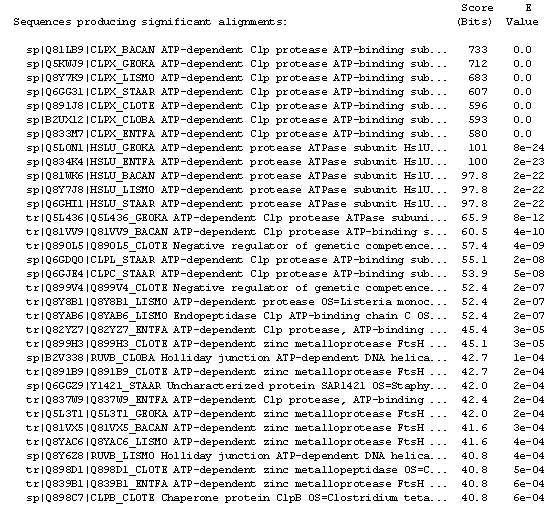
blastp –query CLPX_BACSU.fasta –evalue 0.001 –db dbprot.fasta –out resultВсего было найдено 33 гомолога (рис.2)
Рис.2. Список найденных гомологов.
Далее последовательности были выровнены в Jalview алгоритмом Muscle (рис.3).

Рис.3.Выравнивание.
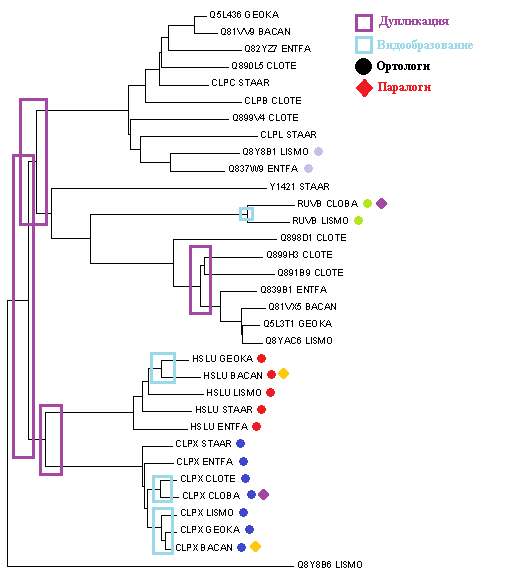
В программе MEGA было построено дерево гомологов белка CLPX_BACSU (рис. 4,5). На рисунке 5 отмечены некоторые ортологи, паралоги и эволюционные процессы.
Рис.4.
Рис.5.