Поиск сигналов. Теория.
1.Определение биологической роли транскрипционного фактора в бактерии.
Для работы я выбрала бактерию Lactococcus lactis subsp. lactis Il1403. Файл с участками ДНК, с которыми связывается ТФ: MurR — Streptococcaceae.
1.1. MEME
Сначала было необходимо найти мотивы связывания с помощью программы МЕМЕ (Multiple EM for Motif Elicitation). MEME – это алгоритм и одноимённый инструмент, являющийся реализацией алгоритма, для поиска мотивов в биологических последовательностях белков и ДНК. Алгоритм основан на многократном применении метода максимального правдоподобия. Под мотивом понимается короткая последовательность нуклеотидов или аминокислот, общая для некоторого набора последовательностей.
Описание определенного МЕМЕ сайта.
A C G T
0.000000 0.000000 1.000000 0.000000
1.000000 0.000000 0.000000 0.000000
1.000000 0.000000 0.000000 0.000000
1.000000 0.000000 0.000000 0.000000
0.000000 0.000000 0.000000 1.000000
1.000000 0.000000 0.000000 0.000000
0.500000 0.000000 0.000000 0.500000
0.000000 0.000000 0.000000 1.000000
1.000000 0.000000 0.000000 0.000000
0.500000 0.000000 0.000000 0.500000
0.000000 0.000000 0.000000 1.000000
0.000000 0.000000 0.000000 1.000000
0.000000 1.000000 0.000000 0.000000
0.250000 0.000000 0.500000 0.250000
0.000000 0.000000 0.000000 1.000000
0.000000 0.000000 0.000000 1.000000
0.000000 0.000000 0.250000 0.750000
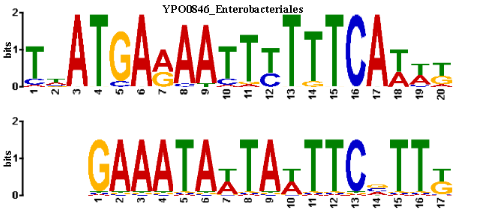
Паттерн:GAAATAWTAWTTCVTTK
E-value: 7.0e-003
PWM: ссылка на файл
Длина мотива: 17
МЕМЕ нашел мотив длиной в 17 нуклеотидов, при том что исходные последовательности были длиной 21 н.о. Стоит отметить, что 13 позиций из 17 консервативны, e-value также очень маленький, в связи с этим можно предположить, что полученный результат достоверен.
1.2. Tomtom
Далее при помощи сервиса Tomtom среди других бактерий был найден похожий по PWM мотив, предсказанный для указанного выше ТФ в базе RegTransBase. Tomtom сравнивает заданный мотив с известными мотивами из базы данных.
Поиск производился по БД RegTransBase >> Prokaryote DNA. На вход был подан найденный мотив в МЕМЕ.
Информация о лучшей находке:
Учитывая, то e-value на самом деле не такой уж и низкий, говорить о правдоподобности находки достаточно тяжело. Также отметим, что и позиции в мотиве сильно отличаются.A C G T
0.000000 0.000000 1.000000 0.000000
1.000000 0.000000 0.000000 0.000000
1.000000 0.000000 0.000000 0.000000
1.000000 0.000000 0.000000 0.000000
0.000000 0.000000 0.000000 1.000000
1.000000 0.000000 0.000000 0.000000
0.500000 0.000000 0.000000 0.500000
0.000000 0.000000 0.000000 1.000000
1.000000 0.000000 0.000000 0.000000
0.500000 0.000000 0.000000 0.500000
0.000000 0.000000 0.000000 1.000000
0.000000 0.000000 0.000000 1.000000
0.000000 1.000000 0.000000 0.000000
0.333333 0.333333 0.333333 0.000000
0.000000 0.000000 0.000000 1.000000
0.000000 0.000000 0.000000 1.000000
0.000000 0.000000 0.500000 0.500000
Паттерн:GAAATAWTAWTTCVTTK
E-value: 1.52e+00
PWM: ссылка на файл.
Длина мотива: 20
Покрытие: полностью (17)
1.3. FIMO.
Сервис FIMO сканирует БД последовательностей в поисках входного мотива. Для исходного мотива GAAATAWTAWTTCVTTK в файле представлен список находок (всего было найдено 23 штук). Так как нас интересовали лишь ТФ (а они как известно связываются с предпромоторной областью),поиск производили по БД Upstream DB с параметром p-value<e-5. Лучшие находки представлены в таблице ниже.
№
Название последовательности
Старт
Стоп
Цепь
p-value
Совпавшая последовательность
1
NP_267285.1 (ген yleB)
-
8.43e-09
GAAATATTATTTCATTG
2
NP_266423.1 (ген ycgF)
265711
266175
+
6.93e-07
GAAATATTTTTTCATTT
3
NP_266422.1 (ген ycgE)
-
6.93e-07
GAAATATTTTTTCATTT
4
NP_266747.1 (ген rmaJ)
583404
583835
-
2.01e-06
GAAAACATAATTCCTTG
Ссылка на аннотированый геном http://www.genome.jp/dbget-bin/www_bget?refseq+NC_002662
NP_267285.1 – (2-полипренил-3-метил-6-метокси-1,4-бензохинон гидролаза (участвует в биосинтезе кофермента Q(убихинона). Не является ТФ.) белок с неизвестной функцией
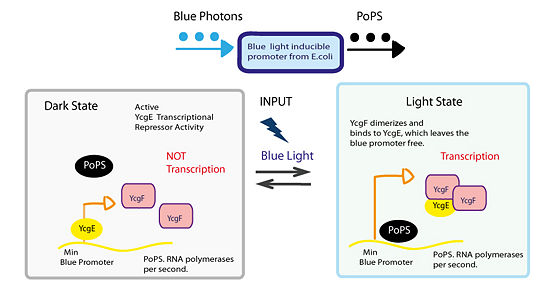
NP_266423.1 – белок с неизвестной функцией у Lactococcus lactis subsp. lactis Il1403. Однако известно, что у Escherichia coli этот белок является фотосенсором и содержит так называемый BLUF домен. Данный белок также содержит EAL-домен, однакоYcgF не связывается с ц-ди-ГМФ и его EAL-домен не обладает светоиндуцированной фосфодиэстеразной активностью. Установлено, что YcgF функционирует как антагонист транскрипционного регулятора YcgE . Действие YcgE в качестве репрессора осуществляется путем его связывания с промоторами в опероне, кодирую щем белки, которые могут активировать вещества матрикса биопленки. Фотовозбужденный YcgF временно формирует гомодимеры, вызывая диссоциацию комплекса YcgE — YcgF и высвобождая репрессор из оперона. Это указывает на фотосенсорную функцию YcgF при модуляции формирования биопленки клетками E. coli.
Ссылка http://vestnik-bio-msu.elpub.ru/jour/article/viewFile/299/292
https://www.ncbi.nlm.nih.gov/pubmed/19240136
NP_266422.1 – S-ribosylhomocysteinase, участвует в метаболизме аминокислот (цистеина и метионина) у Lactococcus lactis subsp. lactis Il1403.NP_266747.1 – транскрипционный фактор; относится к семейству ТФ MarR (Multiple Antibiotic Resistance Regulator). Известно, что например у E. coli, белки MarR регулируют оперон, кодирующий эфллюксный насос для некоторых веществ. Мутации в белках, которые участвуют в этой системе, приводят к развития фенотипа, устойчивого к различным антибиотикам. http://www.cell.com/current-biology/abstract/S0960-9822(13)00016-X
1.4 Поиск регулируемых генов.
Для проверки консервативности геномного окружения генов, регулируемых одним ТФ была использована база данных KEGG (STRING).
Был найден белок NP_267285.1, входящий в ортологический ряд К09963, метаболический путь не указан.
Для белка NP_266423.1 и ТФ L583407, к сожалению, не было найдено ортологического ряда и метаболического пути. Для белка NP_266422.1 был найден метаболический путь 4.4.1.21, относится к ортологическому ряду K07173.2. Влияние метилирования на связывание ТФ со своим сайтом.
Было необходимо найти все сайты, пересекающиеся с предсказанными нами сайтами связывания ТФ, в нашей бактерии, используя fuzznuc из пакета EMBOSS с помощью команды
fuzznuc -sequence X -pattern @MT-sites.pat -outfile Yгде X - входной файл, Y - выходной. Была взята последовательность: мотив (из п.1.3гена rmaJ ) + по 20 нуклеотидов с каждой стороны от него. Также был использован файл с сайтами метилирования
Информация о взятом участке, а также результаты программы представлены в таблице ниже.
Название гена
Исходные координаты мотива
Длина
участкаЧисло найденных пересечений
Выходной файл
rmaJ
583404..583835
57
45
Около половины пересечений состояли всего лишь из одного нуклеотида, что не позволяет сделать каких-либо четких выводов на счет того, влияет ли метилирование гена на его транскрипцию или нет. Самые длинные пересечения (включающие N-позиции) составили 12 нуклеотидов.
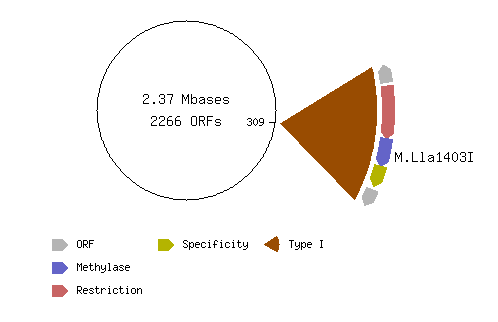
В БД REBASE были найденные закодированные в геноме нашей бактерии метилтрансферазы. На картинке ниже представлена найденная метилтрансфераза M.Lla1403I ( к сожалению, для нее не указан специфический сайт связывания).
Это N-6 аденин-ДНК метилтрансфераза.
http://tools.neb.com/genomes/summary.php?genome_id=111http://meme-suite.org/opal-jobs/appFIMO_4.12.01500806240475-1505087148/fimo.html