Поиск по PDB и содержание PDB файла
1. Поиск атомов с коэффициентом Occupancy, не равным 1
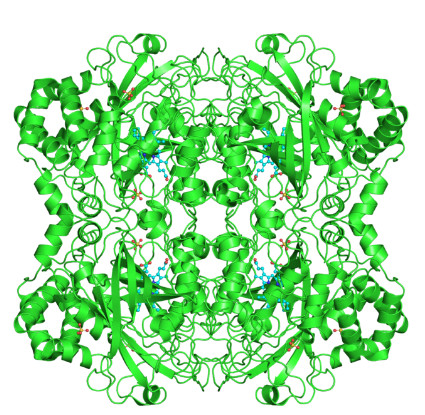
В соответствии с требованиями была взята структура белка Micrococcus Lysodeikticus catalase, PDB ID 1gwe с разрешением 0.88Å. Белок представлен на рисунке 1.
Рис. 1. Micrococcus Lysodeikticus catalase
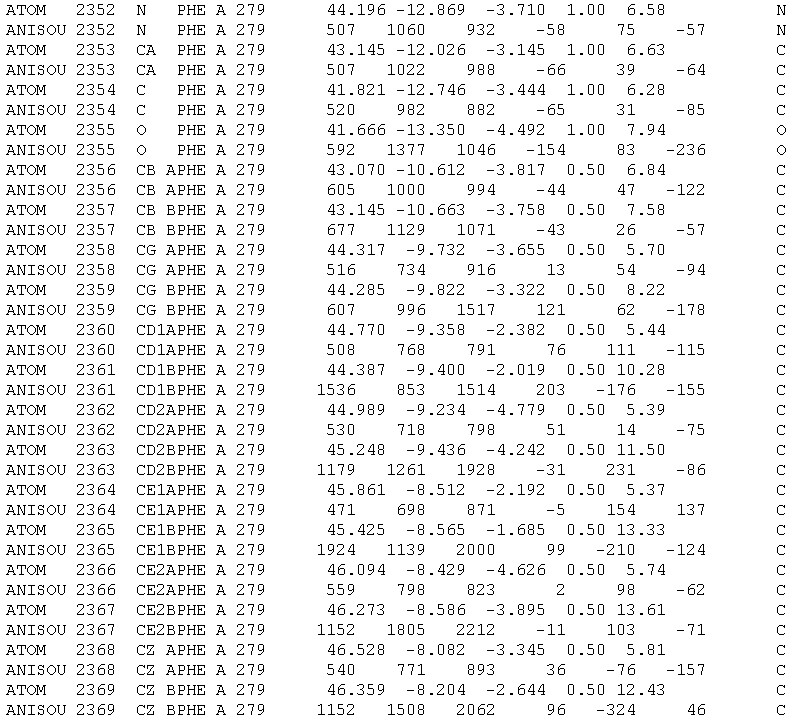
Поиск подходящей структуры был проведен с параметрами Experimental method: x-ray diffraction; Resolution: 0.5 - 1.0 Å. На рисунке 2 представлен фрагмент файла PDB, где коэффициент заполнения не равен единице.
Рис. 2. Фрагмент файла PDB
2. Поиск остатков, которые не удалось расшифровать с помощью РСА
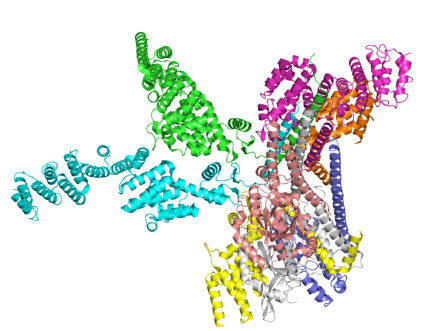
Для поиска Missing residues была найдена структура человеческого COP9 signalosome, PDB ID 4d10, с низким разрешением в 3.8Å. На рисунке 3 представлен данный белок.
Рис. 3. COP9 signalosome
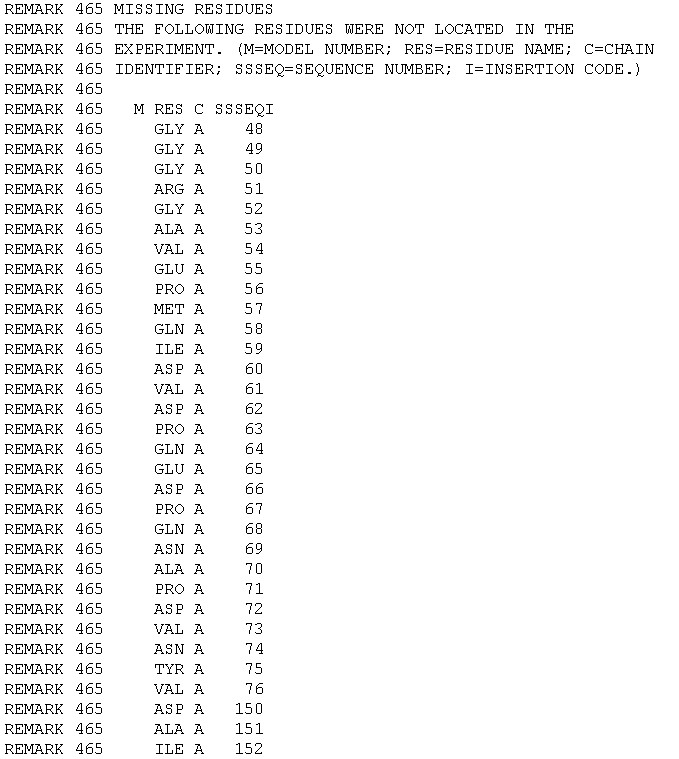
В PDB файле оказалось 721 таких остатков (рис. 4).
Рис. 4. Фрагмент файла PDB с missing residues
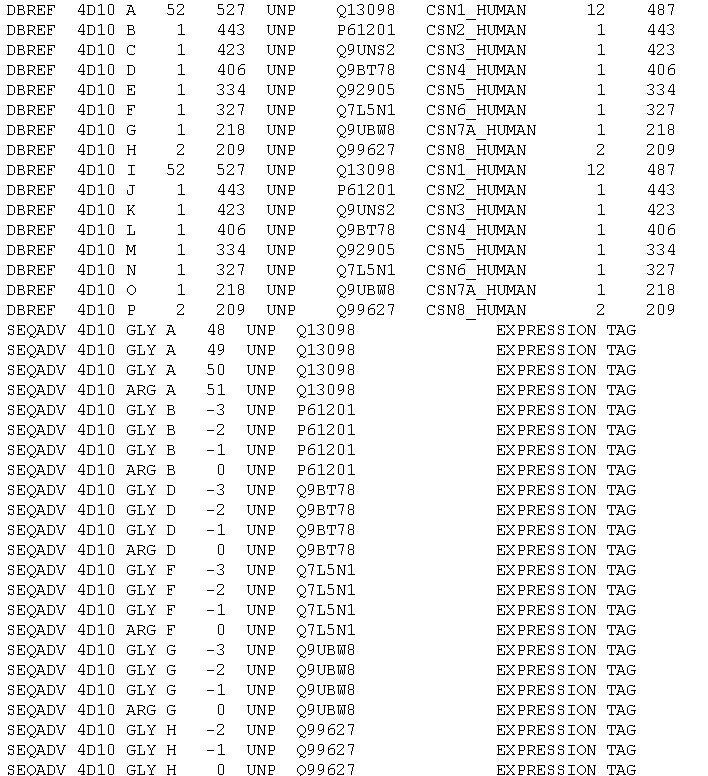
3. Несовпадения последовательностей природного белка из Uniprot и белка, кристаллизованного для РСА
Для этого задания был взят тот же белок, что и для поиска Missing residues.
DBREF – здесь указываются кросс-референсы между данной последовательностью и последовательностями из других БД
SEQADV – показывает различия между последовательностями, описанными в поле DBREF и последовательностью данного pdb-файлаФрагмент файла PDB представлен на рисунке 5.
Рис. 5. Фрагмент файла PDB
4. Температурный фактор
Из-за того, что атом может колебаться и быть по разному расположенным в разных ячейках вводят так называемый B-фактор(температурный фактор), который характеризует дисперсию (здесь размазанность пика вокруг атома). Хорошими являются значения 10-20. На рисунке 6 и 7 оказаны строчки PDB-файла структуры все того же белка COP9 signalosome, в которых прописаны атомы с лучшим и худшим B-фактором.
. Рис. 6. Лучший В-фактор
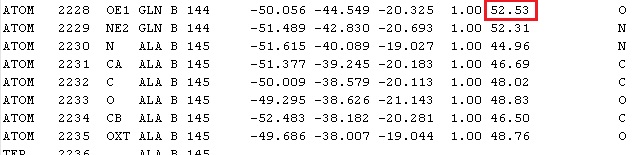
Рис. 7. Худший В-фактор