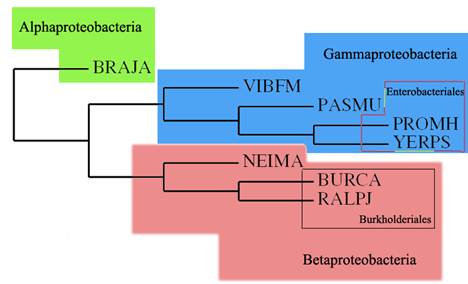
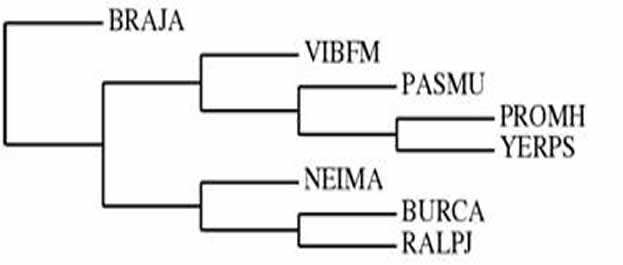
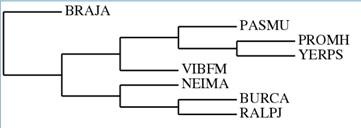
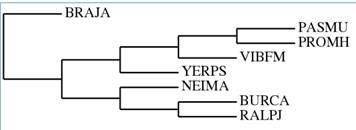
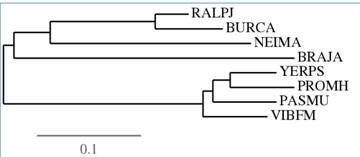
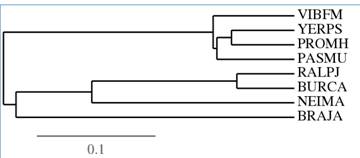
Задание 2 (выполнено Борисовой Мариной)
1.
|
Название |
Мнемоника |
Таксономия |
|
Bradyrhizobium japonicum |
BRAJA |
Alphaproteobacteria; Rhizobiales |
|
Burkholderia cenocepacia |
BURCA |
Betaproteobacteria; Burkholderiales |
|
Ralstonia pickettii |
RALPJ |
Betaproteobacteria; Burkholderiales |
|
Neisseria meningitidis |
NEIMA |
Betaproteobacteria; Neisseriales |
|
Yersinia pseudotuberculosis |
YERPS |
Gammaproteobacteria; Enterobacteriales |
|
Proteus mirabilis |
PROMH |
Gammaproteobacteria; Enterobacteriales |
|
Pasteurella multocida |
PASMU |
Gammaproteobacteria; Pasteurellales |
|
Vibrio fischeri |
VIBFM |
Gammaproteobacteria; Vibrionales |
2. Последовательности белков
|
Функция |
Мнемоника |
|
Энолаза |
ENO |
3. файл после обработки muscle
4. диагностические позиции (файл):
| Позиция | Betaproteobacteria | Gammaproteobacteria |
| 3 | A (+BRAJA) | K |
| 6 | D (+BRAJA) | K |
| 23 | C | A |
| 24 | D (+BRAJA) | E |
| 26 | L | H |
| 28 | S | - |
| 31 | - | F |
| 37 | V (+BRAJA) | A |
| 70 | H | A (+BRAJA) |
| 86 | E | - |
| 90 | L | I |
| 106 | L (+BRAJA) | F |
| 116 | M | L (+BRAJA) |
| 118 | V | - |
| 124 | E | A |
| 126 | - | K |
| 134 | - | I |
| 137 | - | L |
| 138 | - | N |
| 140 | G (+BRAJA) | T |
| 144 | M | - |
| 148 | V (+BRAJA) | L |
| 161 | N | D (+BRAJA) |
| 163 | S | N |
| 164 | L | V |
| 179 | F (+BRAJA) | L |
| 180 | F | K |
| 185 | C | - |
| 192 | A | - |
| 194 | K (+BRAJA) | A |
| 196 | - | V |
| 220 | - | A |
| 227 | V,L | - |
| 230 | - | V |
| 237 | A (+BRAJA) | L |
| 239 | E | K |
| 242 | L | T |
| 245 | L (+BRAJA) | M |
| 269 | - | F |
| 276 | - | H |
| 302 | W (+BRAJA) | F |
| 305 | L (+BRAJA) | Q |
| 311 | - | D |
| 344 | - | F |
| 349 | T (+BRAJA) | S |
| 358 | D, E | K |
| 362 | R | D |
| 379 | S (+BRAJA) | A |
| 389 | N (+BRAJA) | A |
| 398 | L (+BRAJA) | M |
| 411 | L (+BRAJA) | I |
| 416 | - | A |
| 420 | - | K,R |
| 422 | - | P |
| 423 | Y | F |
| 424 | P | - |
| 426 | K (+BRAJA) | L |
| 427 | - | K |
| 429 | F | V |
| 430 | Y | K (+BRAJA) |
| 431 | - | G |
| 432 | L (+BRAJA) | Q |
| Всего | 45 | 54 |