Изучение BLAST
Поиск гомологов белка ACF11151.1 в бд swissprot
Поиск проводился с помощью разновидности программы BLAST -BLASTp с использованием следующих параметров:
- Database:"swissprot" (выбираем бд, в которой будем искать гомологичные последовательности);
- Общие параметры:
-Max target sequences:"20000" (выбираем, сколько выравненных последовательностей выведет BLAST; если гомологичных(по мнению BLASTa) последовательностей окажется больше, то программа будет выводить те последовательности,которые в выравнивнивании с query будут иметь наибольший вес);
-Expect threshold:"10" (выбираем порог на E-value; E-value - это математическое ожидание количества случайных последовательностей в банка рандомных последовательностей такого же размера, как и тот в котором ищем мы,выравнивание которой с query имеет вес такой же или больше, чем выравнивание query и subject; таким образом близкое к нулю значение e-value говорит о том, что две последовательности гомологичны и имеют высокий процент идентичности);
-Word size:"6" (длина фрагментов query, с которыми BLAST будет строить выравнивания, чтобы найти гомологичные последовательности);
-Max matches in a query range:"0" (выбираем максимально допустимое число находок BLAST в одной последовательности; если выбрали 0, то ограничений нет); - Параметры веса выравнивания:
-Matrix:"BLOSUM62"(выбираем, по какой матрице будем присваивать вес заменам аминокислот в выравнивании);
-Gap costs:"Existence: 11 Extension:1"(выбираем штраф за гэпы и индели; в моём случае 11 за появление инделя и 1 за каждый следующий гэп в инделе);
-Compositional adjustment:"Conditional compositional score matrix adjustment" (выбираем, как будем бороться с участками малой сложности).
Множественное выравнивание последовательностей
Из таблицы находок BLAST были выбраны 6 последовательностей:
- Q59334.1-Delta-aminolevulinic acid dehydratase из организма Chlorobaculum parvum NCIB 8327
- Q8KCJ0.1-Delta-aminolevulinic acid dehydratase из организма Chlorobaculum tepidum TLS
- P43087.2-Delta-aminolevulinic acid dehydratase из организма Synechococcus elongatus PCC 7942
- Q9SFH9.1-Delta-aminolevulinic acid dehydratase 1 из организма Arabidopsis thaliana
- P42504.1-Delta-aminolevulinic acid dehydratase из организма Rhodobacter capsulatus
- P10518.1-Delta-aminolevulinic acid dehydratase из организма Mus musculus
- P13716.1-Delta-aminolevulinic acid dehydratase из организма Homo sapiens
Было построено множественное выравнивание этих последовательностей. Из полученного выравнивания можно сделать вывод о том, что рассмотренные белки гомологичны, т.к. в выравнивании имеется 2 участка, начинающихся и заканчивающихся абсолютно консервативной позицией, с длинной более 6 а.о., без колонок с гэпами и с высокой плотностью консервативных позиций.
Участки множественного выравнивания
.png)
Карта локального сходства
С помощью программы BLASTP была построена карта локального сходства двух альдолаз дигидронеоптерина S8FKT8_FOMPI и S7RMD1_GLOTA из организмов двух агарикоидных грибов: Fomitopsis pinicola и Gloeophyllum trabeum соответственно. На горизонтальной оси находится последовательность S7RMD1_GLOTA, а на вертикальной S8FKT8_FOMPI
Карта локального сходства
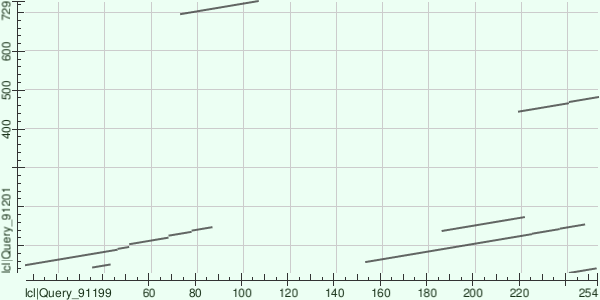
У S8FKT8_FOMPI произошли дупликации участка последовательности, мелкие делеции и одна довольно крупная транслокация. У S7RMD1_GLOTA имеется несколько участков которые у S8FKT8_FOMPI представлены несколько раз, но не рядом, а в разных частях последовательности (т.е. это либо дупликация с последующей транслокацией, либо непонятно что).
Игры с BLAST
Я взяла такую последовательность:"isawthecakeitwasverytastyandbeautifulandideceideditisanidealandveryverytasty" и пыталась с помощью BLAST найти её гомологов в бд swissprot.
Первый сеанс поиска производился со стандартными параметрами(как в задании 1, только Max target sequences был 100 и
была отключена опция "Automatically adjust parameters for short input sequence"), в результате этого сеанса гомологи не обнаружились. Далее я изменила параметр Word size на "3" и "2",
в обоих случаях нашлась одна последовательность со следующими значениями:

Единственное отличие двух сеансов - во времени работы, так сеанс с параметром Word Size "2" работал дольше. Это понятно ведь данный параметр задаёт длину фрагментов query,
с которыми BLAST будет строить выравнивания, чтобы найти гомологичные последовательности, следовательно, чем короче фрагмент, тем больше выравниваний будет строиться и
тем больше времени тратиться.
Ещё один сеанс поиска я провела со следующими параметрами: Word size:"2" и Matrix:"BLOSUM45", остальные параметры по умолчанию, и получила следующие результаты:

Т.е. в этом случае нашлись 4 последовательности, правда все с довольно большим значением E-value.Это говорит о том, что найденные последовательности, вероятнее просто случайность, чем действительно гомологичные.
Также у полученных выравнивания довольно низкий вес,но в данном случае это не очень важно, поскольку query сама по себе короткая.