Сборка de novo
1.Подготовка чтений программой trimmomatic
Команда - 'java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240380.fastq trim_reads.fastq ILLUMINACLIP:adapters.fasta:2:7:7'
Результат - 'Input Reads: 5217318 Surviving: 5119143 (98,12%) Dropped: 98175 (1,88%)'
Команда - 'java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 trim_reads.fastq trim2_reads.fastq TRAILING:20 MINLEN:32'
Результат - 'Input Reads: 5119143 Surviving: 4865356 (95,04%) Dropped: 253787 (4,96%)'
2.Подготовка k-меров длины k=31 программой velveth
Команда - ' velveth 'velveth' 31 -fastq -short trim2_reads.fastq'
Результат - 3 файла в папке velveth : Log, Roadmaps (k-меры), Sequences (чтения)
3.Сборка на основе k-меров
Команда - 'velvetg 'velveth''
N50= 11628
| контиг(ID) | длина | покрытие |
| 3 | 25915 | 27.418676 |
| 20 | 23850 | 24.763816 |
| 23 | 23807 | 25.725921 |
"Аномальные" контиги
| контиг(ID) | длина | покрытие |
| 11 | 2083 | 126,050408 |
| 57 | 934 | 130,479657 |
| 110 | 86 | 2,651163 |
4.Анализ
1)контиг 3 (ложится на + цепь):
| координаты участка хромосомы | Identity | число гэпов | |
| 1 | 2004 - 11103 | 7229/9221(78%) | 252/9221(2%) |
| 2 | 613658 - 620926 | 5850/7385(79%) | 190/7385(2%) |
| 3 | 621055 - 627104 | 4678/6170(76%) | 240/6170(3%) |
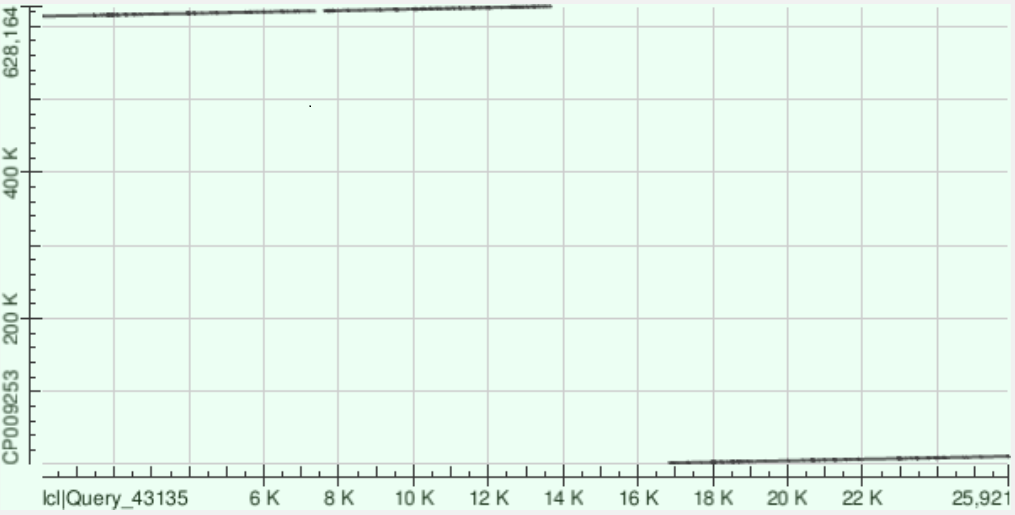
Контиг ложится практически непрерывно (вспоминаем, что хромосома кольцевая).
2)контиг 20 (ложится на '-'цепь):
| координаты участка хромосомы | %Identity | число гэпов | |
| 1 | 236918 - 247596 | 8182/10884(75%) | 391/10884(3%) |
| 2 | 232358 - 236859 | 3466/4581(76%) | 130/4581(2%) |
| 3 | 229411 - 232057 | 2156/2685(80%) | 71/2685(2%) |
| 4 | 248967 - 252161 | 2527/3246(78%) | 94/3246(2%) |
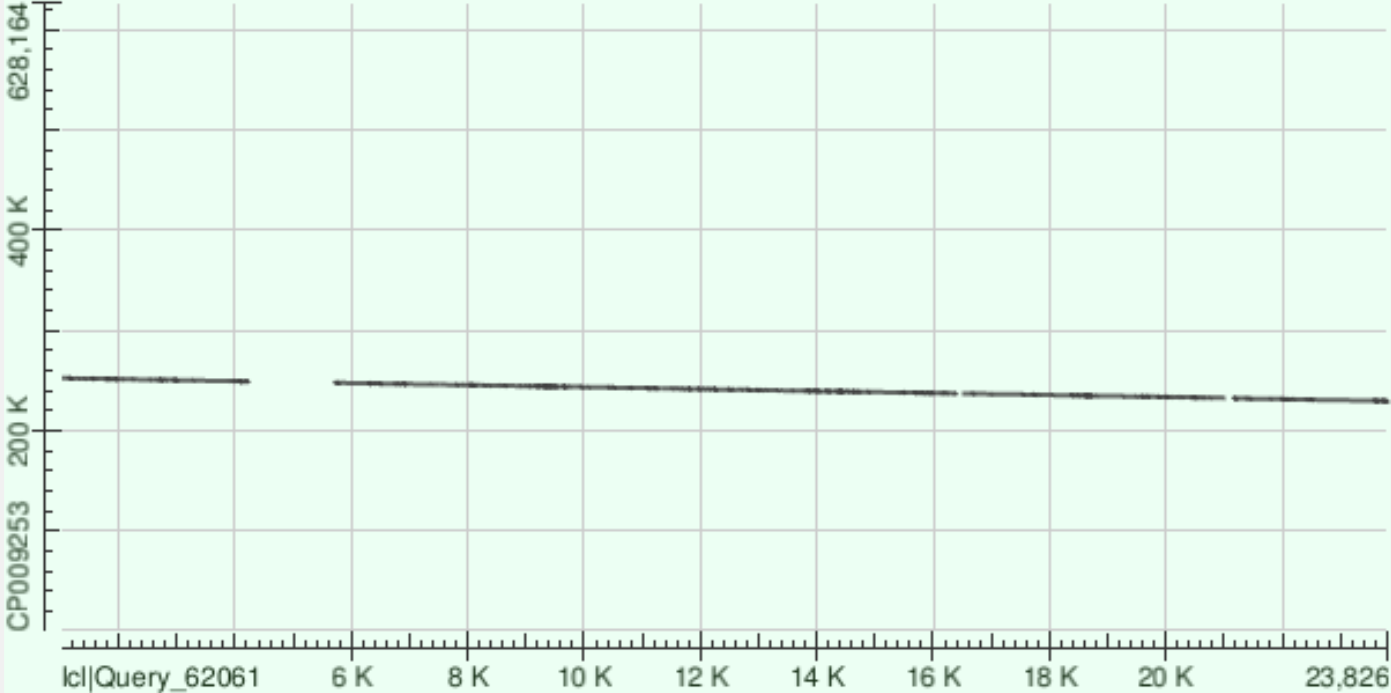
Контиг ложится на хромосому с разрывами, и при этом часть контига никак не выровнялась с хромосомой.
3)контиг 23 (ложится на + цепь):
| координаты участка хромосомы | %Identity | число гэпов | |
| 1 | 573092 - 582686 | 7212/9822(73%) | 461/9822(4%) |
| 2 | 584329 - 587055 | 2100/2777(76%) | 108/2777(3%) |
| 3 | 593743 - 594099 | 289/359(81%) | 4/359(1%) |
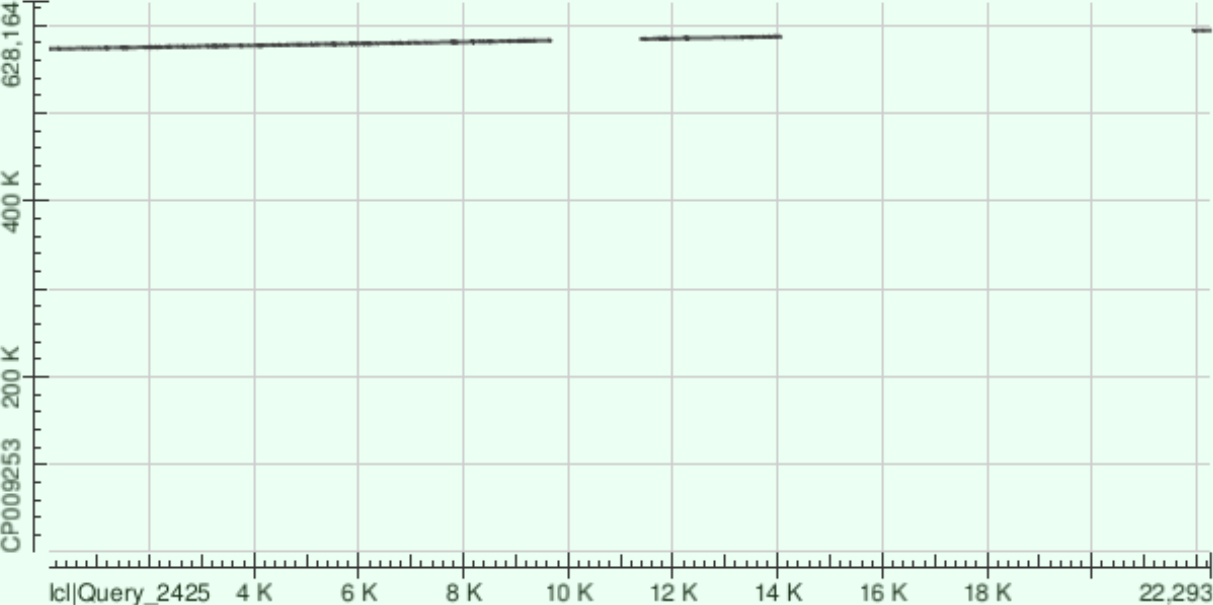
Контиг также ложится с разывами, причём его значительная часть никак не выравнивается с хромосомой.