Секвенирование по Сэнгеру
1.Получение последовательности ДНК на основании данных, полученных из капиллярного секвенатора
исходный файл с прямой последовательностью
исходный файл с обратной последовательностью
Файл с консенсусной последовательностью
Файл с выравниванием последовательностей
Работа с хроматограммами проводилась в программе Chromas, т.к. GeneStudio (по непонятным мне причинам) считал один из исходных файлов пустым,
а также на некоторых участках хроматограммы не отображал последовательность.
В Chromas обратная последовательность была "перевёрнута и сделана комплементарной", потом были скачаны прямая и 'перевёрнутая' обратная последовательности в fasta и выравнены с помощью needle
(выравнивание исходных последовательностей), чтобы было понятно, какие участки ДНК можно проверять с помощью второй последовательности.
Удаление нечитаемых 5' и 3'-концов:Б
В прямой последовательности: 5'-конец - я бы удалила первые 17 нуклеотидов (Chromas убрал 21), 3'-конец - на мой взгляд всё чётко видно (программа убрала 3 последних нукдеотида)
В обратной последовательности (уже развернутой и комплементарной): 5'-конец - я бы ничего не удаляла (Chromas согласен),
3'-конец - на мой взгляд нужно удалить 36 нуклеотидов (программа убрала 88)
В целом хроматограммs довольно хорошо чита.тся, уровень шума низкий (на глаз), редактировались в основном полиморфизмы.
Проблемные нуклеотиды
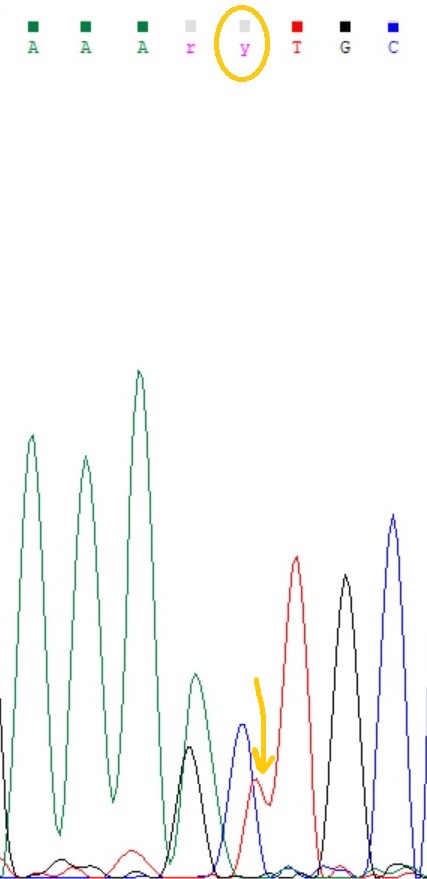
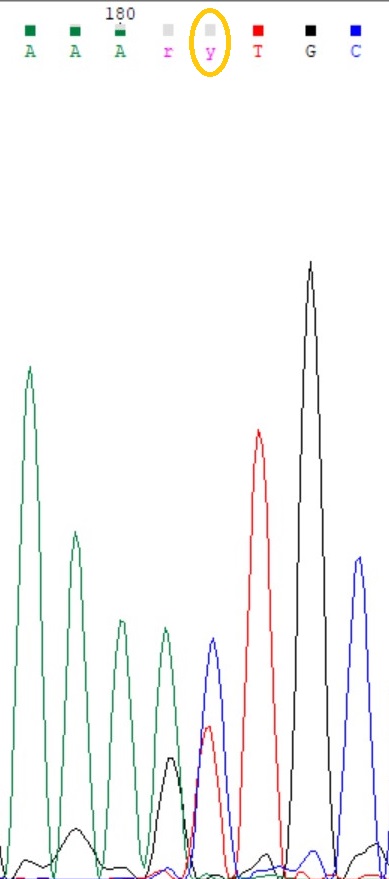
1)На рис.1 изображён участок обратной цепи, выделенный нуклеотид я посчитала проблемным, поскольку не очень понятно к чему относится смещённый пик, это полиморфизм или ошибка фореза. На рис.2 изображён тот же участок но уже прямой цепи, и здесь пик не смещён, т.е. это был полиморфизм(Y). (решение Chromas по данному нуклеотиду - N)
|
|
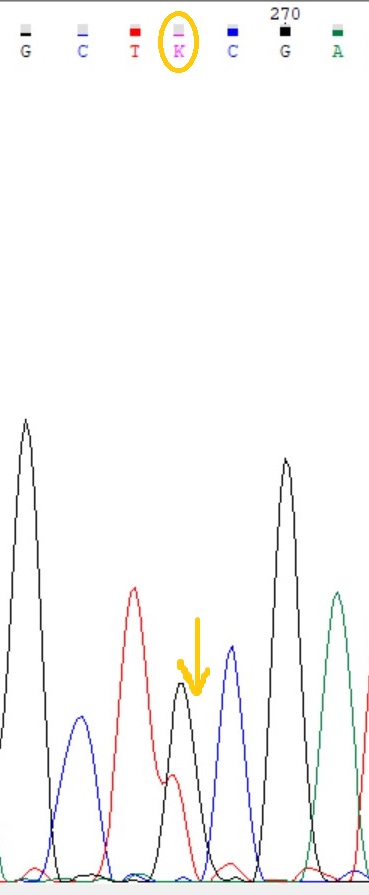
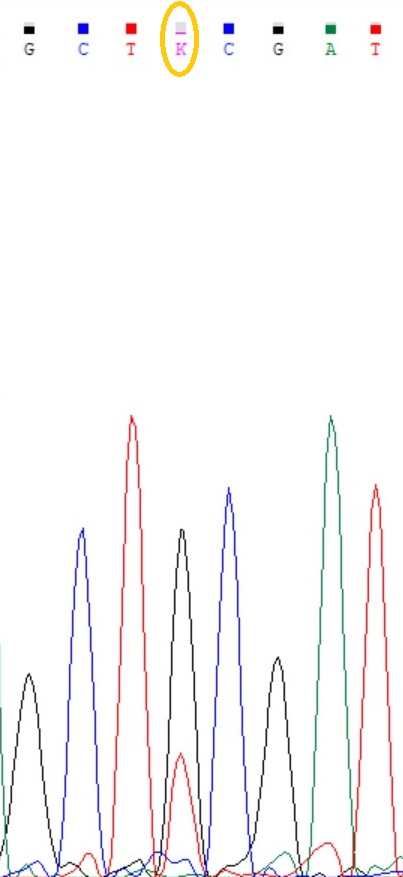
2)На рис.3 изображён участок обратной цепи, ситуация аналогична предыдущей. На рис.4 - тот же участок но уже прямой цепи, пик не смещён, т.е. это был полиморфизм(K). (программа приняла такое же решение)
|
|
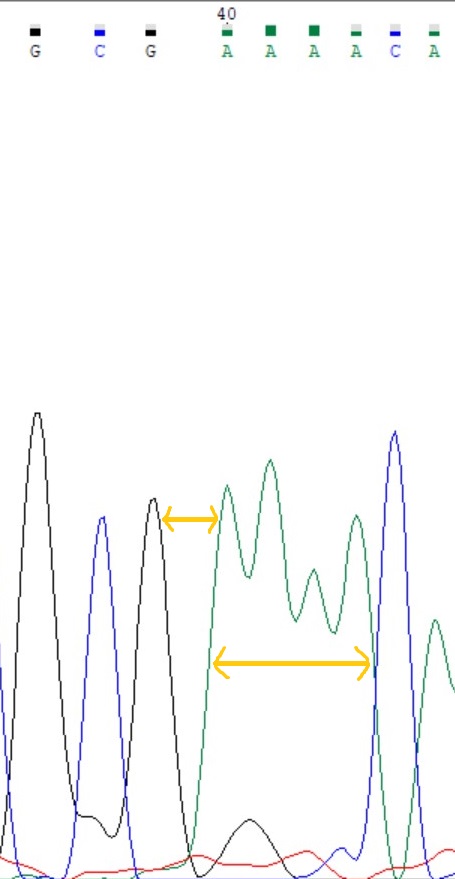
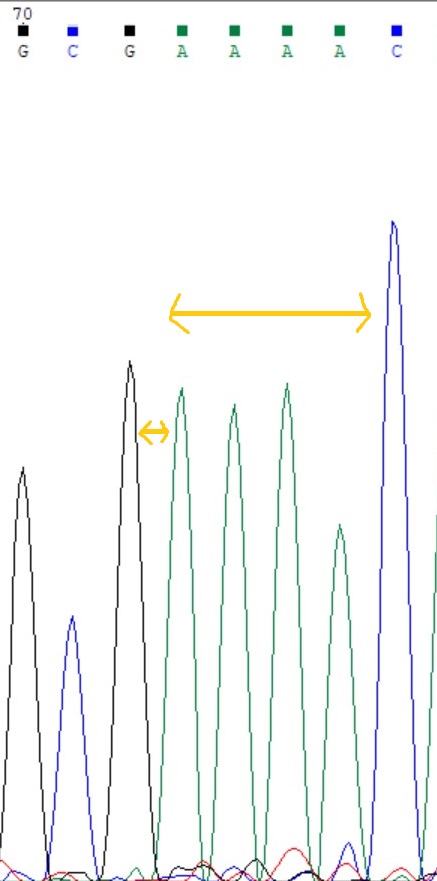
3)4)На рис.5 - участок прямой цепи, здесь сразу два непонятных места. Во-первых, расстояние между пиками G и A больше среднего, возможно там должен стоять нуклеотид. Но нет, см рис.6: пики чёткие и расстояние нормальное. Во-вторых, пики аденинов (рис.5) частично слиты, что наводит на мысль об ошибке. Но опять же смотрим обратную цепь (рис.6) с 4 чёткими пиками аденинов, значит эта неточность фореза не привела к ошибке.
|
|
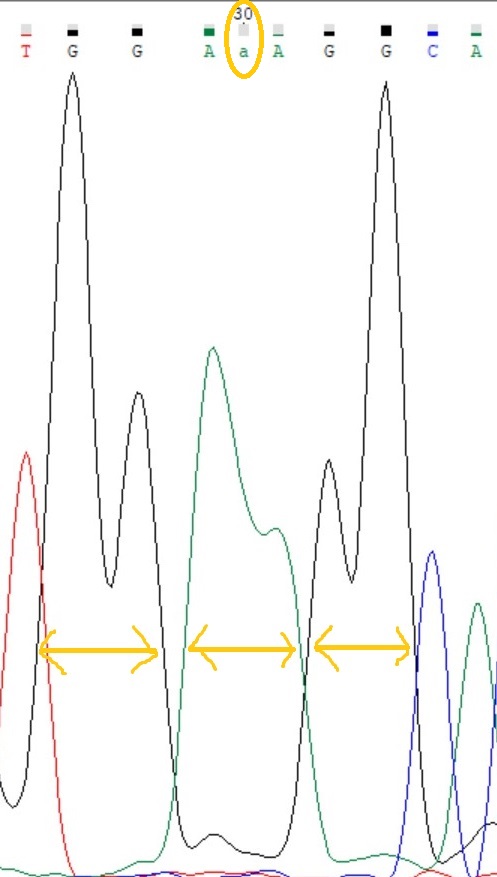
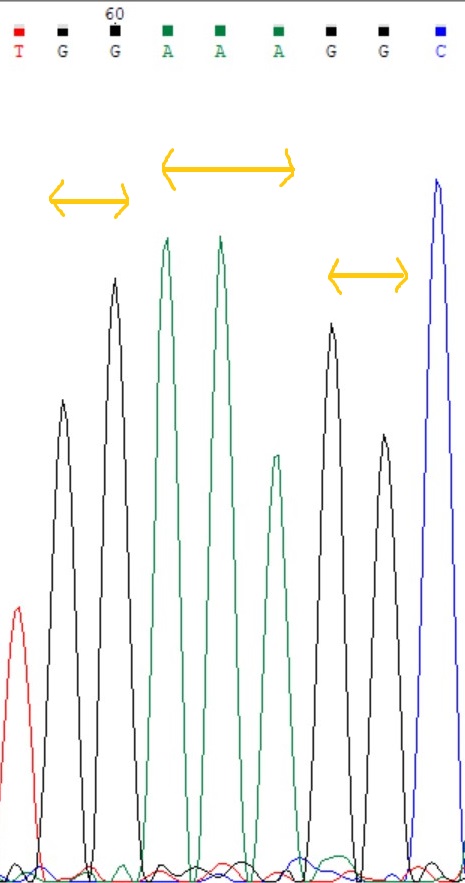
5)На рис.7 - участок прямой цепи, где мы опять видим несколько слившихся пиков, и возможно в ДНК больше нуклеотидов, чем выдала программа.
Смотрим на обратную цепь (рис.8), действительно, один аденин был пропущен.
Неточности на данном участке прямой цепи, не очень удивительны, т.к. он расположен довольно близко к 5'-концу.
|
|
2.Нечитаемый фрагмент хроматограммы
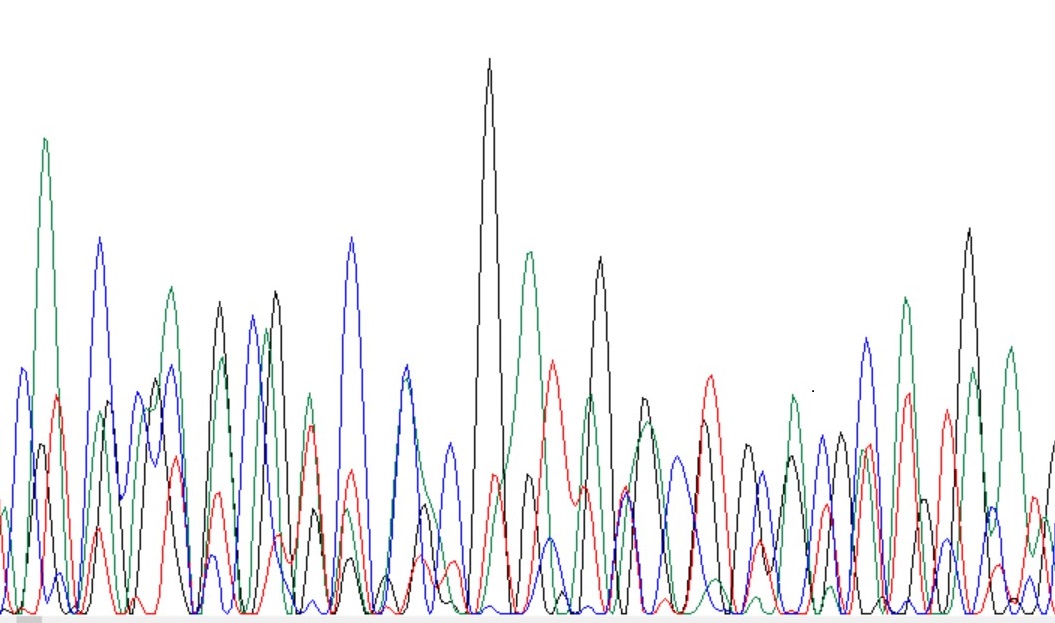
Я выбрала хроматограмму из паки bad/NN_G10.ab1. На рис.9 представлен её участок, Chromas не справился с его прочтением, я тоже, т.к. пики накладываются друг на друга, и совершенно непонятно, какую букву им присвоить. Возможно, наложение пиков происходит, потому что в образце было 2 разные ДНК.