Нуклеотидный BLAST
1.Определение таксономии и функции прочтённой нуклеотидной последовательности
Для поиска гомологичных последовательностей использовался blastn (cоответственно БД-Nucleotide), т.к. неизвестно кодирует ли данная ДНК белок или РНК. Blastn запускался с параметрами по умолчанию, результаты его работы представлены на рис.1.
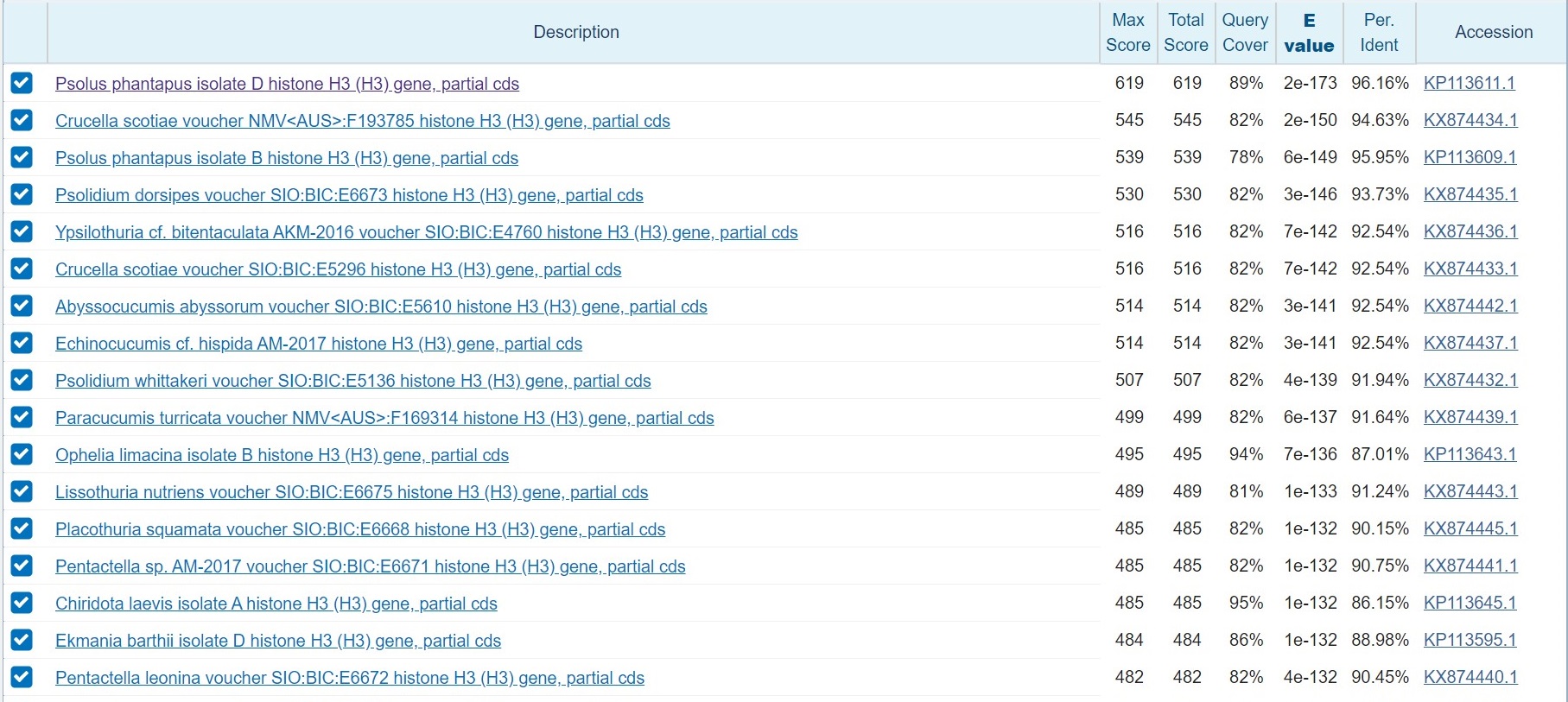
1)Функция последовательности
Данная последовательность кодирует гистон H3, т.к. все последовательности (из выдачи blastn) с хорошим значением E-value и %идентичности делают то же самое.
2)Таксономия последовательности- вид Psolus phantapus
Сначала было построено выравнивание Query и первых восьми находок blastn(рис.2)

Выравнивание выглядит довольно хорошо, но проблема в том,
что из-за этого сложнее становится определить таксономию. Дело в том, здесь выравнивались последовательности нескольких семейств отряда
Dendrochirotida, и тут два пути: остановиться на отряде или выбрать из тех 8 последовательностей
наиболее близкие к Query.
Но в этом случае даже если оставить 2 находки с наибольшим %Identity встаёт вопрос
какое семейство выбрать: Paracucumidae или Psolidae. Ещё больше напрягает,
что перед этим были выброшены две последовательности из сем-ва Psolidae.
При этом если посмотреть на локальное выравнивание Query с самой первой находкой (рис.3),
то можно заметить, что две последовательности отличаются только в одной позиции,
все остальные отличия происходят из-за того, что в Query есть позиции, в которых могут стоять разные нуклеотиды.
Поэтому хочется верить, что последовательность относится к виду Psolus phantapus,
а отличие в одной позиции, это погрешность (хроматографии или её интерпретации)

2.Сравнение списка находок нуклеотидных последовательностей тремя разными вариантами blast
1)последовательность из пункта 1.
Поиск проводился в отряде Dendrochirotida, поскольку поиск в семействе Psolidae, по 2 из 3 алгоритмов выдавал меньше 5 находок. Для этого в поле Entrez Query было введено выражение: "Dendrochirotida[Organism] and not Psolus[Organism]".(ещё из поиска исключается род Psolus, к которому принадлежит последовательность).
| алгоритм | кол-во находок | |
| megablast параметры по умолчанию |
17 | 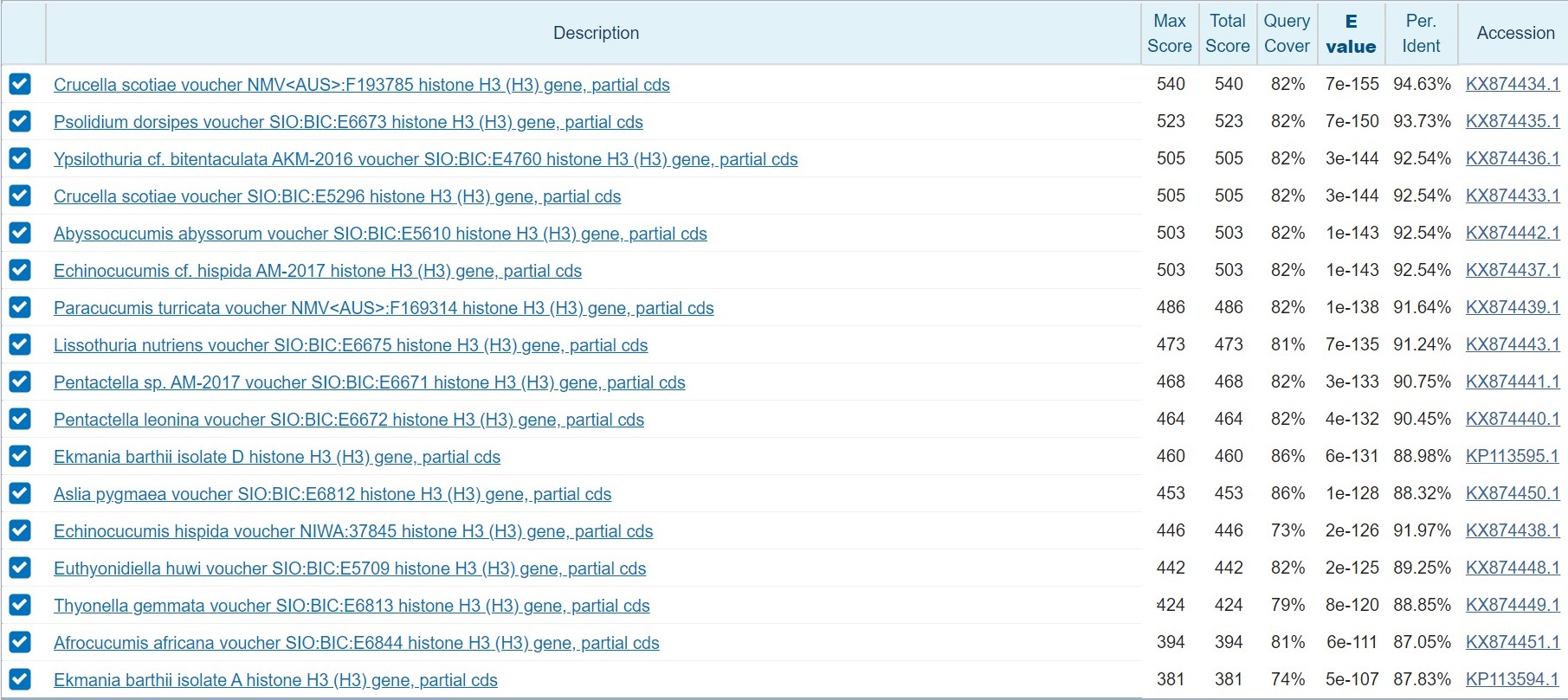 |
| blastn Expect threshold: 0.001 |
25 | .jpg) |
| blastn Word size:7 Match/Mismatch Scores:4,-5 Expect threshold: 0.001 |
25 | .jpg) |
2)CDS белка Escherichia phage vB_EcoP_KAW1A4500
Для алгоритмов blastn был поставлен порог на e-value(меньше 0.001), т.к. без этого ограничения появлялись сомнительные находки.Также из поиска была исключена Query (в поле Organism - exclude)
| алгоритм | кол-во находок | |
| megablast | 15 | 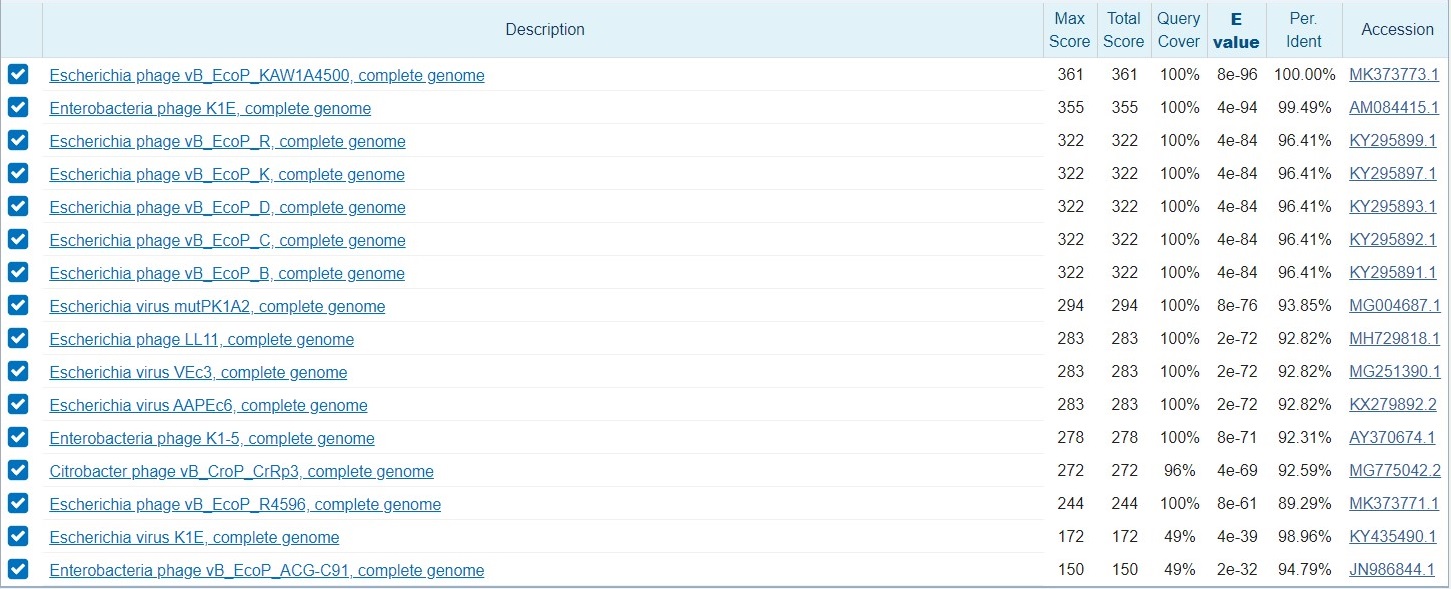 |
| blastn Expect threshold: 0.001 |
24 | .jpg) |
| blastn Word size:7 Match/Mismatch Scores:4,-5 Expect threshold: 0.001 |
26 | .jpg) |
Количество находок megablast меньше, чем у алгоритмов blastn. Что ожидаемо, поскольку параметры поиска megablast жёстче, чем у blastn, благодаря чему мы получаем близких гомологов. Результаты работы blastn с разными параметрами практически не отличаются. В случае с более мягкими параметрами в находках появляются последовательности с очень маленькими e-value.
3.Проверка наличия гомологов трех белков в неаннотированном геноме
Команда для создания нуклеотидной базы: makeblastdb -in X5.fasta -dbtype nucl
Последовательности белков были из получены из UniProt(поиск:"название белка"->этот белок у человека->format->fasta)
Ccылки на последовательности белков: histone h3,
Tubulin alpha-1A chain,
Kinesin-like protein KIF2A
Команда для поиска гомологов: tblastn -query protein_tubuline\(pr8\).fasta -db X5.fasta -out protein_tubuline -outfmt 7
1)Histone H3
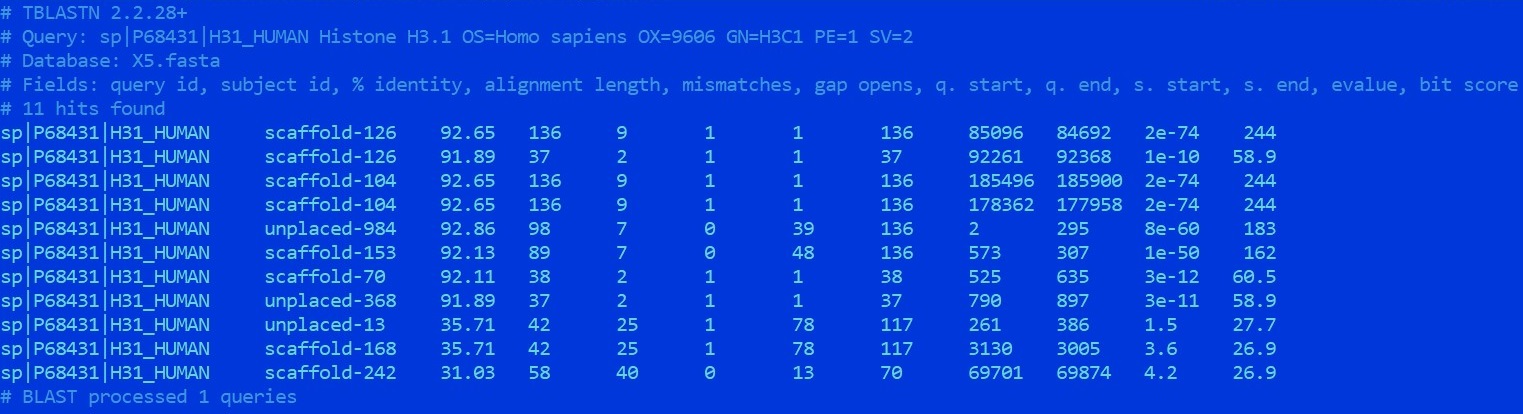
Выбрана последовательность белка histone h3.1 человека. Лучшая
находка blast имеет %Identity=92% и порытие 100%, поэтому у организма Amoeboaphelidium protococcarum
имеется гомолог рассматриваемого белка.
Гистоны- основной компонент нуклеосом, задача которых упаковка ДНК. Т.е. они имеются у всех эукариот и очень
консервативны.
2)Tubulin
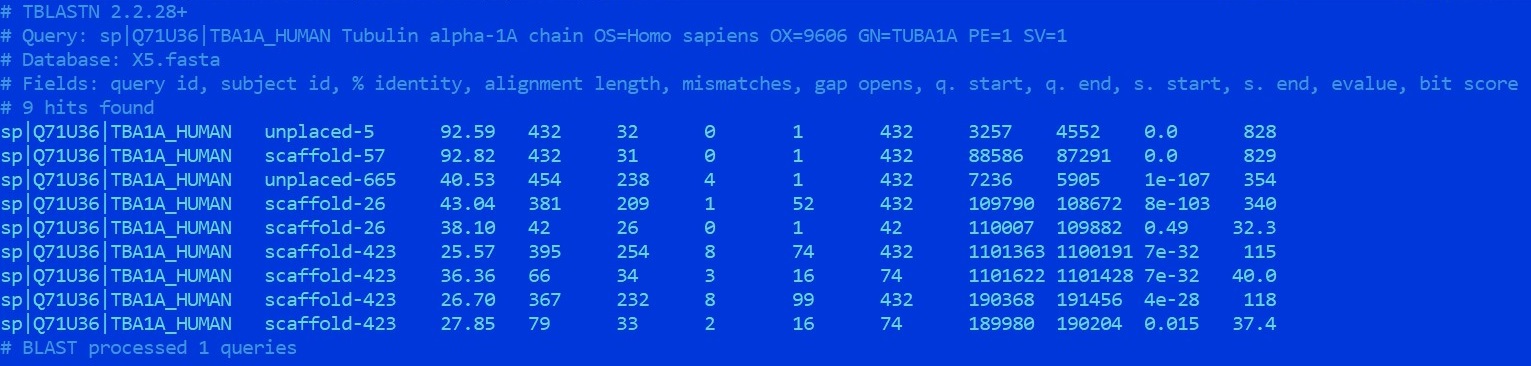
Последовательность альфа-1А цепи белка тубулина. Лучшая находка blast имеет %Identity=92%,score=828,покрытие ~96%, поэтому у организма Amoeboaphelidium protococcarum
имеется гомолог рассматриваемого белка.
Тубулин- основной компонент микротрубочек, которые являются основным компонентом цитоскелета эукариот,
образуют митотическое веретено, участвуют в транспортировке "частиц" в клетке и т.д.
3)Kinesin-like protein KIF2A
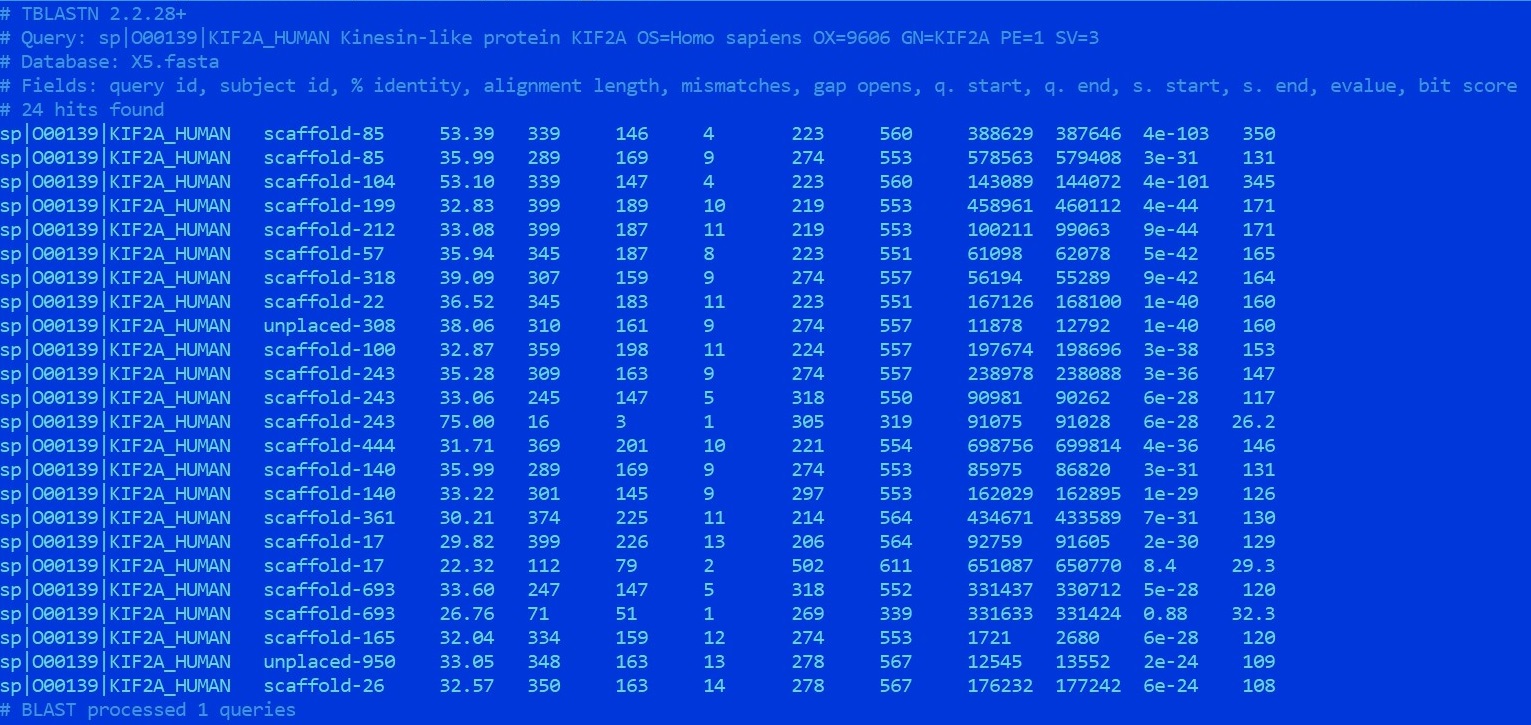
Лучшая находка blast имеет %Identity=53% и покрытие ~50%. Белок - условно гомологичный,
т.к. гомологичны отделные домены(рис. 4).
Кинезины - сем-во моторных белков эукариот, они двигаются по микротрубочкам, используя
энергию гидролиза АТФ, принимают участие в различных клеточных процессах: митозе, мейозе, везикулярном транспорте и т.д.
Типичный кинезин- это димер,каждая половина которого состоит из тяжёлой и лёгкой цепей.
Тяжёлая образует глобулярную головку, которая связывается с микротрубочками и имеет
консервативную аминокислотную последовательность(несколько доменов).

4.Какой-нибудь ген белка в одном из контигов
Был выбран scaffold-700 из сборки Amoeboaphelidium X5(длина- 38453). Поиск проводился с помощью blastx(ограничения: DB-RefSeq, таксон - Fungi). Результаты на рис.5
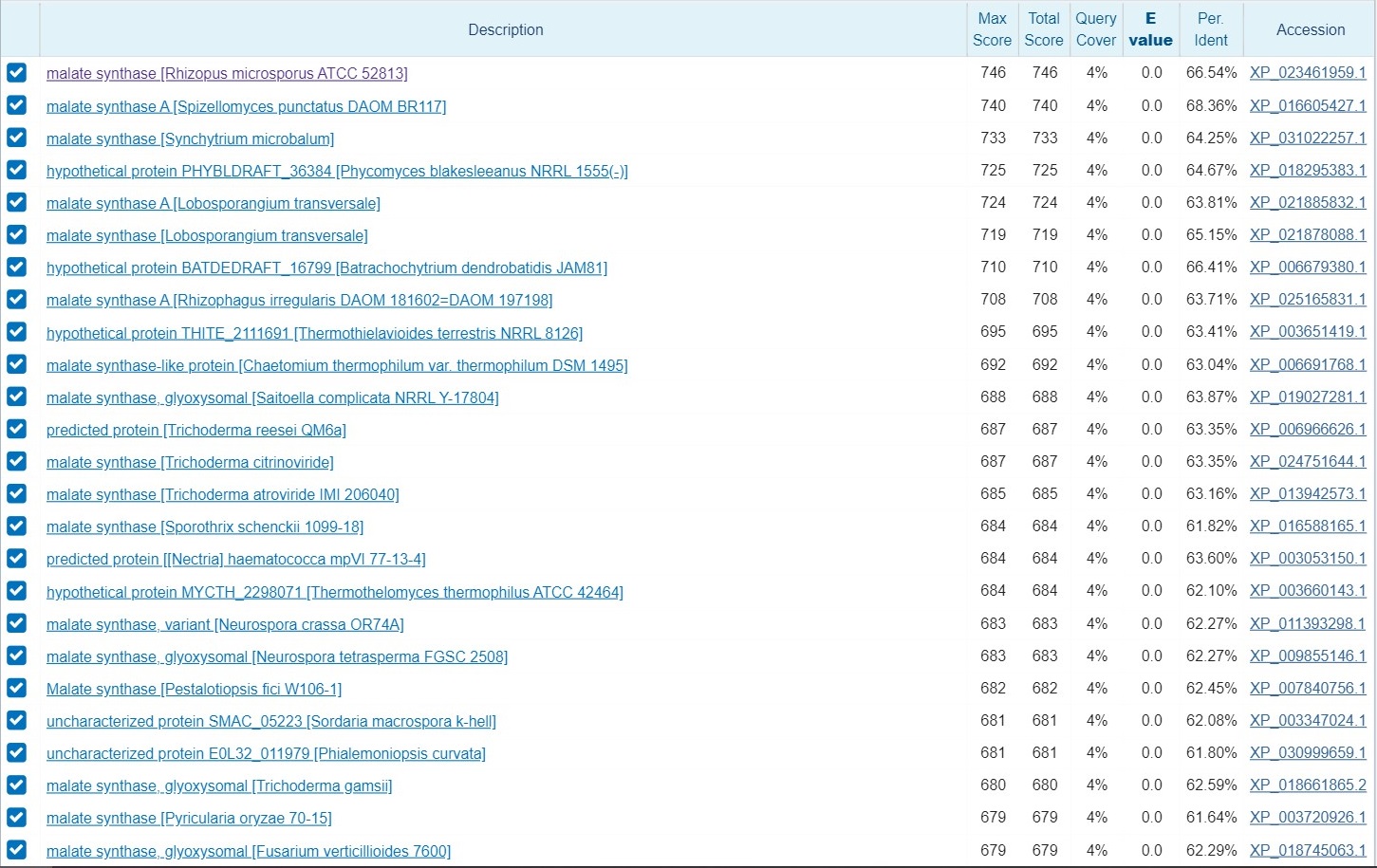
Большинство найденных последовательностей являются малат-синтетазой,а их выравнивания с query имеют e-value=0, %Identity>60, %Positives>70. Это позволяет сделать вывод о наличии гена малат-синтетазы в рассматриваемом скэффолде.(для ещё большей уверенности можно посмотреть на выравнивание с какой-нибудь из находок - рис.6).
