Практикум 6. Валидация
Задание 1.
В данном практикуме изучалась структура 4PV0. Данная структура представляет собой тирозинкиназу селезёнки (Syk) в комплексе с ингибитором
имидазопиразином. SYK – это внутриклеточная цитоплазматическая нерецепторная тирозинкиназа, имеющая в своём составе SH2-домен. Благодаря этому домену Syk
способен присоединяться к фосфорилированным остаткам тирозина, содержащимся в других белках. Данный белок является важным посредником в иммуно-рецепторном
сигнальном пути. Включение иммунорецептора вызывает фосфорилирование пары остатков тирозина в мотивах ITAM (immunoreceptor tyrosine-based activation motif),
после чего к ним присоединяется Syk с помощью своего домена SH2. Syk регулирует работу иммунных клеток, участвуя в некоторых сигнальных каскадах (например,
PI3K, MAPK, Btk, и PLCγ). Он активирует эти пути через фосфорилирование непосредственных мишеней, таких как BLNK/SLP65. В результате активация этих путей в
иммунных клетках приводит к их пролиферации, дифференциации, выбросу цитокинов. [1]
Тирозинкиназа Syk является мишенью для ингибиторов, использующихся при лечении некоторых воспалений, аутоиммунных и онкологических заболеваний.
Структура 4PV0 была получена в этой статье. В данной работе разрабатывались и изучались серия имидазо[1,2-а]пиразинов,
ингибирующих Syk, и эффективность их ингибирования.
А рассматриваемая в этом практикуме структура 4PV0 является одним из таких ингибиторов, связанным с киназным доменом тирозинкиназы Syk.
Качество структуры
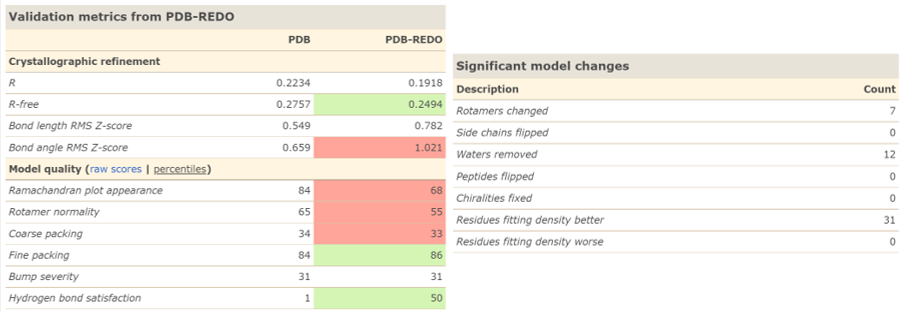
Разрешение структуры хорошее и составляет 2А с полнотой 96.7%. Значения R-фактора и R-free составляют соответственно 0.224 и 0.278 (по данным депозитора). Это удовлетворительные значения (хорошими считаем < 20%, плохими > 40%), также хорошо, что разница между Rfree и R-фактором меньше 10%, т.е. во время оптимизации модели переобучения не произошло.
По картам Рамачандрана из 262 остатков 3%(8 а.о.) лежат в разрешённой области, и есть один outlier. Проанализировано 262 из 275 остатков. У 30% PDB моделей с таким же разрешением этот показатель хуже.
По ротамерам боковых цепей остатков - 3% (7 а.о.) маргинальных. Проанализировано 233 из 238 остатков. У 41% PDB моделей с таким же разрешением этот показатель хуже.
По показателю RSR Z-score 9% (26 а.о.) остатков маргиналы, у них RSR Z-score>2. Проанализировано 268 из 275 остатков. У 7% PDB моделей с таким же разрешением этот показатель хуже.
Clash-score равен 3. Всего в модели обнаружено 14 перекрываний Ван-дер-Ваальсовых радиусов. При этом 4 из них это перекрывания радиусов водородов, и поскольку они были расставлены автоматически, то вряд ли они будут сильно снижать качество модели.
В структуре есть два участка, для которых нет подтверждающей их ЭП- 380-381 и 405-409. По данным Uniprot остатки 380-381 входят в состав сайта, связывающего АТФ.
Задание 2.
В этом задании рассматривались некоторые остатки, являющиеся маргинальными по различным показателям качества согласно отчёту PDB.
1)Клэш между NZ Lys-397 и OE2 Glu-449 (рис.2) Расстояние между центрами атомов составляет 2.17, перекрывание Ван-дер-ваальсовых радиусов - 0.68.
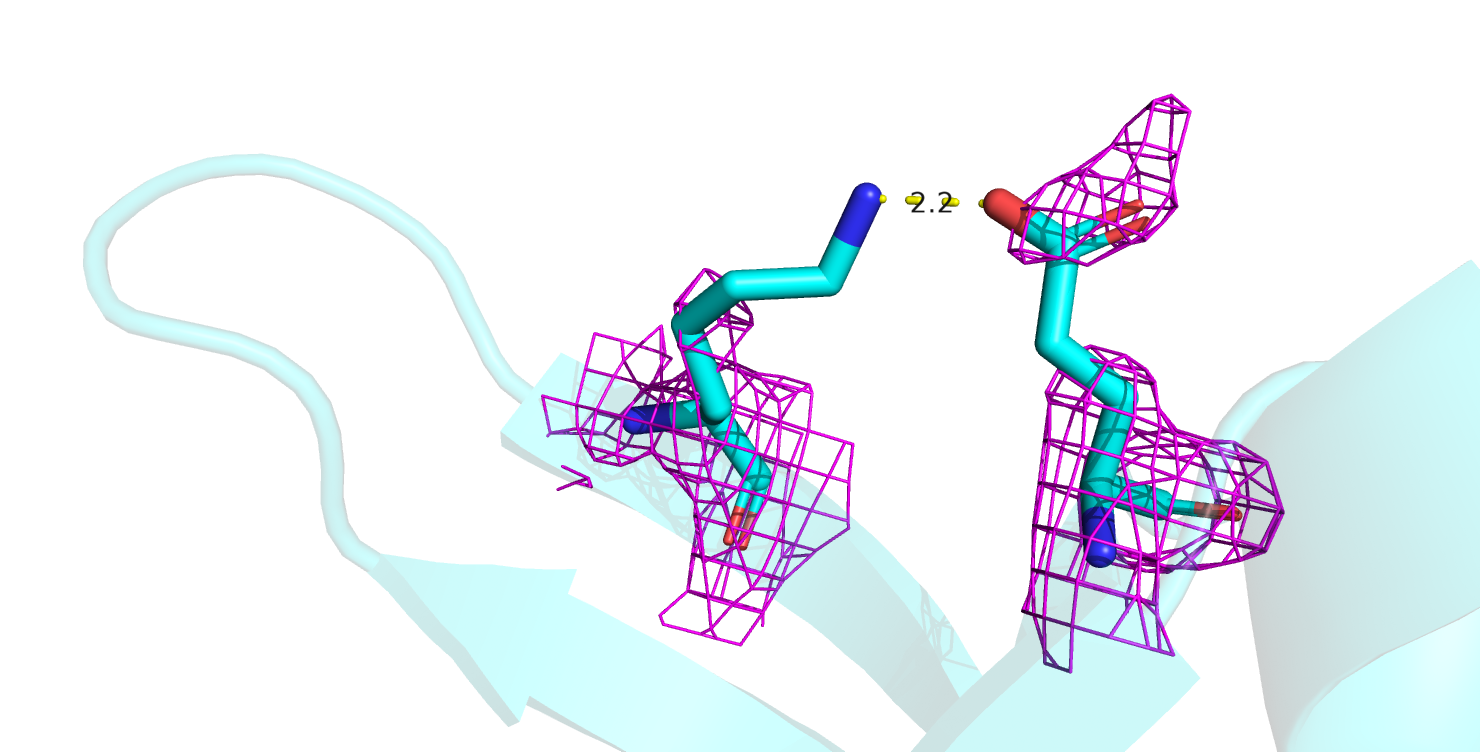
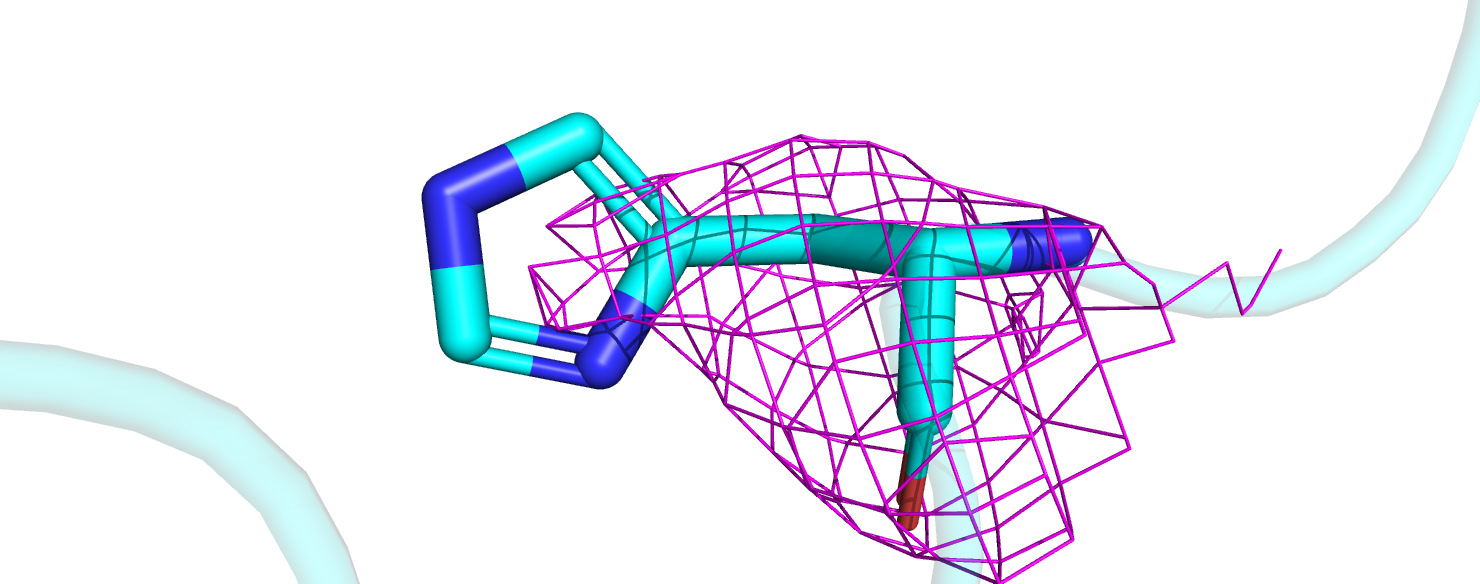
2)Остаток, являющийся выбросом по картам Рамачандрана, - His-531 (рис.3). Данный остаток также маргинал по RSRZ-score (RSRZ-score=4.9).
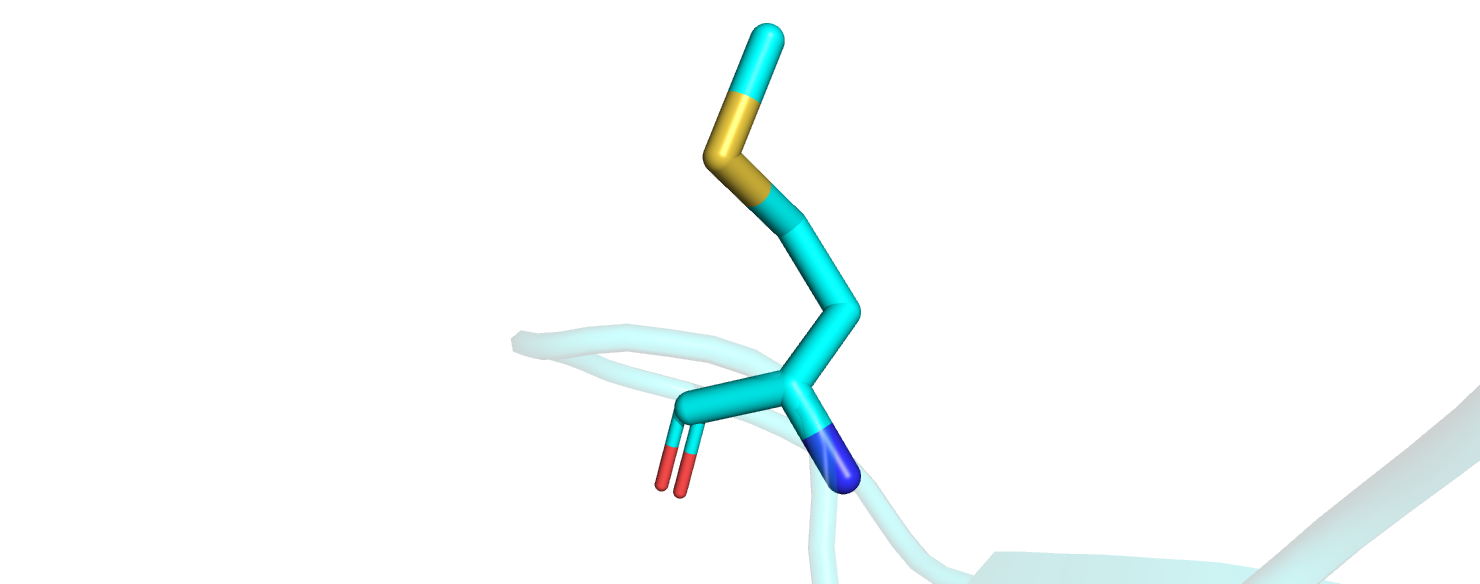
3)Мet-392 имеет неротамерную конформацию боковой цепи. (рис.4)
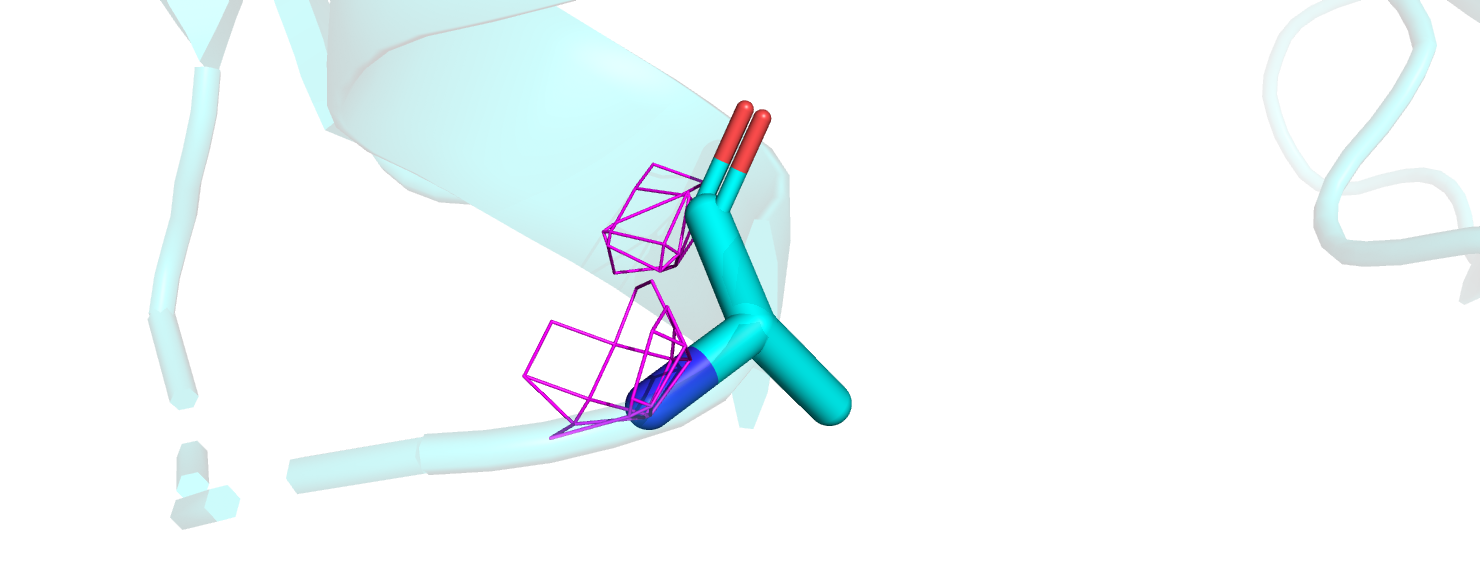
4)Ala 412 плохо вписан в ЭП, RSRZ score= 4.8. (рис.5)
5)Leu 369 имеет неротамерную конформацию боковой цепи. (рис.6)
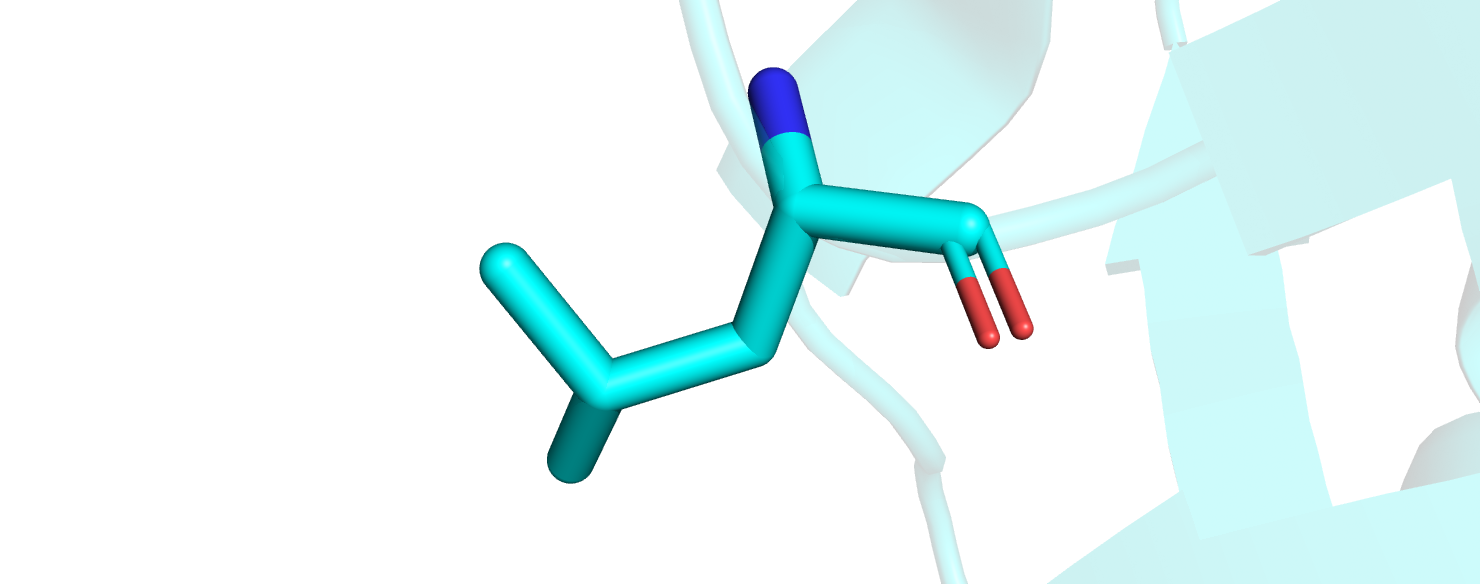
По результатам анализа геометрии в MolProbity было обнаружено 2 маргинальных остатков, отсутствующих в отчёте PDB.
Это Val-363 и Lys-533 – маргиналы по RSRZ-score в отчёте PDB и по неротамерности боковой цепи в MolProbity. После добавления водородов в MolProbity и
проверки структуры на инверсии были найдены 3 остатка Asn-470, Asn-499, His-531, рекомендованные для разворота. В отчёт PDB эти остатки также присутствовали.
Далее был проведён анализ качества геометрии со всеми доступными контактами (Analyze all-atom contacts and geometry) и были обнаружены два клеша, отсутствующие
в отчёт PDB: Asn470 C c водородом HD22 Asn470 (водород с водородом, так что этот клэш не информативный) и Met-601 HE3 c TYR-629 CD2.
В рассматриваемой структуре также присутствуют 5 ионов Cl- (рис.7). Не очень понятно, что эти ионы делают в структуре, потому что судя по рис.7
они просто весят в воздухе. Выдача CheckMyMetal, содержала только два иона Cl – 705 и 706, хотя в структуре их 5 (702, 703, 704, 705, 706).
Проанализированные ионы хлора оказались outlierами по геометрии (free), т.е. вокруг этих ионов нет никаких лигандов.
Таким образом, расположению этих ионов в структуре нельзя доверять.
Задание 3.
Разрешение структуры 4PV0 высокое с хорошей полнотой (2А с 96,7%).
По значениям R-фактора и R-free качество среднее. По картам Рамачандрана есть только один выброс – His-531, который также обладает неротамерной боковой цепью.
Он находится вне какой-либо вторичной структуры, а значит довольно подвижен, и на мой взгляд, не сильно влияет на качество. Судя по отчёту Uniprot в структуре
4PV0 есть три функционально значимых участка, в одном из них (позиции 377-385 – сайт связывания АТФ) есть маргинальный Phe-382 с неротамерной боковой цепью,
а также отсутствует информация о 2 остатках в позициях 380 и 381. Рассматриваемая структура 4PV0 была получена для изучения взаимодействия киназы с ингибитором,
который занимает тот же карман, что и АТФ. И хотя ингибитор удерживается в этом кармане посредством взаимодействий с другими а.о., отсутствие ЭП, соответствующей
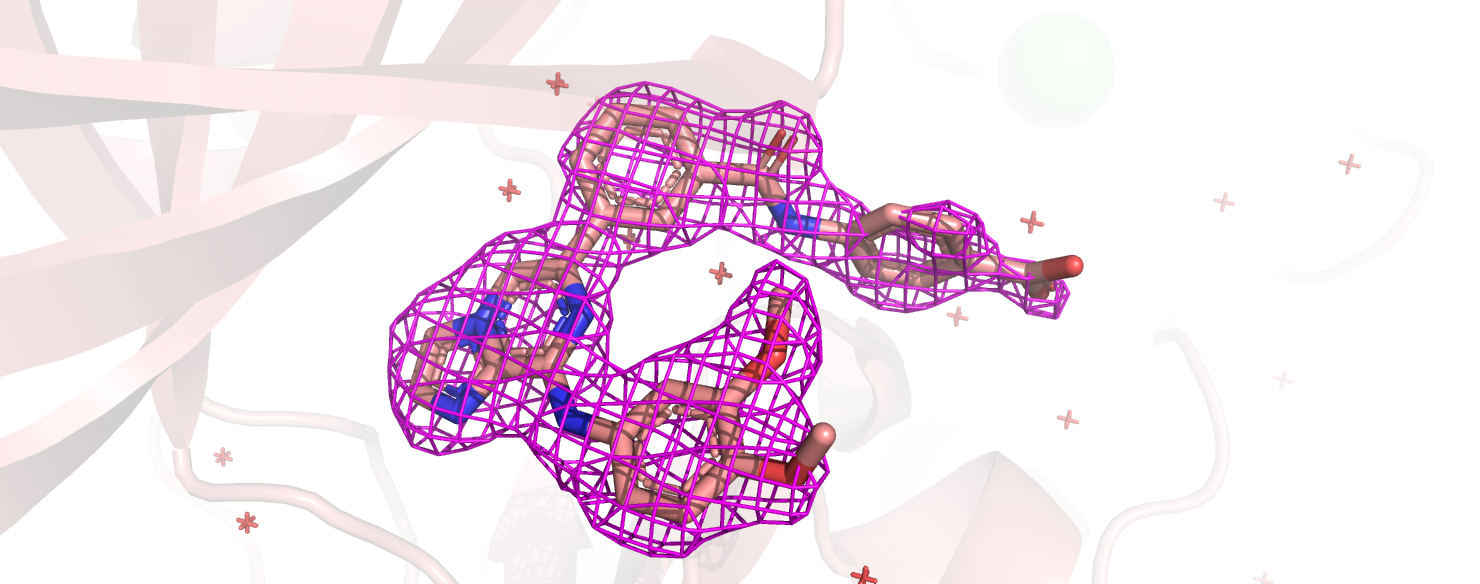
функционально значимым участкам, не внушает доверия. ЭП ингибитора хорошо описывает положение атомов.(рис.8)
В целом ЭП плохо поддерживает маргинальные конформации. Рис.2, 3, 5, 6 подтверждают это: визуализированная поверхность с Z-score >=2 или >=1 неполностью описывает конформацию остатков.
Задание 4.
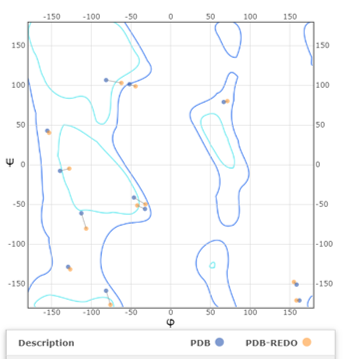
В этом задании изучалась улучшенная структура 4PV0, полученная с сайта PDB Redo (показатели модели приведены на рис. 9). Структура с PDB Redo имела лучшие показатели R-фактора и R-free - 0.1918 и 0.2494 соответственно. По отчёт MolProbity в улучшенной структуре появилось несколько связей с плохими значениями длин (7 шт) и 5 плохих значений углов. По картам Рамачандрана для остатков, находящихся в разрешённой зоне, и аутлаеров (рис.10) видно, что качество многих остатков не улучшилось. Тем не менее у ~трети остатков наблюдалось значимое изменение соответствия ЭП в лучшую сторону, при этом значимого ухудшения у других остатков не наблюдалось. Рассмотрим несколько остатков, для которых значимое улучшение наблюдалось.
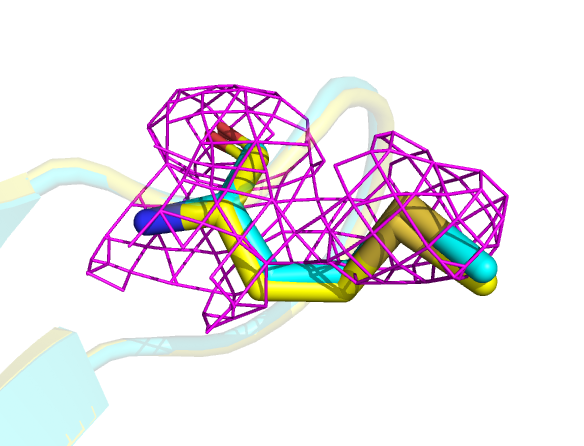
1)Met-392 обладает одним из самых высоких значимых изменений RSCC (delta RSCC=0.132). Этот остаток также рассматривался в задании 2, в котором он был маргиналом по ротамеру. После улучшения остаток перешёл в разрешённую зону (рис. 11)
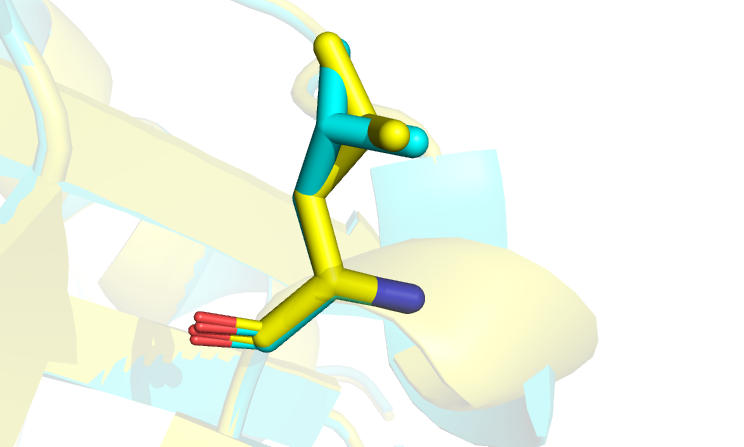
2)У Asp-522 также увеличился RSCC (незначимо на 0.024), но при этом он стал outlierом и по длине связи между CG--OD1 (1.5А) и по углу CA-CB-CG.(рис.12) Вот и пример, когда попытка получше вписать а.о. в ЭП приводит к плохому результату.
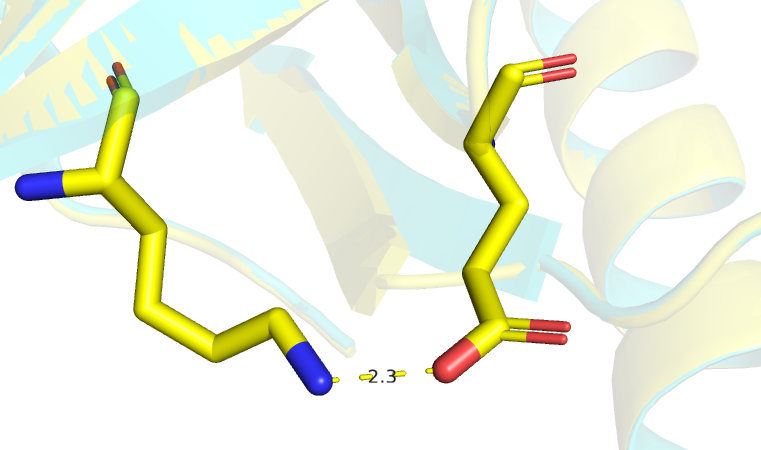
3)Клэш между NZ Lys-397 и OE2 Glu-449 не исчез в модели Redo, хотя расстояние между атомами увеличилось с 2.17 до 2.3 (рис.13)
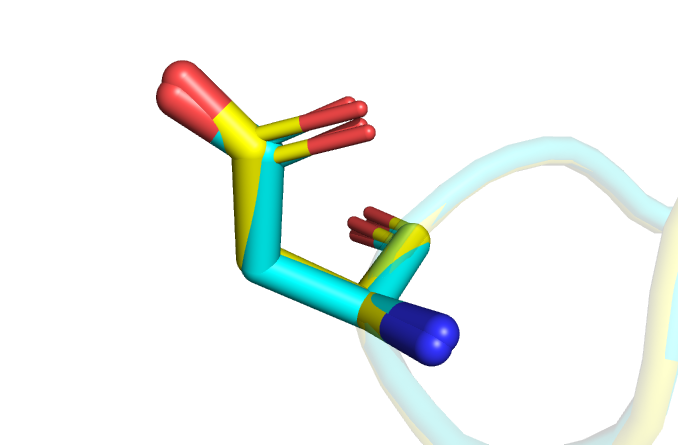
4)Leu-369 также увеличил свой RSCC (на 0.091). Данный остаток рассматривался в задании 2 и был маргинальным за счёт неротамерной конформации бокового радикала. В модели Redo от этой проблемы удалось избавиться.(рис.14)
На мой взгляд, для изучения данного белка лучше брать структуру из PDB, а не из PDB Redo. Хотя модель Redo выигрывает по основным метрикам качества, тем не менее в результате улучшения этих метрик в модели появились ошибки, которых раньше не было. Например, неправдоподобные длины связей и значений углов.


