Отобранные бактерии:
| Название
|
Мнемоника |
| Pasteurella multocida |
PASMU |
| Haemophilus influenzae |
HAEIN |
| Proteus mirabilis |
PROMPH |
| Escherichia coli |
ECOLI |
| Neisseria meningitidis |
NEIMA |
| Burkholderia cenocepacia |
BURCA |
| Rhodobacter sphaeroides |
RHOS4 |
| Bradyrhizobium japonicum |
BRAJA |
Скобочная формула дерева:
((((PROMPH, ECOLI), (PASMU, HAEIN), (NEIMA, BURCA)), (RHOS4,
BRAJA));
Изображение дерева:
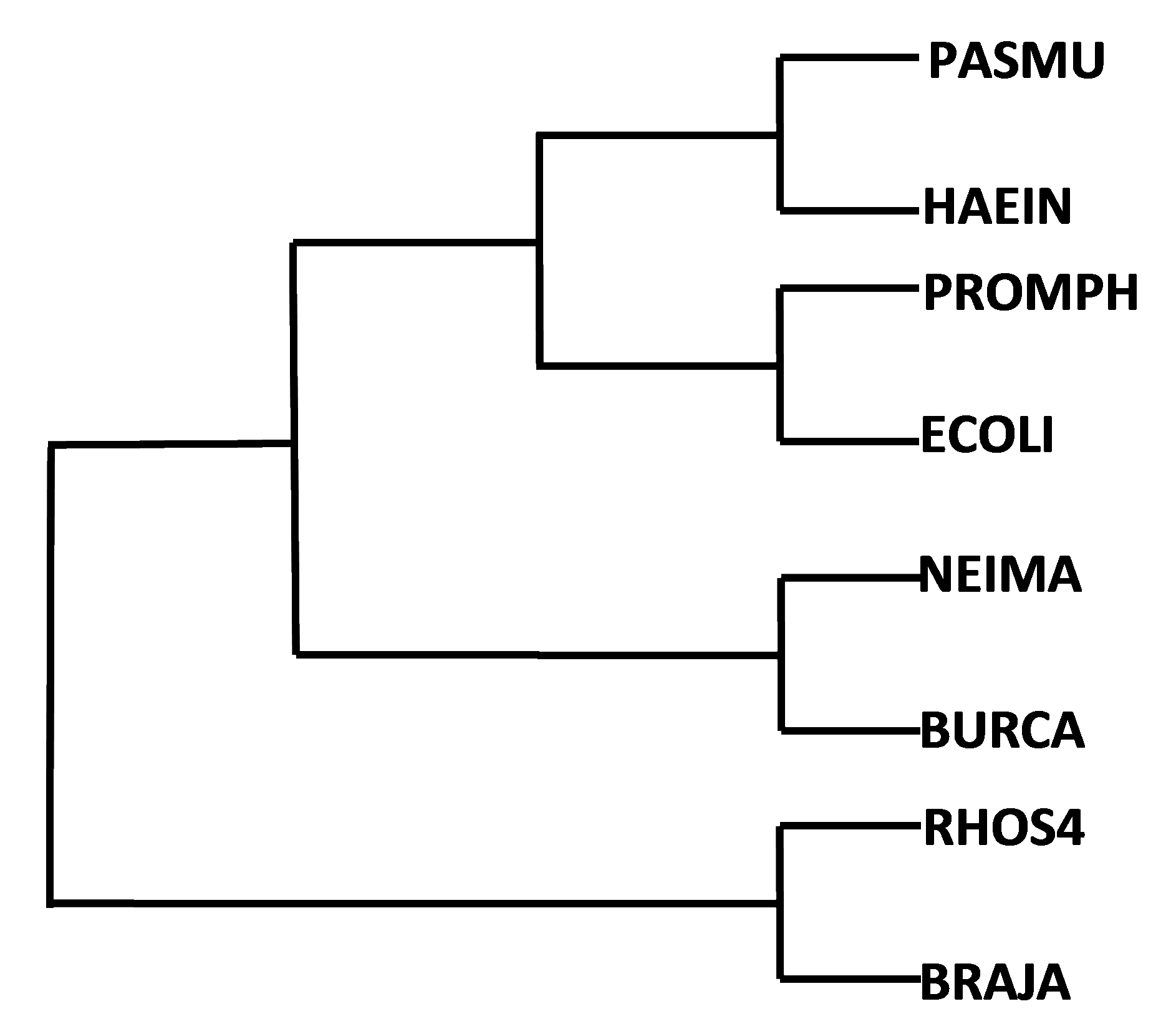
Нетривиальные ветви дерева:
{PASMU, HAEIN} против {PROMPH, ECOLI, NEIMA, BURCA, RHOS4,
BRAJA}
{PROMPH, ECOLI} против {PASMU, HAEIN, NEIMA, BURCA, RHOS4,
BRAJA}
{PASMU, HAEIN, PROMPH, ECOLI} против {NEIMA, BURCA, RHOS4,
BRAJA}
{PASMU, HAEIN, PROMPH, ECOLI, NEIMA, BURCA} против {RHOS4,
BRAJA}
{NEIMA, BURCA} против {PASMU, HAEIN, PROMPH, ECOLI, RHOS4,
BRAJA}
Таксономия отобранных бактерий:
|
Названия
|
Мнемоника
|
Таксономия
|
|
Pasteurella multocida
|
PASMU
|
Bacteria; Proteobacteria; Gammaproteobacteria;
Pasteurellales; Pasteurellaceae; Pasteurella
|
|
Haemophilus influenzae
|
HAEIN
|
Bacteria; Proteobacteria; Gammaproteobacteria;
Pasteurellales; Pasteurellaceae; Haemophilus
|
|
Proteus mirabilis
|
PROMPH
|
Bacteria; Proteobacteria; Gammaproteobacteria;
Enterobacteriales; Enterobacteriaceae; Proteus
|
|
Escherichia coli
|
ECOLI
|
Bacteria; Proteobacteria; Gammaproteobacteria;
Enterobacteriales; Enterobacteriaceae; Escherichia
|
|
Neisseria meningitidis
|
NEIMA
|
Bacteria; Proteobacteria; Betaproteobacteria;
Neisseriales; Neisseriaceae; Neisseria
|
|
Burkholderia cenocepacia
|
BURCA
|
Bacteria; Proteobacteria; Betaproteobacteria;
Burkholderiales; Burkholderiaceae; Burkholderia; Burkholderia
cepacia complex
|
|
Rhodobacter sphaeroides
|
RHOS4
|
Bacteria; Proteobacteria; Alphaproteobacteria;
Rhodobacterales; Rhodobacteraceae; Rhodobacter
|
|
Bradyrhizobium japonicum
|
BRAJA
|
Bacteria; Proteobacteria; Alphaproteobacteria;
Rhizobiales; Bradyrhizobiaceae; Bradyrhizobium
|
Ветви,
выделяющие таксоны:
|
Ветвь
|
Таксономия
|
| {PASMU, HAEIN} против {PROMPH, ECOLI,
NEIMA, BURCA, RHOS4, BRAJA} |
Bacteria; Proteobacteria; Gammaproteobacteria;
Pasteurellales; Pasteurellaceae; |
| {PROMPH, ECOLI} против {PASMU, HAEIN,
NEIMA, BURCA, RHOS4, BRAJA} |
Bacteria; Proteobacteria; Gammaproteobacteria;
Enterobacteriales; Enterobacteriaceae; |
| {PASMU, HAEIN, PROMPH, ECOLI} против {NEIMA,
BURCA, RHOS4, BRAJA} |
Bacteria; Proteobacteria; Gammaproteobacteria; |
| {NEIMA, BURCA} против {PASMU, HAEIN, PROMPH,
ECOLI, RHOS4, BRAJA} |
Bacteria; Proteobacteria; Betaproteobacteria; |
| {RHOS4, BRAJA} против {PASMU, HAEIN,
PROMPH, ECOLI, NEIMA, BURCA} |
Bacteria; Proteobacteria; Alphaproteobacteria; |
По следующему выбранному белку:
|
Функция
|
Мнемоника
|
|
Рибосомный белок L2
|
RL2
|
были получены из Swiss-Prot последовательности белков с
данной функцией из отобранных мной бактерий и были созданы
выравнивания с помощью программы muscl
со следующими входным
и выходным
файлами.
Реконструкцию дерева
программой fprotpars:
После ввода в программу выравненных последовательностей
программой muscl на
выходе получаем скобочную формулу:
((((RS7_NEIM0,RS7_BURCA),(RS7_HAEI8,(RS7_PASMU,(RS7_PROMH,RS7_ECO24)))),RS7_RHOS1),RS7_BRAJA);
и изображение самого дерева, которое
было единственным:
+--RS7_NEIM0
+-----------7
! +--RS7_BURCA
+--6
! ! +--------RS7_HAEI8
! +-----4
! ! +-----RS7_PASMU
+--2 +--5
! ! ! +--RS7_PROMH
! ! +--3
1 ! +--RS7_ECO24
! !
! +-----------------RS7_RHOS1
!
+--------------------RS7_BRAJA
Новые ветви:
{PASMU, PROMPH, ECOLI} против {HAEIN, NEIMA, BURCA, RHOS4,
BRAJA}
Матрица расстояний, построенная
с помощью программы fprotdist
|
|
BRAJA
|
RHOS4
|
ECOLI
|
PROMPH
|
HAEIN
|
PASMU
|
BURCA
|
NEIMA
|
|
BRAJA
|
0.000000
|
0.327701
|
0.534181
|
0.562545
|
0.442330
|
0.492769
|
0.485777
|
0.495099
|
|
RHOS4
|
0.327701
|
0.000000
|
0.473184
|
0.458584
|
0.453083
|
0.464802
|
0.521511
|
0.510698
|
|
ECOLI
|
0.534181
|
0.473184
|
0.000000
|
0.037619
|
0.107705
|
0.113360
|
0.434550
|
0.429982
|
|
PROMPH
|
0.562545
|
0.458584
|
0.037619
|
0.000000
|
0.121596
|
0.115191
|
0.445370
|
0.439064
|
|
HAEIN
|
0.442330
|
0.453083
|
0.107705
|
0.121596
|
0.000000
|
0.077387
|
0.432272
|
0.399643
|
|
PASMU
|
0.492769
|
0.464802
|
0.113360
|
0.115191
|
0.077387
|
0.000000
|
0.471159
|
0.397012
|
|
BURCA
|
0.485777
|
0.521511
|
0.434550
|
0.445370
|
0.432272
|
0.471159
|
0.000000
|
0.240543
|
|
NEIMA
|
0.495099
|
0.510698
|
0.429982
|
0.439064
|
0.399643
|
0.397012
|
0.240543
|
0.000000
|
Оценим, насколько расстояния
отклоняются от ультраметричности:
Рассмотрим 3 объекта: BURCA,RHOS4, BRAJA
d(BURCA,RHOS4) = 0,521511
d(BURCA, BRAJA) = 0,485777
d(RHOS4, BRAJA) = 0,327701 - в одной кладе
d(BURCA,RHOS4) > d(RHOS4,BRAJA), но d(BURCA, BRAJA) не
равно d(BURCA, RHOS4)
Разница отклонения между d(BURCA, BRAJA) и d(BURCA, RHOS4)
= 0,035734
Следовательно они не удовлетворяют условию ультраметричности.
Оценим, насколько расстояния
отклоняются от аддитивности:
Рассмотрим 4 объекта: PASMU, HAEIN, BURCA, NEIMA
d(PASMU, HAEIN) + d(BURCA, NEIMA) = 0,317917
d(PASMU, BURCA) + d(HAEIN, NEIMA) = 0,903431
d(PASMU, NEIMA) + d(HAEIN, BURCA) = 0,829284
Из 3х сумм две наиболее близкие по значению d(PASMU, BURCA)
+ d(HAEIN, NEIMA) и d(PASMU, NEIMA) + d(HAEIN, BURCA) действительно
больше третьей d(PASMU, HAEIN) + d(BURCA, NEIMA), но они
отклоняются от равенства на 0,074147.
Реконструкция дерева программой fneighbor.
Для того, чтобы поменять алгоритм с Neighbor-Joining, который
стоит по умолчанию, на UPGMA, необходимо ввести команду:
fneighbor -treetype u
Результаты с алгоритмом Neighbor-Joining: all_seq_al_NJ.treefile,
all_seq_al_NJ.fneighbor
+--------RS7_RHOS1
!
! +-------RS7_BURCA
! +-----2
! ! +------RS7_NEIM0
1------3
! ! +-RS7_HAEI8
! ! !
! +--------5 +RS7_ECO24
! ! +--4
! +-6 +-RS7_PROMH
! !
! +--RS7_PASMU
!
+---------RS7_BRAJA
Результаты с алгоритмом UPGMA: all_seq_al_UPGMA.treefile,
all_seq_al_UPGMA.fneighbor
+---------RS7_BRAJA
+----5
! +---------RS7_RHOS1
!
! +RS7_ECO24
--7 +--1
! ! +RS7_PROMH
! +--------3
! ! ! +--RS7_HAEI8
! ! +-2
+-6 +--RS7_PASMU
!
! +------RS7_BURCA
+-----4
+------RS7_NEIM0
Топология дерева, построенного с использованием алгоритма
UPGMA, полностью совпадает с правильным деревом. Топология
дерева, построенного fprotpars, сходится с топологией построенного
программой fneighbor с использованием алгоритма Neighbor-Joining
за исключением того, что в последнем BRAJA и RHOS1 находятся
на одной ветви, а в дереве fprotpars на разных ветвях. Единственное
различие между алгоритмом Neighbor-Joining и правильным
деревом:
{PASMU, PROMPH, ECOLI} против {HAEIN, NEIMA, BURCA, RHOS4,
BRAJA}