Первый этап: описание функциональных
особенностей заданной группы.
Заданный мне белок-прототип LACI_Ecoli является репрессором
транскрипции lac-оперона.
Лактозный ингибитор Lacl является ДНК-связывающимся транскрипционным
фактором репрессирует транскрипцию оперона участвующего
в транспорте и катаболизме лактозы. При отсутствии алолактозы,
LacI репрессирует lac оперон. В данной системе репрессии
LacI присоединяеться к двум операторам, а образование репрессионной
петли является критичным. Этот репрессор присоединяет в
тандеме к обратным повторяющимся последовательностям, длина
которых составляет в 21 нуклеотид, являющиеся консервативными
мотивами.
Lacl кодирует белок репрессор который может обратимо соединяется
с операторным районом и препятствует транскрипции.
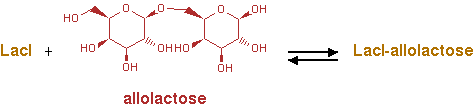
Индукция происходит когда физиологический индуктор, аллолактоза,
связывается с lac репрессором, препятствуя ему связаться
с оператором. Нефизиологические аналоги, такие как тиогалактозиды
могут тоже работать как индукторы.
Белок Lacl принадлежит GalR/LacI семейству и как член данной
семьи транскрипционных регуляторов, Lacl состоит из 3х доменов:
спираль - петля - спиральный мотив расположенный в N-конце,
центральный домен, который присоединяется к лигандом сахаров,
и C-терминальный, который включает тетрамеризационный домен.
Реакционная формула с аллолактозой:
Второй этап: создание множественного
выравнивания доменов с разметкой по группам специфичности.
1. Создать хорошее множественное выравнивание
доменов заданной группы белков
Доменная структура LACI_Ecoli белка-прототипа
заданной группы (страница InterPro):
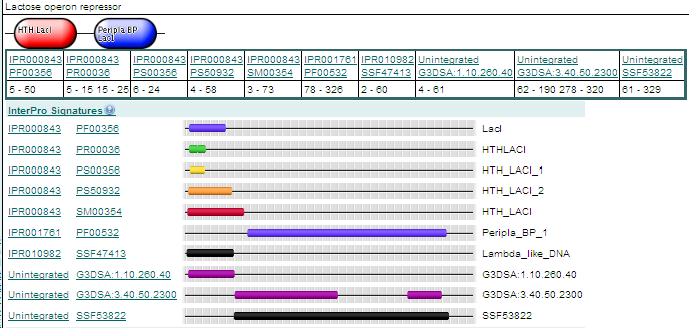
Для дальнейшей работы мы берем домен из БД
SMART: SM00354. Данный домен является самым длинным из правильно
расположенных доменов. БД SMART хоть является довольно избыточной,
но в ней нет идентичных белков.
Все последовательности ДНК-связывающих доменов
семейства LACI были получены из БД
SMART через поиск по имени домена HTH_LACI. По которому
было найдено представительское выравнивание доменов в формате
FASTA: SMART.fasta и последовательности
всех бактериальных доменов данного семейства в формате FASTA,
которые были сохранены в текстовом файле: allseq.txt
Откуда были полученны последовательности белков
указанных в списке идентификаторов
последовательностей, по мнению эксперта, обладающих
данной специфичностью в файле dna_laci.fasta
Далее было произведено выравнивание с помощью программой
ClustalW2. На выходе данной программы было получено выравнивание.
После чего из него были удалены все последовательности представительского
выравнивания. В итоге получился файл.
2. Создать единое множественное выравнивание заданных
доменов всех групп специфичности
В БД Pfam была получена доменная организация белка прототипа
LACI_Ecoli
Домен красного цвета является эффектор связывающим. Далее
были получены выравнивания всех доменов этого типа: PF00532_full,
откуда с помощью скрипта
были получены необходимые выравнивания, которые указаны
в файле laci, и сохранены
в файле PF00532_laci.fasta.
Файл множественного выравнивания: all_align.msf
Графический файл: all_align.jpeg
Позиция 582 является консервативной для всех групп.
3. Создайте лого-изображения полного выравнивания заданных
доменов и выравнивания доменов заданной группы специфичности
Используя ресурс WebLogo были созданы лого-изображения
Третий этап: Построение
профиля
4. Добавление веса в выравнивание
С помощью программы pwf из пакета PFTOOLs
напишем команду, которая добавит вес в выравнивание:
pfw -m PF00532_laci.fasta > out.weighted.fasta
5. Построение профиля
Построение профиля делаем с помощью команды:
pfmake -m out.weighted.fasta /usr/share/pftools23/blosum45.cmp
> myprofile.prf
Затем нормируем профиль относительно случайной базы:
autoscale -m myprofile.prf > myprofile.scaled.prf
6. Поиск по профилю в выборке последовательностей
Выполняем с помощью команды:
pfsearch -f myprofile.scaled.prf Edwardsiella.fasta>
ed.search
Получаем файл, который оказался
пустым, следовательно в данном организме белка, отвечающего
данному профилю нет. Возможно в этом организме нет белка,
выполняющего данную функцию.