1. Для димера пуриновых репрессоров 1JFT были созданы изображения:
a) поверхности контакта мономера белка с симметричным мономером на фоне остовной (ribbon) модели мономера:
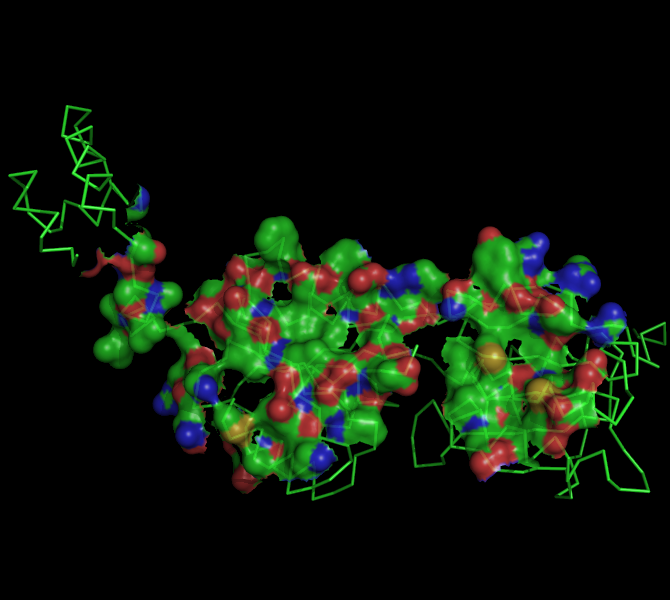
б) поверхности контакта димера белков с двойной спиралью ДНК на фоне остовной модели части белка, вовлечённой в контакт:
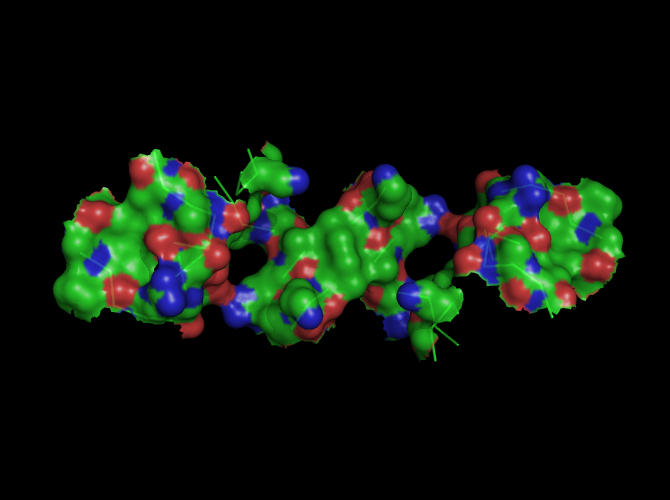
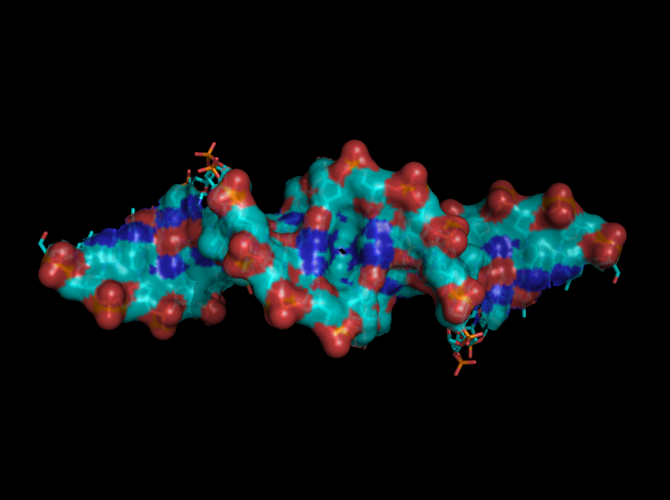
в) поверхности контакта ДНК с димером белков на фоне проволочной (sticks) модели двойной спирали:
2. Определения площади контактов мономеров белка с помощью сервиса PROTORP
По результатам выдачи сервиса PROTORP:
- площадь контакта мономеров белка (Interface Accessible Surface Area) = 2548.21Å2
- часть, приходящаяся на гидрофобные взаимодействия = 43.41%
3. Сервис CluD, определение гидрофобных кластеров на интерфейсе мономеров того же белка
На выходе программы CluD всеравно встречались класеры объемом больше 10 атомов. Поэтому этот файл был отредактирован вручную. В итоге был получен файл. После чего был написан скрипт для построения изображения, что и в упр. 1а, на котором поверхность, относящаяся к атомам, входящим в найденные гидрофобные кластеры, выделена цветом
4. Определение доменной структуры с помощью сервиса pDomains
а) Было произведено опредеделение доменной структуры записи 2AE0. Данный сервис выдал следующие результаты:
| Method |
Domain Id |
Fragment |
| SCOP |
2AE0X1 |
3-337 |
|
| pdp |
2AE0X1 |
3-103 , 244-337 |
|
2AE0X2 |
104-243 |
|
| DHcL |
2AE0X1 |
3-337 |
|
| dp |
2AE0X1 |
3-104 , 243-337 |
|
2AE0X2 |
105-242 |
|
| DDomain |
2AE0X1 |
3-337 |
|
| NCBI |
2AE0X1 |
1-104 , 248-345 |
|
2AE0X2 |
105-247 |
|
| PUU |
2AE0X1 |
1-102 , 241-335 |
|
2AE0X2 |
103-187 , 217-240 |
Метод SCOP, DDomain и DHcL определили всю структуру как 1 домен. Методы pdp, dp и NCBI определили 2 домена в данной структуре, причем исходя из аминокислотной последовательности один домен поделен на 2 части другим. Однако если посмотреть на пространственную структуру с выделением доменов разными цветами, то становится понятно, что данное разделение может иметь смысл.
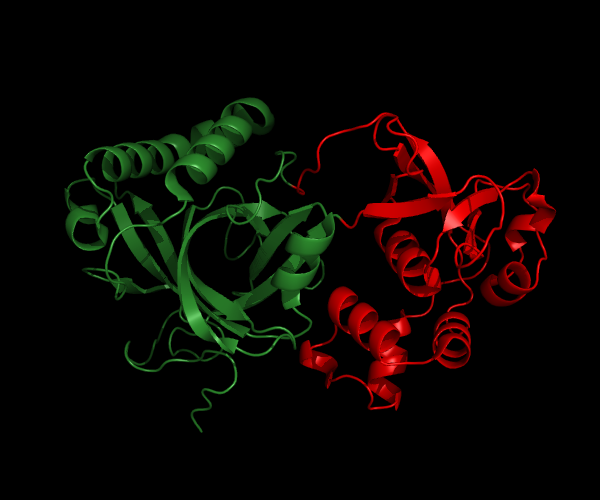
зеленым цветом вылелены остатки 3-103 и 224-337
Метод PUU определил домены примерно так же как и методы pdp, dp и NCBI, за исключением того, что он исключил из 2ого домена последовательность из 30 аминокислот и тем самым разделил его на 2 части. Вот как это выглядет в пространственной структуре
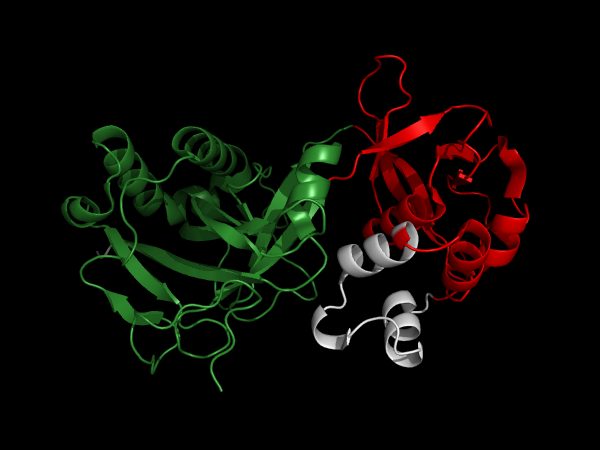
белым цветом выделена часть, неопределенная как домен
Из изображения видно, что неопределенная последовательность является 3 небольшими альфа-спиралями, и небольшая часть верхней альфа-спирали определена как домен. По моему мнению наиболее адекватный результат это определение доменов методами pdp, dp и NCBI. Небольшая разница в координатах доменов между данными методами не касается структурированных участков.
б) Было произведено опредеделение доменной структуры записи 1JFT
| Method |
Domain Id |
Fragment |
| CATH |
1JFTA1 |
2-58 |
|
1JFTA2 |
59-160 , 291-322 |
|
1JFTA3 |
161-290 , 323-339 |
|
| SCOP |
1JFTA1 |
1-57 |
|
1JFTA2 |
58-340 |
|
| pdp |
1JFTA1 |
1-51 |
|
1JFTA2 |
52-141 |
|
1JFTA3 |
142-340 |
|
| DHcL |
1JFTA1 |
1-116 |
|
1JFTA2 |
117-340 |
|
| dp |
1JFTA1 |
1-57 |
|
1JFTA2 |
58-340 |
|
| DDomain |
1JFTA1 |
1-46 |
|
1JFTA2 |
47-340 |
|
| NCBI |
1JFTA1 |
1-45 |
|
1JFTA2 |
58-160 , 293-340 |
|
1JFTA3 |
161-292 |
|
| PUU |
1JFTA1 |
1-46 |
|
1JFTA2 |
58-157 , 293-318 |
|
1JFTA3 |
158-292 , 319-340 |
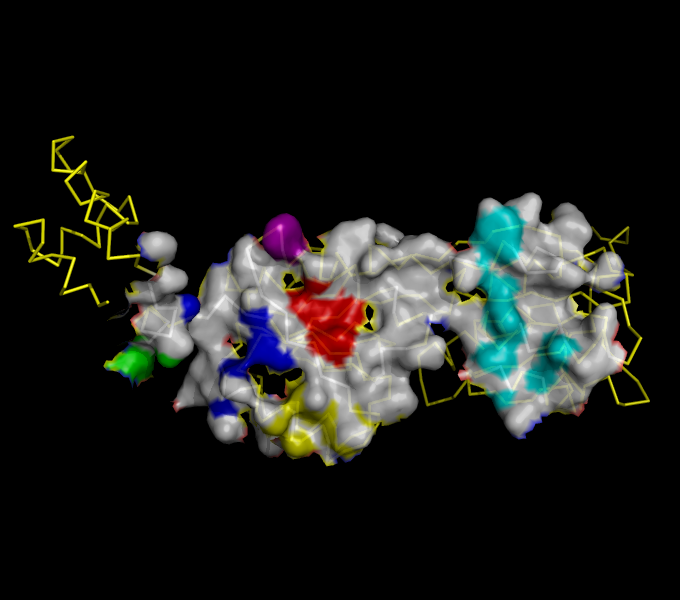
Наиболее правдоподобное определение доменов, на мой взгляд, у методов CATH и PUU:
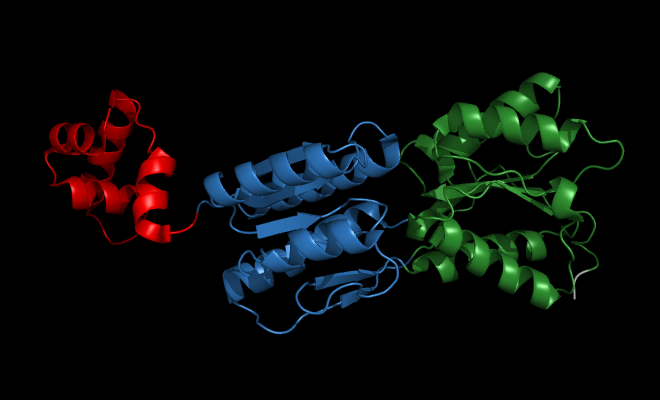
красный -
1JFTA1, голубой - 1JFTA2, зеленый - 1JFTA3, белый - неопределенная как домен;
раскраска по координатам метода CATH