1. Совмещение в PyMOL цепи A из записи 1HY0 и цепи A из записи 1I0A по доменам.
Согласно сервису CATH были определены домены цепей A из обеих записей:
1й домен: 27 - 123, 195 - 234
2й домен:
124 - 194, 235 - 364, 437 - 463
3й домен: 365 - 436
Совмещение цепей А по первой паре доменов RMSD = 0.386:

область выравнивания выделена другим оттенком того же цвета, что и вся цепь
Совмещение цепей А по второй паре доменов RMSD = 0.412:

область выравнивания выделена другим оттенком того же цвета, что и вся цепь
Совмещение цепей А по третьей паре доменов RMS = 0.477:

Пары доменов довольно хорошо совмещаются друг с другом, однако остальная структура имеет достаточное расхождение. Наилучшее совмещение всей структуры получается при совмещении по второй паре доменов. Совмещение по каждой паре имеет хороший RMSD.
2. Построение структурного выравнивания цепей А записей 1HY0 и 1W0T
С помощью программы PDBeFOLD было получено структурное выравнивание цепей А данных записей. Полученный файл был подготовлен (файл) для подачи на вход программе Geometrical core для поиска геометрического ядра с порогом 2 Å. На выходе которой получаем остатки входящие в геометрическое ядро:
| Pos. |
1AKH_A |
1W0T_A |
| 11 |
ALA83 |
LYS389 |
| 12 |
PHE84 |
ASN390 |
| 13 |
LEU85 |
LEU391 |
| 15 |
GLU87 |
SER393 |
| 28 |
LYS100 |
TRP403 |
| 29 |
GLU101 |
SER404 |
| 30 |
GLU102 |
LYS405 |
| 31 |
VAL103 |
ILE406 |
| 41 |
THR110 |
THR416 |
| 42 |
PRO111 |
SER417 |
| 43 |
LEU112 |
VAL418 |
| 44 |
GLN113 |
MET419 |
| 45 |
VAL114 |
LEU420 |
| 46 |
ARG115 |
LYS421 |
| 47 |
VAL116 |
ASP422 |
| 48 |
TRP117 |
ARG423 |
| 49 |
PHE118 |
TRP424 |
| 50 |
ILE119 |
ARG425 |
| 51 |
ASN120 |
THR426 |
| 52 |
LYS121 |
MET427 |
| 53 |
ARG122 |
LYS428 |
| 54 |
MET123 |
LYS429 |
| 55 |
ARG124 |
LEU430 |
Совмещение pair_fit в PyMOL по атомам, относящимся к геометрическому ядру, RMS = 0.865:
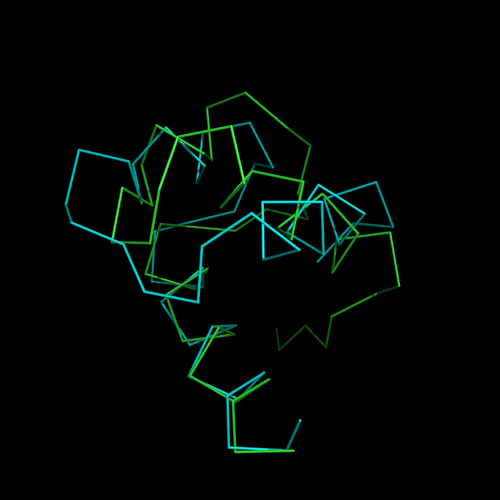
зеленым цветом выделена цепь А 1W0T, голубым - 1AKH
Третья пара спиралей выровнялась лучше всего. Третья спираль цепи А в данных структурах консервативно связывается с ДНК и поэтому является наиболее консервативной частью в этой цепи.
Результатом команды align на полных цепях RMS = 3.088:
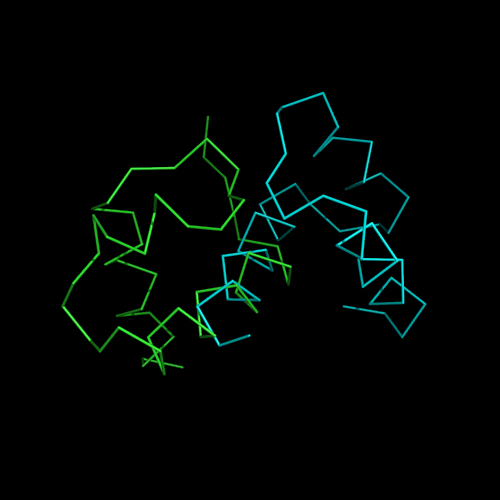
зеленым цветом выделена цепь А 1W0T, голубым - 1AKH
В связи с тем, что последовательность белков достаточно разные выравнивание с помощью align получилось неправильным, тк она сначало делает выравнивание последовательностей,а потом структур.