1. В указанном 2AE0.pdb файле была изучена вторичная структура белка с помощью программы DSSP. Выходной файл 2AE0.dssp был импортиров в excel 2AE0.xls
Результат программы DSSP был сравнен с результатами приведёнными авторами записи
| |
PDB |
DSSP |
| кол-во альфа-спиралей |
12 |
8 |
| кол-во бета-тяжей |
14 |
14 |
| цепи |
одна - Х |
одна - Х |
Координаты бета-тяжей
| № и ID тяжа по pdb |
Координаты PDB |
Координаты DSSP |
| 1 A |
21-24 |
21-24 |
| 2 A |
82-87 |
82-87 |
| 3 A |
326-335 |
326-335 |
| 4 A |
270-278 |
270-278 |
| 5 A |
284-297 |
284-297 |
| 6 A |
257-259 |
257-259 |
| 7 A |
307-313 |
307-313 |
| 8 A |
96-103 |
96-103 |
| 1 B |
118-121 |
118-121 |
| 2 B |
146-150 |
146-150 |
| 3 B |
105-109 |
105-109 |
| 4 B |
165-168 |
165-168 |
| 5 B |
176-183 |
176-183 |
| 6 B |
234-239 |
234-239 |
Как видно из таблицы количество и координаты бета-тяжей выданных программой DSSP и в записях PDB файла полностью совпадают. Из данных результатов можно заключить, что программа DSSP отлично справляется с определением бета-тяжей.
Координаты альфа-спиралей
| № спирали |
Координаты PDB |
Координаты DSSP |
| 1 |
35-50 |
36-49 |
| 2 |
50-70 |
51-69 |
| 3 |
73-79 |
- |
| 4 |
127-130 |
- |
| 5 |
132-138 |
133-137 |
| 6 |
141-144 |
- |
| 7 |
152-163 |
153-162 |
| 8 |
190-198 |
191-197 |
| 9 |
202-205 |
- |
| 10 |
207-217 |
208-216 |
| 11 |
219-228 |
220-227 |
| 12 |
314-322 |
315-321 |
Координаты альфа-спиралей определенных программой DSSP совпадают с записями в PDB за исключением номера первой и последней аминокислоты в каждой спирали (что объясняется алгоритмом программы, который не включает крайние аминокислоты в элементы альфа-спирали). Однако программа DSSP не определила 4 альфа-спирали, которые есть в записе PDB файла. Что, возможно, объясняется их небольшой длинной.
2. Анализ торсионных углов φ
| тип остатка |
номер и цепь |
| глицин |
314 X |
| глицин |
9 X |
| глицин |
312 X |
| глицин |
71 X |
| глицин |
182 X |
| глицин |
3 X |
| глицин |
114 X |
| аргинин |
255 X |
| глицин |
170 X |
| аспарагин |
92 X |
| глицин |
304 X |
| глицин |
72 X |
| глицин |
81 X |
| аланин |
249 X |
| глицин |
15 X |
| глицин |
89 X |
| глицин |
199 X |
| глицин |
282 X |
| глицин |
322 X |
| глицин |
172 X |
| глицин |
94 X |
| глицин |
139 X |
| глицин |
163 X |
| глицин |
268 X |
| глицин |
31 X |
| глицин |
286 X |
Для создания данного изображения в PyMOL был написан скрипт

голубым цветом покрашены альфа-спирали,
желтым бета листы, красным остатки с положительным φ, зеленым все остальное
3. Примеры водородных связей в 2AE0.pdb
Программой HBPlus были определены водородные связи в данной записи. Выдача программы 2AE0.hb2
а) участвующие в стабилизации вторичной структуры
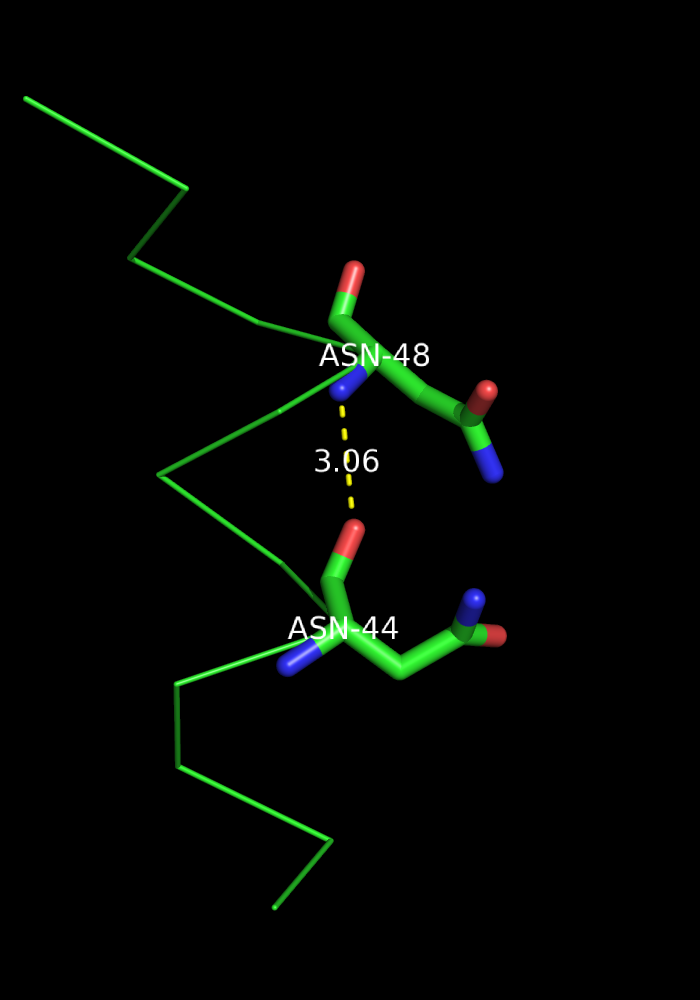
X0048-ASN N X0044-ASN O 3.06 MM 4 6.24 155.3 2.13 146.2 150.9 87
б) между боковыми цепями аминокислотных остатков
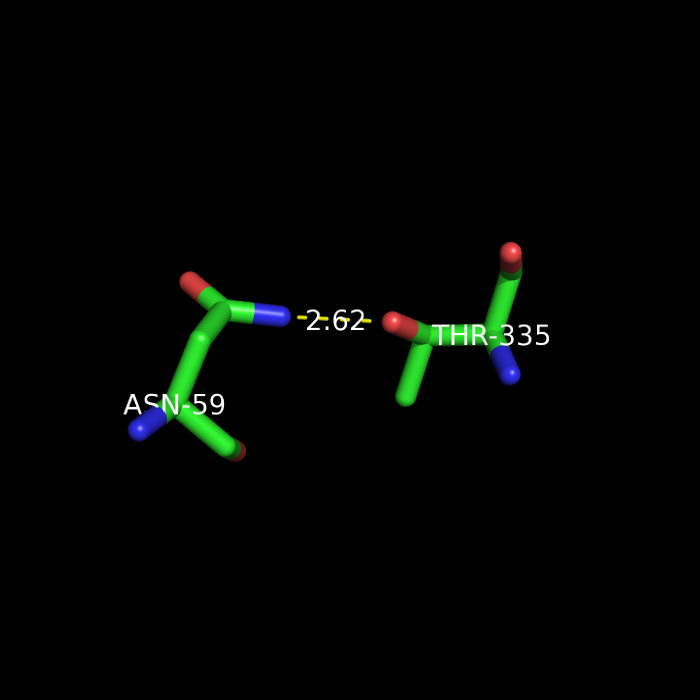
X0059-ASN ND2 X0335-THR OG1 2.62 SS 276 7.42 120.8 1.96 118.1 110.4 136
в) между боковой цепью одного остатка и остовным атомом другого
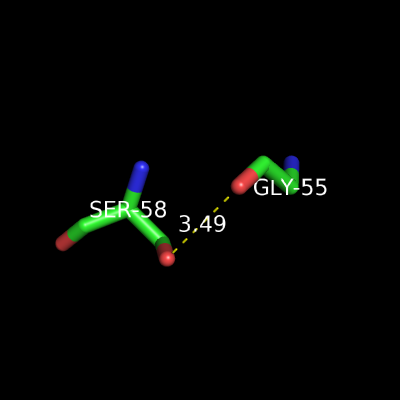
X0058-SER OG X0055-GLY O 3.49 SM 3 5.20 179.8 2.49 115.3 115.3 122
д) водородная связь между белком и этилен гликолем
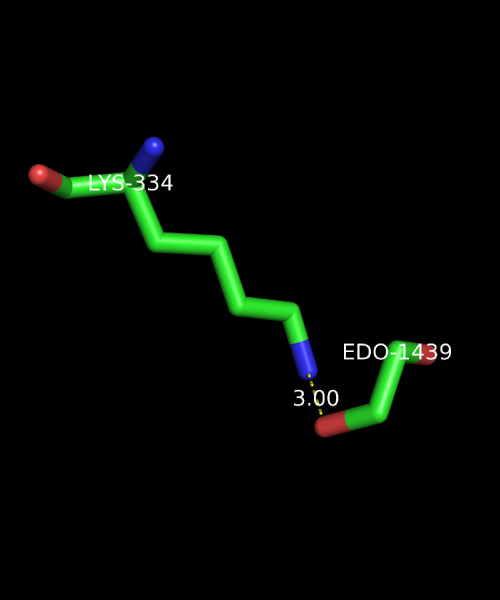
X0334-LYS NZ X1439-EDO O2 3.00 SH -2 -1.00 170.9 1.99 115.1 117.7 690
г) водяной мостик между различными остатками
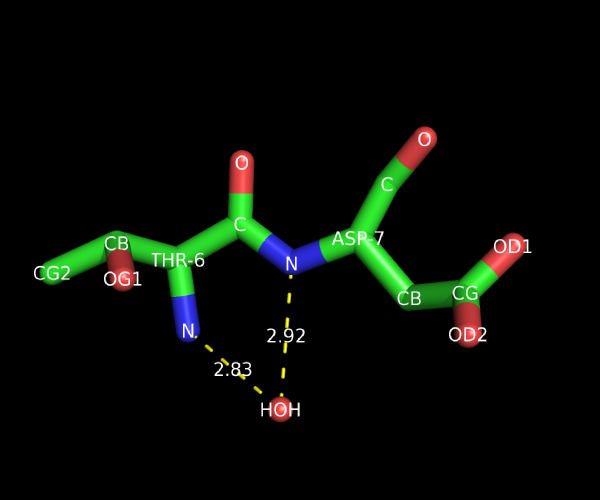
X0006-THR N X1717-HOH O 2.83 MH -2 -1.00 149.3 1.92 -1.0 -1.0 2
X0007-ASP N X1717-HOH O 2.92 MH -2 -1.00 150.6 2.01 -1.0 -1.0 4