Задание 1. Консервативные мотивы
Для работы мной был выбран домен Frizzled/Smoothened family membrane region. В выравнивании seed 71 последовательность, на redundance threshold 90% ни одна последовательность не выделяется, можно работать дальше.
На 90% выделяется некоторое количество консервативных позиций. Самый оформленный вероятный мотив(есть сразу несколько близко располагающихся консервативных позиций) имеет паттерн [FYLV]H...W..[PS], он был найден во всех последовательностях. Причем мотив в выравнивании встретился только единожды.
Далее по этому паттерну(переписанному как [FYLV]-H-x(3)-W-x(2)-[PS]) производился поиск в Swiss-prot с помощью PROSITE.
Был 1471 хит на 1468 последовательности. Похоже, что мотив все же в основном встречается в последовательности один раз, если встречается. То есть, вероятно, действительно нашлось что-то значимое, а не случайно встречающаяся последовательность(тогда бы такой мотив, возможно, чаще бы встречался в белках больше, чем по одному разу).
Задание 2. Специфичный для клады мотив
На основании выравнивания из прошлого задания построено дерево методом Neibhour-joining. Далее я выбрал кладу(в нее вошло 12 последовательностей) и выделил ее в отдельное выравнивание. Даже на identity treshold = 100% в этом выравнивании выделился мотив с паттерном G[I,T]CFV(потом поставил порог чуть ниже, во всех случаях, кроме одного, во второй позиции паттерна стояла T, поэтому записываю именно так). В изначальном паттерн встретился 15 раз. Кроме членов выбранной клады, он есть в A0A1X7VRK9_AMPQE, F7AFK4_CIOIN и H2Y9A5_CIOSA. Они не составляют с выбранной кладой другой клады и разбросаны друг от друга по дереву. Получается, мотив для клады не специфичен, хотя и встречается вне ее редко.
Задание 3. PSI-BLAST
Для проведения PSI-BLAST я выбрал белок с идентификатором P39450. Это S-(гидроксиметил)глутатион дегидрогеназа из бактерии Photobacterium damsela subsp. piscicida. Этот фермент может окислять спирты и NADH-зависимо восстанавливать S-нитрозоглутатион(судя по описанию на Uniprot).
Для этого белка я пытался найти семейство гомологов с помощью PSI-BLAST.Надо сказать, за 6 попыток стабилизации так и не произошло, над- и подпороговые находки были очень близко друг к другу по E-value. Да и количество находок огромное. Возможно, чтобы ограничить семейство белков, в этом случае надо выставлять более жесткий порог на E-value(проверил порог 0.001, не помогло).
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 470 | Q9ZGI4.1 | 5.00E-03 | S4W4F3.1 | 5.00E-03 |
| 2 | 851 | B2SHS9.1 | 4.00E-03 | Q5KWK4.1 | 6.00E-03 |
| 3 | 955 | D1ZA70.1 | 5.00E-03 | C0Q8N5.1 | 5.00E-03 |
| 4 | 1584 | A8AAV7.1 | 5.00E-03 | Q1I4C5.1 | 5.00E-03 |
| 5 | 2811 | A1AGH3.1 | 5.00E-03 | Q4L711.1 | 5.00E-03 |
| 6 | 4185 | B4TJX2.1 | 5.00E-03 | A1SBV0.1 | 5.00E-03 |
Задание 4. Поиск мотивов с помощью MEME
С помощью MEME на kodomo производился поиск мотивов в последовательностях из выравнивания seed, с которым я работал в заданиях 1-2(удалил гэпы). Команда: meme ./term4/fixed.fasta -o meme_res -minw 4 -maxw 30 -nmotifs 3 -o meme_pr10 (то есть ищу три мотива длиной 4-30). Здесь представлен результат работы MEME. Все три мотива нашлись E-value ниже порога. Если обратить внимание на второй мотив, то можно заметить, что он содержит паттерн мотива из первого задания. Отлично, подтвердили наличие мотива двумя способами.
Задание 5. Сайты GATC
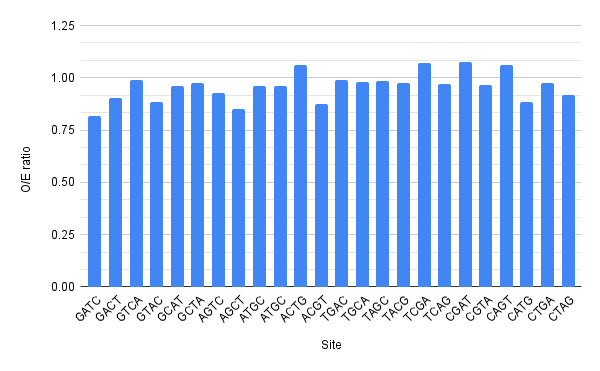
Создал файл со всеми возможными сайтами длины 4, получаемыми перестановками G,A,T,C. Скачал последовательность генома Teredinibacter purpureus. Далее с помощью команды cbcalc -s ~/term4/GATC.txt -M -o ~/term4/result_cbcalc.tsv ~/term4/GCF_014217335.1_ASM1421733v1_genomic.fna на kodomo получил таблицу, по которой построил гистограмму контрастов obs/exp(Рис.2). Частота встречаемости сайта GATC в геноме не очень сильно меньше ожидаемой(0.816).