ЗАДАНИЕ ВЫПОЛНЕНО СОВМЕСТНО С МИХАИЛОМ ПОПОВЫМ
Задание 1. Описание сигнала(oriC)
В процессе репликации есть абсолютно неоходимаю начальная стадия - инициация репликации. В ходе инициации белковые коплексы связвыают определенные части последовательностей нуклеиновой кислоты - точки начала репликации.
Как правило, для прокариот такая точка одна на одну молекулу н.к. (хоромосому).
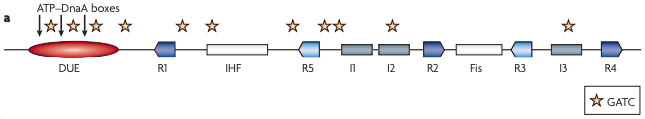
В данном практикуме рассмотрим бактериальную точку инициации репликации - OriC - описанную на модельном организме Escherichia coli. По длине - 245 нуклеотидов, имеет две функциональные области: часть, специфично связывающаяся с DnaA (фактор инициации репликации), и часть первичного расплетания ДНК [1].
Область связывания DnaA состоит из нескольких 9-членных повторов [2]:
Область первичного расплетания ДНК DUE (DNA unwinding element) состоит из трёх АТ-богатых 13-членных повтора, консенсус: [GATCTNTTNTTTT]. Связывается комплексом DnaA-ATP перед инициацией.
Вдобавок ко всему этому OriC содержит 13 палиндромных сайтов метилирования GATC по N6-положению аденина. Для инициации репликации требуется метилированность обеих цепей [2].
Задание 2. Описание сервиса DoriC
DoriC 12.0 – база данных прокариотических ориджинов. В основном в бд хромосомы, но есть и плазмиды. В DoriC можно искать по собственным DoriC ID, RefSeq ID, таксономии. Также можно искать ориджины с помощью BLAST(на ввод подается нуклеотидная последовательность)
У записей есть так называемые первичная и вторичная страницы. На первичной странице есть описание молекулы ДНК(хромосома, топология, организм, уровень сборки, размер хромосомы, GC-состав и т.д.). Также отображается график z-curve(особый график, позволяющий отобразть содержание всех типов нуклеотидов с их расположением в молекуле ДНК), содержащий информацию о расположении ориджина, плотности расположения DnaA-box,
Во вторичной записи об ориджине содержится его идентификатор DoriC; идентификаторы молекулы ДНК, к которой он принадлежит; длину ориджина; AT-содержание ориджина; положение ориджина в молекуле ДНК; карту ориджина(с положением различных элементов(например сайтов посадки DnaA)); последовательность ориджина(с аннотацией); описание содержащихся повторяющихся мотивов; распределение различных генов и типов нуклеотидов между лидирующей и отстающей цепями и прочее.
Задание 3. Поиск ориджина
Искали ориджин поиском по таксономии(а именно выбранную Муравлевым А. в 1 семестре бактерию Teredinibacter purpureus). Выдалось две записи, выбрали запись с уровнем сборки Complete(ORI97024506). В записи(на первичной странице) нашли соответствующий ориджин
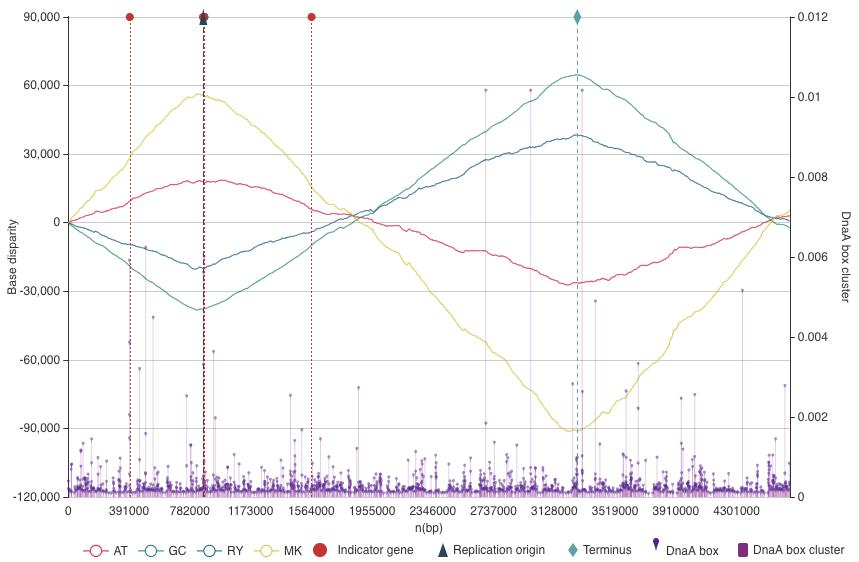
Ориджин расположен в хромосоме длиной в 4643613 нуклеотида. Координаты ориджина – [869872:870591]. На Рис.1 показана z-curve и отображение плотности расположения DnaA-боксов и ориджина с некоторой дополнительной информацией.
Ориджин имеет длину в 720 нуклеотидов, есть три различных представленных повторяющихся мотива.
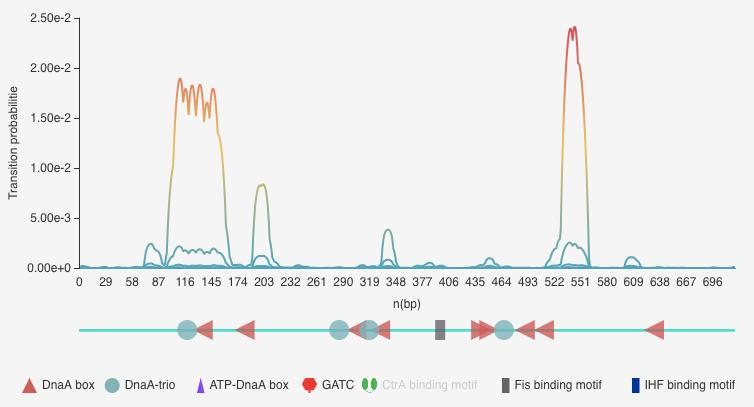
На Рис.2 представлена карта ориджина с функциональными элементами и график, который отображает вероятность изменения вторичной структуры ДНК для каждой пары оснований. Видно два крупных пика, правый, судя по последовательности, совпадает с АТ-богатым участком, где, вероятно, и происходит плавление ДНК.
Также мы решили проверить, найдется ли правильно ориджин в какой-либо последовательности с помощью встроенного BLAST. Для этого в BLAST сервиса загрузили последовательность хромосомы Teridinibacter turnerae из соответствующей сборки. Сервис сработал правильно, со 100% совпадением нашлись только два ориджина(ORI97076196 и ORI97024506), принадлежащие Teredinibacter purpureus.