Сборка de novo
1 Сборка генома
Команды использованные в данном практикуме:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/000/SRR4240360/SRR4240360.fastq.gz cat /mnt/scratch/NGS/adapters/* 1> adapters.fasta java -jar /usr/share/java/trimmomatic.jar SE -phred33 -threads 15 \ SRR4240360.fastq.gz SRR4240360_no_adapters.fastq.gz \ ILLUMINACLIP:adapters.fasta:2:7:7 2>>logs/trimmomatic.log java -jar /usr/share/java/trimmomatic.jar SE -phred33 -threads 15 \ SRR4240360_no_adapters.fastq.gz SRR4240360_good.fastq.gz \ TRAILING:20 MINLEN:32 2>>logs/trimmomatic.log velveth ./SRR4240360_velvet 31 -fastq.gz -short SRR4240360_good.fastq.gz \ &>>logs/velvet.log velvetg ./SRR4240360_velvet &>>logs/velvet.log
В рамках данного практикума производилась сборка de novo генома бактерии Buchnera aphidicola из чтений с кодом доступа SRR4240360, полученных из базы EMBL (первая команда). Данные чтения содержат в себе последовательности адаптеров, поэтому предварительно был получен файл, содержащий все возможные адаптеры, используемые прибором Illumina (вторая команда). После удаления адаптеров из чтений (третья команда) количество чтений сократилось с 8 254 632 до 8 212 774, при этом 41 858 (0.51%) оказались остатками адаптеров. Далее было призведено удаление чтений по качеству (четвёртая команда), в результате чего было удалено 297 300 чтений. Размеры файлов чтений до и после очистки:
SRR4240360.fastq.gz 194M SRR4240360_no_adapters.fastq.gz 193M SRR4240360_good.fastq.gz 184M
Далее из оставленных чтений были получены k-меры, длиной 31 bp (пятая команда). Эти k-меры были использованы для сборки контигов (шестая команда).
2 Аналих полученных контигов
Для полученной сборки N50 составил 43 100 bp. Самые длинные контиги имеют следующие характеристики:
| Имя контига | Длина (bp) | Покрытие |
|---|---|---|
| NODE_1_length_113474_cov_33.525459 | 113 574 | 33,53 |
| NODE_5_length_83603_cov_33.646065 | 83 633 | 33,65 |
| NODE_4_length_64155_cov_35.847324 | 64 185 | 35,85 |
Контиги с очень маленьким и очень большим покрытием имеют длину менее 61 bp, поэтому они не включены в файл configs.fa. Среди контигов, которые есть в файле contigs.fa наибольшее покрытие у контига NODE_40_length_69_cov_109.391304, наименьшее – у контига NODE_565_length_31_cov_1.612903.
Далее самые длинные контиги из Таблицы 1 были выравнены с референсным геномом Buchnera aphidicola (GenBank/EMBL AC — CP009253). Для этого был использован алгоритм megablast с длиной слова 28. Также для получения дополнительной карты локального сходства был запущен алгоритм blastn с длиной слова 11.
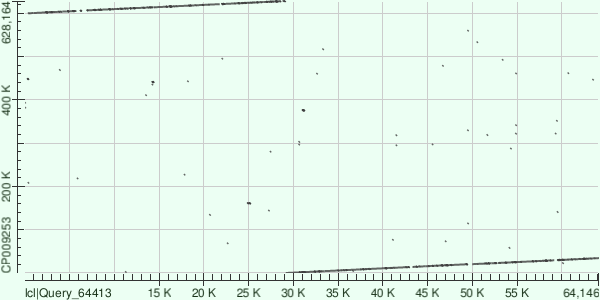
Контиг NODE_1_length_113474_cov_33.525459 хорошо выровнялся с прямой цепью референсного генома. При чём крупных делеций или вставок не наблюдается, есть только участки, содержащие большее сисло нуклеотидных замен, и потому не найденные алгоритмом megablast.
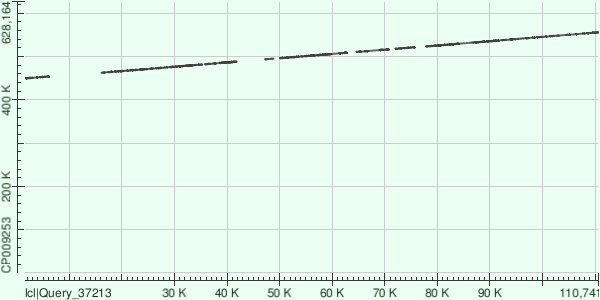
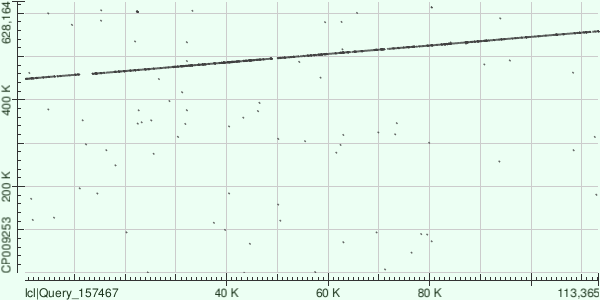
| Процент идентичности | Число однонуклеотидных различий | Число гэпов | Координаты в референсном геноме | |
|---|---|---|---|---|
| Начало | Конец | |||
| 81,433 | 3 488 | 399 | 528 794 | 550 219 |
| 80,866 | 955 | 97 | 550 361 | 555 905 |
| 77,020 | 1 490 | 168 | 467 412 | 474 667 |
| 75,609 | 1756 | 259 | 500 370 | 508 806 |
| 78,455 | 1150 | 143 | 510 438 | 516 539 |
| 76,895 | 1 104 | 161 | 523 105 | 528 679 |
| 77,009 | 991 | 135 | 462 496 | 467 421 |
| 74,078 | 1309 | 241 | 481 997 | 488 106 |
| 74,125 | 1 295 | 196 | 474 844 | 480 660 |
| 75,465 | 1 009 | 112 | 449 411 | 454 069 |
| 77,261 | 761 | 79 | 517 766 | 521 500 |
| 75,249 | 917 | 121 | 496 111 | 500 325 |
| 80,058 | 263 | 13 | 493 487 | 494 864 |
| 82,216 | 102 | 18 | 480 874 | 481 545 |
| 89,167 | 8 | 4 | 495 033 | 495 148 |
Контиг NODE_5_length_83603_cov_33.646065 хорошо выровнялся с прямой цепью референсного генома. Крупных делеций или вставок нет, но на фрагменте контига 15 kb – 32 kb наблюдается высокая вариативность последовательности.
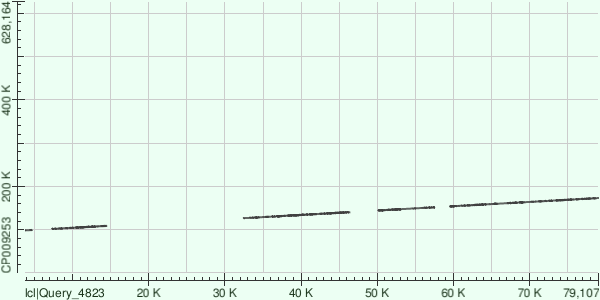
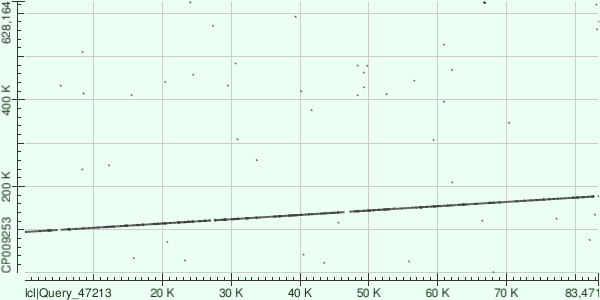
| Процент идентичности | Число однонуклеотидных различий | Число гэпов | Координаты в референсном геноме | |
|---|---|---|---|---|
| Начало | Конец | |||
| 74.950 | 2711 | 430 | 127825 | 140555 |
| 77.804 | 1549 | 191 | 153752 | 161738 |
| 77.747 | 1434 | 178 | 144368 | 151796 |
| 76.533 | 1492 | 188 | 101712 | 108876 |
| 79.589 | 891 | 92 | 161898 | 166752 |
| 76.216 | 1391 | 138 | 166750 | 173180 |
| 83.736 | 184 | 9 | 126623 | 127815 |
| 81.132 | 161 | 8 | 98408 | 99303 |
Контиг NODE_4_length_64155_cov_35.847324 хорошо выровнялся с прямой цепью референсного генома. При чём данный контиг содержит в себе точку, которая была взята в качестве начала хромосомы в референсном геноме.
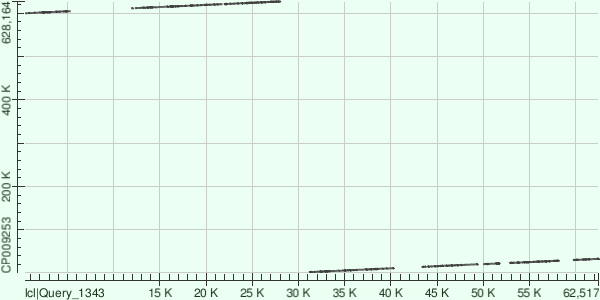
| Процент идентичности | Число однонуклеотидных различий | Число гэпов | Координаты в референсном геноме | |
|---|---|---|---|---|
| Начало | Конец | |||
| 78,380 | 1 738 | 201 | 2 004 | 11 103 |
| 79,211 | 1 350 | 144 | 613 658 | 620 926 |
| 78,201 | 930 | 114 | 599 832 | 604 795 |
| 75,782 | 1 247 | 207 | 621 055 | 627 104 |
| 76,551 | 1 055 | 166 | 23 067 | 28 363 |
| 85,253 | 299 | 23 | 17 962 | 20 182 |
| 75,976 | 687 | 68 | 14 727 | 17 919 |
| 77,422 | 543 | 71 | 30 013 | 32 745 |
| 81,524 | 291 | 41 | 20 358 | 22 183 |
| 77,900 | 395 | 42 | 611 633 | 613 671 |
| 82,218 | 76 | 8 | 13 994 | 14 465 |
| 79,461 | 59 | 2 | 611 229 | 611 524 |
3 Сравнение разных сборок
С исполбзованием того же самого файла с чтениями была получена сборка программой velvet с длиной k-мера 27 bp. Также были получены три сборки программой SPAdes с длинами k-меров 27 bp и 31 bp. Также программа SPAdes была запущена без указания длины k-меров. По умолчанию, последовательно используется несколько длин k-меров, после чего формируется окончательная сборка. Параметр --only-assembler позволяет проводить сборку без предварительной коррекции.
velveth ./SRR4240360_velvet_27 27 -fastq.gz -short SRR4240360_good.fastq.gz \ &>>logs/velvet_27.log velvetg ./SRR4240360_velvet_27 &>>logs/velvet_27.log spades.py -s SRR4240360_good.fastq.gz -k 27 --only-assembler -o SRR4240360_SPAdes \ 2>> ./logs/SPAdes.log spades.py -s SRR4240360_good.fastq.gz -k 31 --only-assembler -o SRR4240360_SPAdes \ 2>> ./logs/SPAdes.log spades.py -s SRR4240360_good.fastq.gz --only-assembler -o SRR4240360_SPAdes \ 2>> ./logs/SPAdes.log
Далее полученные сборки были сравнены по таким параметрам, как количество контигов, N50, длина самого длинного контига и его покрытие. Сравнение показывает, что при уменьшении длины k-мера до 27 качество сборки, полученной программой velvet заметно падает. При этом сборка, полученная программой SPAdes не настолько сильно зависит от длины k-мера и по качеству превосходит сборку, полученную velvet. Также хочу отметить, что программа SPAdes выдаёт все длины в bp, в отличие от velvet, где длины привидены в количестве k-меров и для получения корректных значений длин к ним нужно прибавлять k-1.
| Сборка | Количество контигов |
N50 | Самый длинный контиг (bp) |
Покрытие самого длинного контига (%) |
|---|---|---|---|---|
| velvet k=31 | 565 | 43 100 | 113 504 | 33,53 |
| velvet k=27 | 3 262 | 15 848 | 83 755 | 50,01 |
| SPAdes | 502 | 83 739 | 174 223 | 17,20 |
| SPAdes k=31 | 108 | 83 633 | 113 504 | 27,69 |
| SPAdes k=27 | 258 | 341 021 | 341 021 | 51,92 |