Нуклеотидный BLAST
1 Применение BLAST для контиг
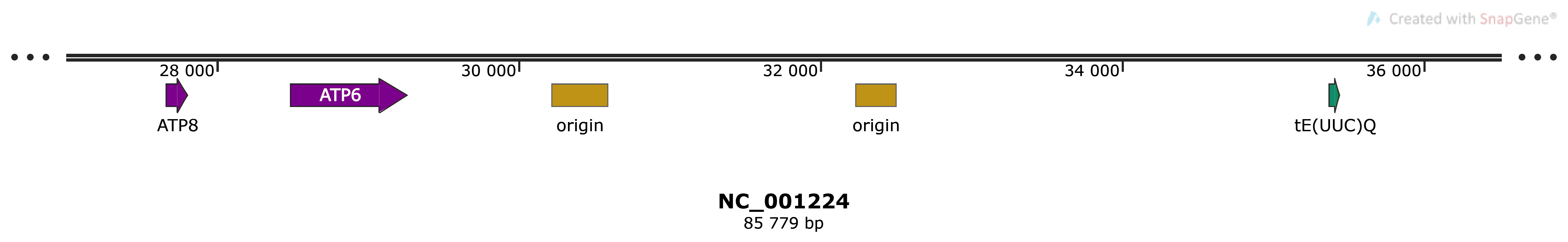
Для работы выбрана мтДНК Saccharomyces cerevisiae с идентификатором NC_001224.1. Длина этого контига составляет 85 779 bp, поэтому для дайнешей работы выбран фрагмент длиной 9,5 kb с координатами 27 001..36 500. На этом фрагмете находятся гены субъединиц 6 и 8 АТФ-синтазы, тРНК глутамина с антикодоном UUC и два ориджина репликации. Вырезанный фрагмент сохранён в файл contig.fna.
Для поиска BLAST по эукариотическим последовательностях для данного фрагмента, были исключены из поиска виды из царства Fungi (taxid:4751). При запуске blastn использованы следующие параметры: длина слова – 11; E-value – 0.05; максимальное число находок – 5 000. По результатам запроса было пулучено 4 991 находок, среди которых были синтетические конструкции, содержащие гены субъединиц АТФ-синтазы, выравнивания этих генов из запроса с последовательностями из разных организмов. Про остальные результаты выдычи нельзя сказать, что они соответствуют каким-либо особенностям последовательности запроса.
При запуске megablast использованы следующие параметры: длина слова – 28; E-value – 0.05; максимальное число находок – 100. По результатам запроса было пулучено 10 находок, среди которых были синтетические конструкции, содержащие гены субъединиц АТФ-синтазы и выравнивание фрагмета гена субъеницы 6 с последовательностью из Apteryx australis mantelli (Северный бурый киви).
При запуске blastx использованы следующие параметры: длина слова – 6; E-value – 0.05; максимальное число находок – 5 000; генетический код: Yeast mitochondrial (3). По результатам запроса было пулучено 5 000 находок, представляющих собой субъединицу A АТФ-синтазы из разных организмов.
При запуске tblastx даже при использовании максимального числа находок – 10 сервер выдал ошибку о нехватке вычислительных ресурсов.
Разные алгоритмы blast используются для решения разных задач. С помощью blastn можно искать гомологи генов, некдирующих белки. С помощью megablast можно картировать фрагмент на геном, либо искать близкородственные штаммы. С помощью blastx возможно предсказывать CDS на последовательности запроса.С помощью tblastx можно найти похожие непредсказанные белки.
2 Поиск в геноме эукариота генов основных рибосомальных РНК по далекому гомологу
С помощью локально установленного BLAST была создана локальная база данных на основе генома. Для этого выполнена команда:
makeblastdb -in GCF_000146045.2_R64_genomic.fna -dbtype nucl
Далее с помощью команд
blastn -task blastn -query query1.fna -db GCF_000146045.2_R64_genomic.fna -out 1.out -outfmt 7
blastn -task blastn -query query2.fna -db GCF_000146045.2_R64_genomic.fna -out 2.out -outfmt 7
был проведён локальный поиск BLAST.
Запросы query1.fna и query2.fna содержат в себе последовательности 16s рРНК и 23s рРНК Escherichia coli соответственно. Для поиска использован алгоритм blastn, так как последовательность не является кордирующей белок, а организмы не близкородственные. Файлы с выдачей: 1.out и 2.out. Запросы дали 9 и 25 находок соответственно. В результате работы blastn были найдены следующие гомологи гена 16s рРНК Escherichia coli: два гена 18s рРНК на хромосоме XII и ген 15s рРНК на мтДНК. Ещё одна находка соответствует гену гипотетический белка на хромосоме VII. Для гена 23s рРНК Escherichia coli были найдены ген 21s рРНК на мтДНК, два гена 25s рРНК на хромосоме XII. Остальные находки либо накладываются на аннотированные белки, либо соответствуют неаннотированным участкам генома.