Трансмембранные белки
Мой белочек
В рамках данного практикума мне был выдан белок YUEB_BACSU. Этот белок, вероятно, является частью системы секреции ESX у Bacillus subtilis. Также этот белок играет роль в связывании бактериофага SPP1 с клеткой. Для данного белка были скачаны последовательность и трёхмерная структура, предсказанная с помощью AlphaFold.
Знакомство с базой данных OPM
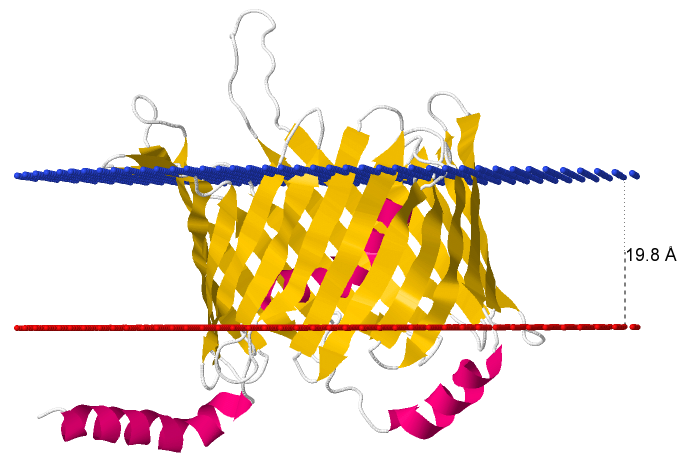
В базе данных OPM был найден белок, содержащий β‑бочонок: субъединица рецептора импорта митохондрий Tom40 из организма Neurospora crassa. Идентификатор PDB: 5O8O, идентификатор Uniprot: TOM40_NEUCR. Красным цветом показана положительно заряженная сторона мембраны (направлена в межмембранное пространство митохондрии). Синим цветом показана отрицательно заряженая сторона мембраны (направлена в сторону цитоплазмы).
Толщина гидрофобной части белка в мембране:
Количество трансмембранных участков:
Координаты трансмембранных участков:
Среднее количество остатков
в одном β‑тяже белка:
Локализация белка:
19.8 Å
19
1(68‑74), 2(81‑86), 3(95‑100), 4(106‑112), 5(119‑124), 6(132‑136), 7(143‑150), 8(156‑166), 9(173‑180), 10(189‑196), 11(206‑214), 12(219‑226), 13(231‑237), 14(245‑251), 15(268‑277), 16(279‑286), 17(293‑298), 18(305‑311), 19(323‑330)
6
Внешняя мембрана митохондрии
DeepTMHMM: Предсказание трансмембранных элементов по последовательности белка
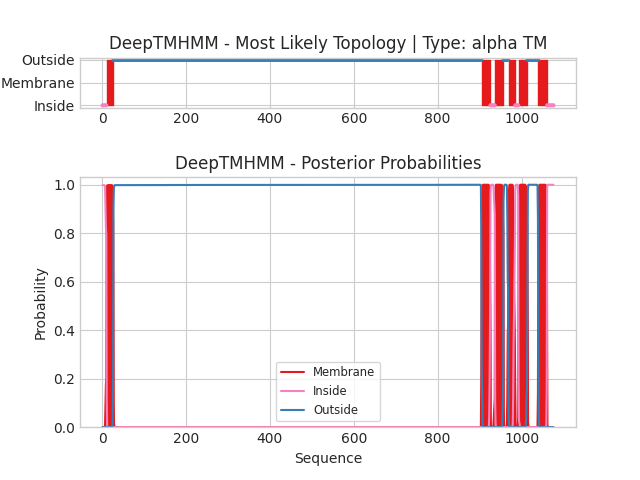
Запуск сервиса для α‑спирального белка
Текстовая выдача DeepTMHMM для белка YUEB_BACSU доступна по ссылке. На графике по оси абсцисс отложены координаты белка (номер аминокислотного остатка). По оси ординат отложена вероятность определённой локализации белка. Верхний рисунок показывает предсказанную топологию в соответствии с вероятностями на графике. Красным цветом показаны трансмембранные участки, розовым и голубым – локализация со стороны внутренней и внешней мембраны соответственно. Для белка YUEB_BACSU предсказано шесть трансмембранных участков, причём и N‑конец, и C‑конец были определён как внутриклеточные.
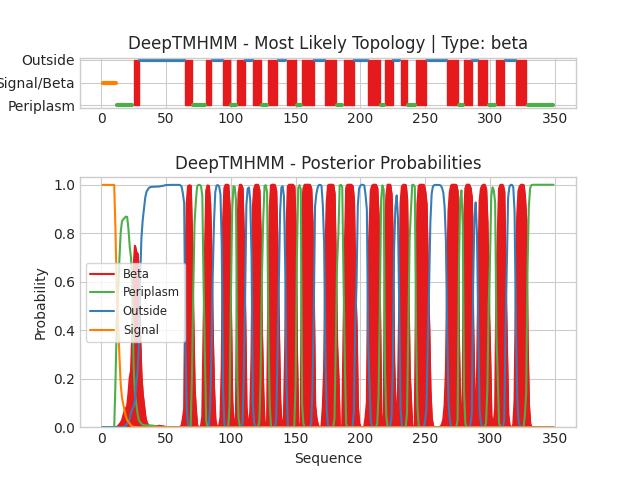
Запуск сервиса для β‑листового белка
Текстовая выдача DeepTMHMM для белка TOM40_NEUCR доступна по ссылке. На графике по оси абсцисс отложены координаты белка (номер аминокислотного остатка). По оси ординат отложена вероятность определённой локализации белка. Верхний рисунок показывает предсказанную топологию в соответствии с вероятностями на графике. Красным цветом показаны трансмембранные участки, голубым – локализация со стороны внешней мембраны, зелёным – со стороны периплазмы, оранжевым – сигнал локализации (для данного белка – митохондриальный). Для белка TOM40_NEUCR предсказано двадцать трансмембранных участков, причём и N‑конец, и C‑конец были определён как периплазматические.
Интересно, что первый предсказанный трансмембранный участок (имеющй вероятность заметно меньше 1) в действительности β‑листом не является, однако соответствует α‑спирали расположенной в межмембранном пространстве митохондрии. При этом участок расположенный внутри поры, образованной бочонком алгоритм определил как внешний. Остальные предсказанные участки в целом соответствуют действительности.
PPM: Предсказание положения выданного белка в мембране
Для белка YUEB_BACSU был запущен алгоритм PP 3.0 со следующими параметрами:
Type of membrane:
Allow curvature:
Topology (N‑ter):
Gram‑positive bacteria inner membrane (ибо B. subtilis является таковой)
no
in (на основе выдачи DeepTMHMM)
Красным цветом показана положительно заряженная сторона мембраны (направлена наружу). Синим цветом показана отрицательно заряженая сторона мембраны (направлена в сторону цитоплазмы).
Толщина гидрофобной части белка в мембране:
Количество трансмембранных участков:
Координаты трансмембранных участков:
Среднее количество остатков
в одном β‑тяже белка:
Локализация белка:
30.9 ± 1.4 Å
6
1(8‑29), 2(908‑929), 3(933‑955), 4(967‑992), 5(993‑1011), 6(1039‑1060)
21
Мембрана грам‑положительной бактерии
Сравнение алгоритмов предсказания трансмембранных спиралей
Трансмембранные участки, предсказанные для белка YUEB_BACSU разными алгоритмами похожи, однако DeepTMHMM выдаёт меньшую длину трасмембранных участков. Стоит отметить, что положение каждого остатка в предсказанной с помощью AlphaFold структуры характеризуется своим доверительным score'ом (насколько точно верно предсказание). Участки трансмембранных α-спиралей обладают высоким весом, в то время как низким весом обладает лишь часть белка, нраходящаяс за пределпми мембраны. Такимом образом достоверность модели не могла оказать влияние на результаты предсказания сервера PPM.