Поиск и визуализация ортологов и паралогов
Составление списка гомологичных белков, включающих паралоги
Для дальнешей работы были найдены гомологи белка CLPX_ECOLI в бактериях со следующими мнемониками:
Arthrobacter sp.
Leifsonia xyli
Mycobacterium leprae
Mycobacterium tuberculosis
Mycolicibacterium vanbaalenii
Rubrobacter xylanophilus
Streptomyces avermitilis
ARTS2
LEIXX
MYCLE
MYCTU
MYCVP
RUBXD
STRAW
Скачанные протеомы бактерий были объедины в файл proteomes.fasta, и был произведён поиск гомологов с помощью blastp:
cat ./proteomes/*.fasta > proteomes.fasta
makeblastdb -dbtype prot -in proteomes.fasta
blastp -query P0A6H1.fasta -evalue 0.001 -outfmt 7 -db proteomes.fasta 1>> out.txt
В результате были получены следующие 20 находок:
CLPX_STRAW ATP-dependent Clp protease ATP-binding subunit ClpX
Q1AVT0_RUBXD ATP-dependent Clp protease ATP-binding subunit ClpX
CLPX_MYCVP ATP-dependent Clp protease ATP-binding subunit ClpX
CLPX_MYCTU ATP-dependent Clp protease ATP-binding subunit ClpX
CLPX_MYCLE ATP-dependent Clp protease ATP-binding subunit ClpX
CLPX_ARTS2 ATP-dependent Clp protease ATP-binding subunit ClpX
CLPX_LEIXX ATP-dependent Clp protease ATP-binding subunit ClpX
A0K1M3_ARTS2 ATPase AAA-2 domain protein
Q1AU05_RUBXD ATPase AAA-2
Q1AY82_RUBXD ATPase AAA-2
Q6ACQ0_LEIXX ATP-dependent zinc metalloprotease FtsH (EC 3.4.24.-)
A1TG29_MYCVP ATPase AAA-2 domain protein
RUVB_ARTS2 Holliday junction branch migration complex subunit RuvB (EC 3.6.4.12)
Q82QV8_STRAW Putative AAA family ATPase
A0JR82_ARTS2 ATP-dependent zinc metalloprotease FtsH (EC 3.4.24.-)
A0K236_ARTS2 AAA ATPase, central domain protein
Q82EE9_STRAW ATP-dependent zinc metalloprotease FtsH (EC 3.4.24.-)
A1TG43_MYCVP ATP-dependent zinc metalloprotease FtsH (EC 3.4.24.-)
Q82EB8_STRAW Putative ATP-dependent Clp protease
FTSH_MYCTU ATP-dependent zinc metalloprotease FtsH (EC 3.4.24.-)
Реконструкция и визуализация
Для реконструкции дерева найденных гомологов использована программа FastME со следующими параметрами:
Gamma distributed rates across sites
Starting tree
No refinement
Number of bootstrap replicates
No
BIONJ
100
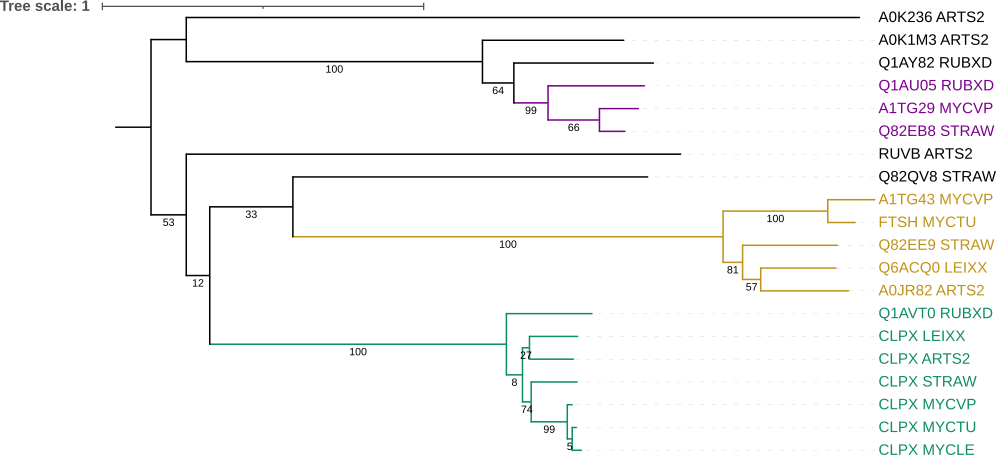
Скобочная форула полученного дерева доступна по ссылке.
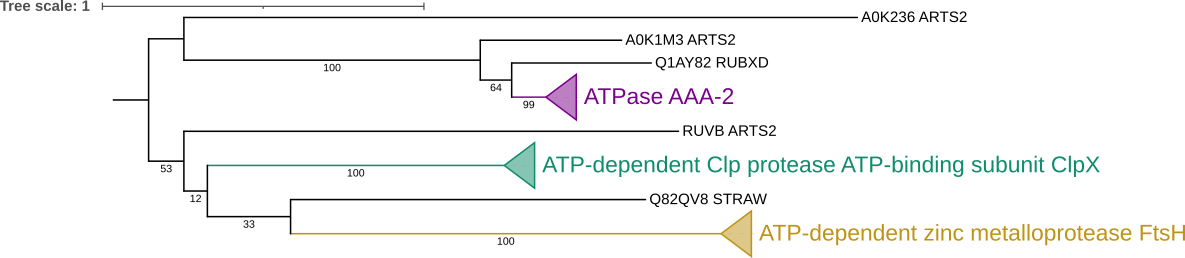
Полученное дерево было переукренено в среднюю точку. Также были добавлены значения бутстреп‑поддержки. На данном дереве можно выделить следующие пары ортологов: CLPX_MYCLE и CLPX_MYCTU, CLPX_LEIXX и CLPX_ARTS2, A0JR82_ARTS2 и Q6ACQ0_LEIXX; и следующие пары паралогов: A0K1M3_ARTS2 и A0K236_ARTS2, FTSH_MYCTU и CLPX_MYCTU, Q82EE9_STRAW и Q82EB8_STRAW. На основе описания описания белков можно выделить две ортологические группы: ATP-dependent zinc metalloprotease FtsH и ATP-dependent Clp protease ATP-binding subunit ClpX. Остальные клады содержат плохо описанные белки с неопределённой функцией, однако все они кроме RUVB_ARTS2 являются АТФазами. Среди них можно выделить ортологическую группу ATPase AAA-2, однако она содержит только три белка, поэтому не уверен, что её стоит рассматривать. В ортологическую группу ATP-dependent zinc metalloprotease FtsH попали только белки из пяти бактерии, при этом реконструированная филогения белков соответствует филогении бактерий. В ортологическую группу ATP-dependent Clp protease ATP-binding subunit ClpX попали белки из всех семи бактерий, однако реконструированная филогения неполностью совпадает с эталонной, но при этом большая часть нетривиальных ветвей правильные. (В целом данное несоответсвие можно было бы легко исправить переукоренением всей клады.)