ColabFold
АХТУНГ!!! Сессии PyMol являются слишком тяжелыми файлами, поэтому они хранятся в архиве и будут удалены с кодомо 25 декабря 2024 года. pse.zip
B. Helices
В данной части практикума сравнивались модели предсказанные для трёх последовательностей, которые отличаются между собой заменой, фрагмента цепи на пролины (рис. 1). Так как пролин не может быть донором водородных связей, а также имеет сильные ограничения на торсионные углы, можно ожидать, что в различающихся участках произойдёт нарушение вторичной структуры белка. Однако известно, что возможно формирование левозакрученной полипролиновой спирали с шагом в три аминокислоты [1].
В предсказанных структурах действительно наблюдаются деформации спиралей (рис. 2). В случае последовательности seqB наличие трёх пролинов подряд не приводит к полному исчезновению α-спирали, однако средний пролин является точкой изгиба. В то время как последовательность из 13 пролинов приводит к исчезновению участка α-спирали до неё (остатки 120 – 126), а также к формированию левозакрученной пролиновой спирали. Также стоит отметить, что в последовательности seqC разворот спирали E169N170R171 по каким-то причинам сломался. Ещё модели значительно различаются участками без вторичной структуры, в целом так и должно быть, однако участок 97 – 105 в последовательности seqC как-то неприлично сильно отличается от аналогичных участков в seqA и seqB. Если честно у меня есть подозрения, что предсказания для этих остатков похожи на напряжённые конформации. Для проверки этого предположения можно было бы измерить торсионные углы в модели и сопоставить с соответствующими картами Рамачандрана.
C. Proteases
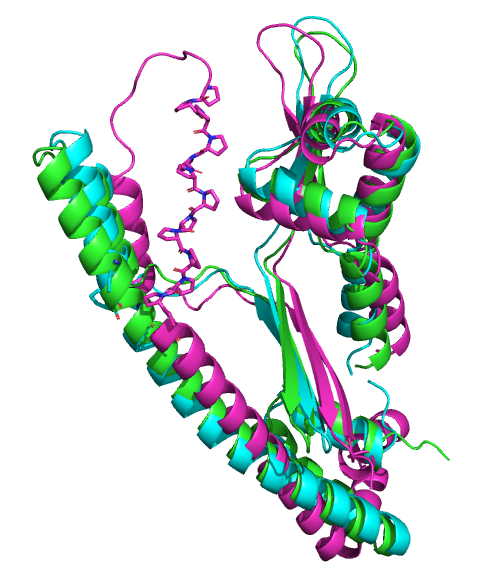
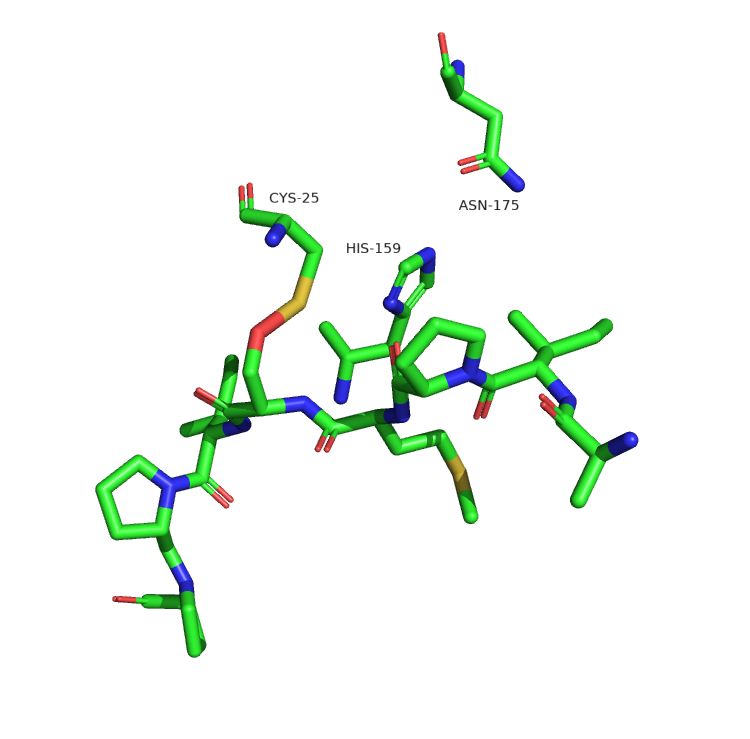
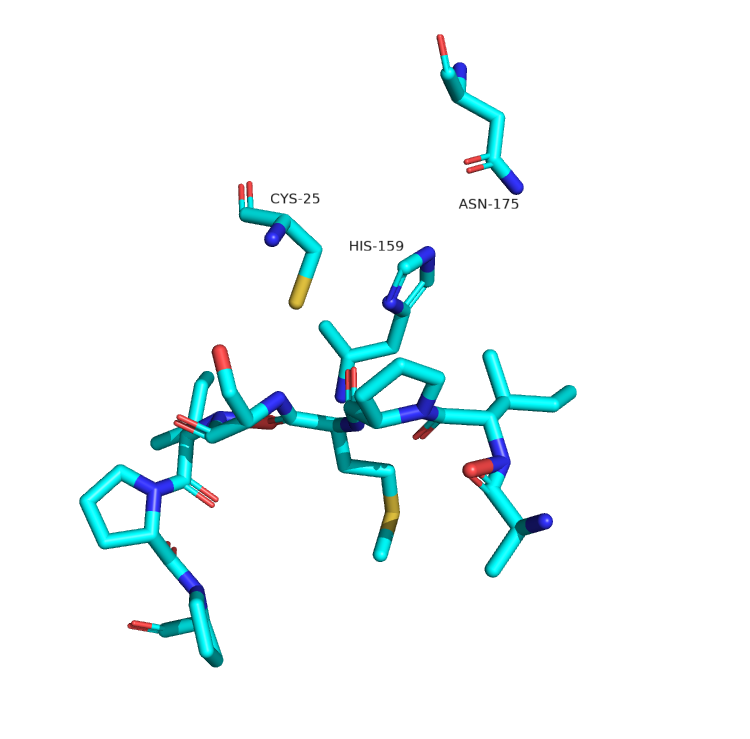
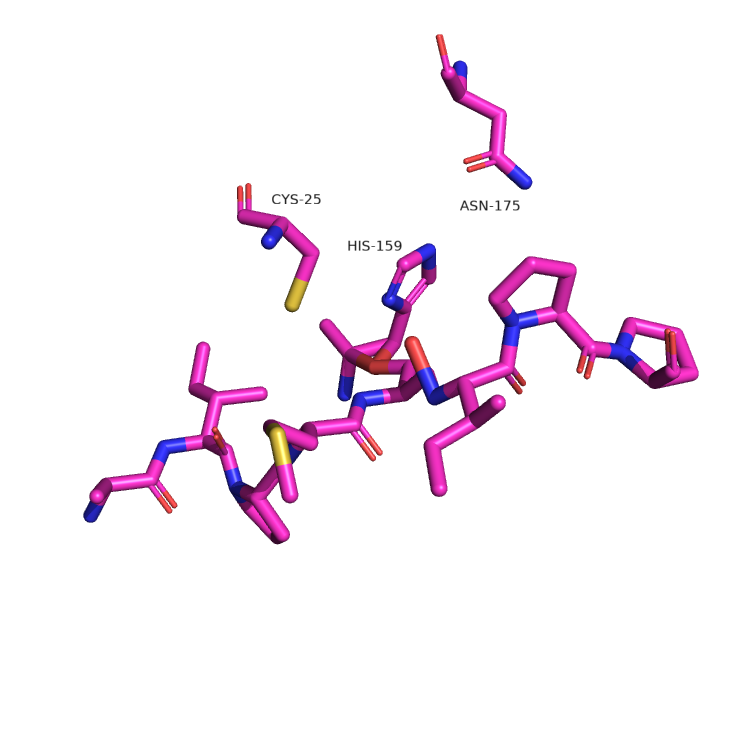
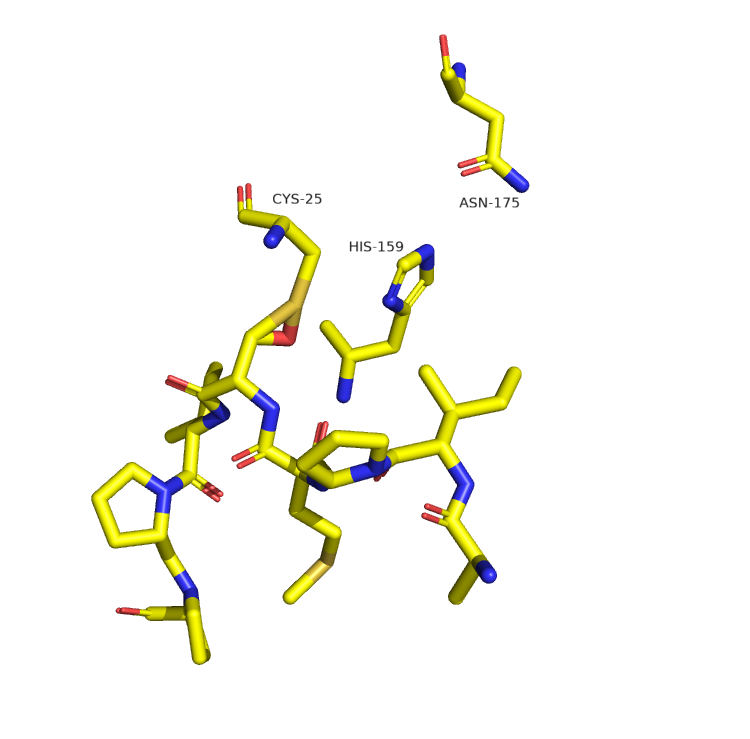
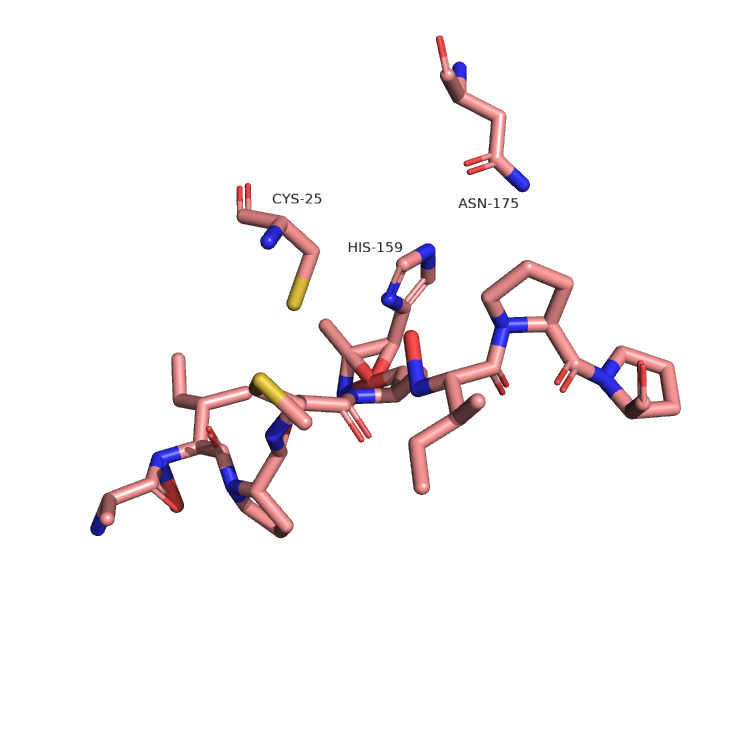
В данной части практикума ColabFold использовался для предсказания комплекса фермент-субстрат. Исследуемый фермент – папаин – является цистеиновой протеазой. Согласно литератeрным данным в гидролизе пептидной связи участвует каталитическая диада Cys-25 и His-159, также важную роль в стабилизации переходного состояния играет остаток Asn-175 [2]. Иследуемый субстрат представлен октопептидом AIPMSIPP, для которого показано узнавание ферментом и гидролиз связи между метионином и серином [3]. Известно, что фермент подвергается протеолизу в процессе созревания, поэтому для моделирования использована последовательность активной формы, а не энзимогена.

В результате работы ColabFold получается пять различных моделей. Предсказание структуры самого фермента одинаковое и по всей видимости достаточно хорошее для всех моделей. От структуры, полученной в эксперименте модель отличается наличием или отсутствием коротких фрагментов α-спирали внутри длинного участка без вторичной структуры (рис. 3). Если же посмотреть на предсказанную структуру субстрата, то можно только ужануться (рис. 4). А именно в модели длины связей и торсионные углы могут сильно отличаться от стандартных, вплоть то невозможных. Как следствие в моделях можно наблюдать тетраэдрические атомы азота в пролине, перекрывание кислорода карбонильной группы с водородом (не указывается в модели) аминогруппы другого остатка, близость радикалов субстрата к остову фермента. Также стоит отметить, что субстрат в модели может быть ориентирован как в направлении от N к C, так и в обратном (что не имеет биологического смысла), а с каталитически активным остатком Cys-25 сбилижено что угодно, кроме карбонильного углерода метионина, по которому должна идти нуклеофильная атака. Таким образом ColabFold угадал только в какую щель на поверхности фермента нужно поместить субстрат, но при этом совершенно не позволяет оценить реальное положение субстрата. В качестве контроля можно запустить моделирование с другим биологически релевантными субстратами, причём имеет смысл использовать более длинные субстраты, чтобы оценить способность модели находить сайт пептидазы в последовательности. Также можно использовать инфертированную последовательность субстрата, чтобы оценить всегда ли выбор направления субстрата в модели случайный.
СПИСОК ЛИТЕРАТУРЫ
- 1🠕 Adzhubei, A. A., Sternberg, M. J., & Makarov, A. A. (2013). Polyproline-II helix in proteins: structure and function. Journal of molecular biology, 425(12), 2100–2132.
- 1🠕 Shokhen, M., Khazanov, N., & Albeck, A. (2009). Challenging a paradigm: theoretical calculations of the protonation state of the Cys25-His159 catalytic diad in free papain. Proteins, 77(4), 916–926.
- 1🠕 Johnson, D., & Travis, J. (1979). The oxidative inactivation of human alpha-1-proteinase inhibitor. Further evidence for methionine at the reactive center. The Journal of biological chemistry, 254(10), 4022–4026.