!pip install prody
Requirement already satisfied: prody in /usr/local/lib/python3.10/dist-packages (2.4.1) Requirement already satisfied: numpy<1.24,>=1.10 in /usr/local/lib/python3.10/dist-packages (from prody) (1.23.5) Requirement already satisfied: biopython<=1.79 in /usr/local/lib/python3.10/dist-packages (from prody) (1.79) Requirement already satisfied: pyparsing in /usr/local/lib/python3.10/dist-packages (from prody) (3.2.0) Requirement already satisfied: scipy in /usr/local/lib/python3.10/dist-packages (from prody) (1.13.1) Requirement already satisfied: setuptools in /usr/local/lib/python3.10/dist-packages (from prody) (75.1.0)
import prody
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
import statsmodels.api as sm
!wget https://files.rcsb.org/download/7NW1.cif
xray_7NW1 = prody.parseMMCIF('./7NW1.cif')
nmr_7OVC = prody.parsePDB('7OVC')
--2024-12-18 17:36:10-- https://files.rcsb.org/download/7NW1.cif Resolving files.rcsb.org (files.rcsb.org)... 128.6.159.100 Connecting to files.rcsb.org (files.rcsb.org)|128.6.159.100|:443... connected. HTTP request sent, awaiting response... 200 OK Length: unspecified [application/octet-stream] Saving to: ‘7NW1.cif.1’ 7NW1.cif.1 [ <=> ] 368.33K 314KB/s in 1.2s 2024-12-18 17:36:11 (314 KB/s) - ‘7NW1.cif.1’ saved [377167]
@> WARNING Could not find _atom_site_anisotrop in lines. WARNING:.prody:WARNING Could not find _atom_site_anisotrop in lines. @> WARNING No anisotropic B factors found WARNING:.prody:WARNING No anisotropic B factors found @> 3051 atoms and 1 coordinate set(s) were parsed in 0.21s. DEBUG:.prody:3051 atoms and 1 coordinate set(s) were parsed in 0.21s. @> Connecting wwPDB FTP server RCSB PDB (USA). DEBUG:.prody:Connecting wwPDB FTP server RCSB PDB (USA). @> Downloading PDB files via FTP failed, trying HTTP. INFO:.prody:Downloading PDB files via FTP failed, trying HTTP. @> 7ovc downloaded (7ovc.pdb.gz) DEBUG:.prody:7ovc downloaded (7ovc.pdb.gz) @> PDB download via HTTP completed (1 downloaded, 0 failed). DEBUG:.prody:PDB download via HTTP completed (1 downloaded, 0 failed). @> 3147 atoms and 20 coordinate set(s) were parsed in 0.92s. DEBUG:.prody:3147 atoms and 20 coordinate set(s) were parsed in 0.92s.
Задание 1¶
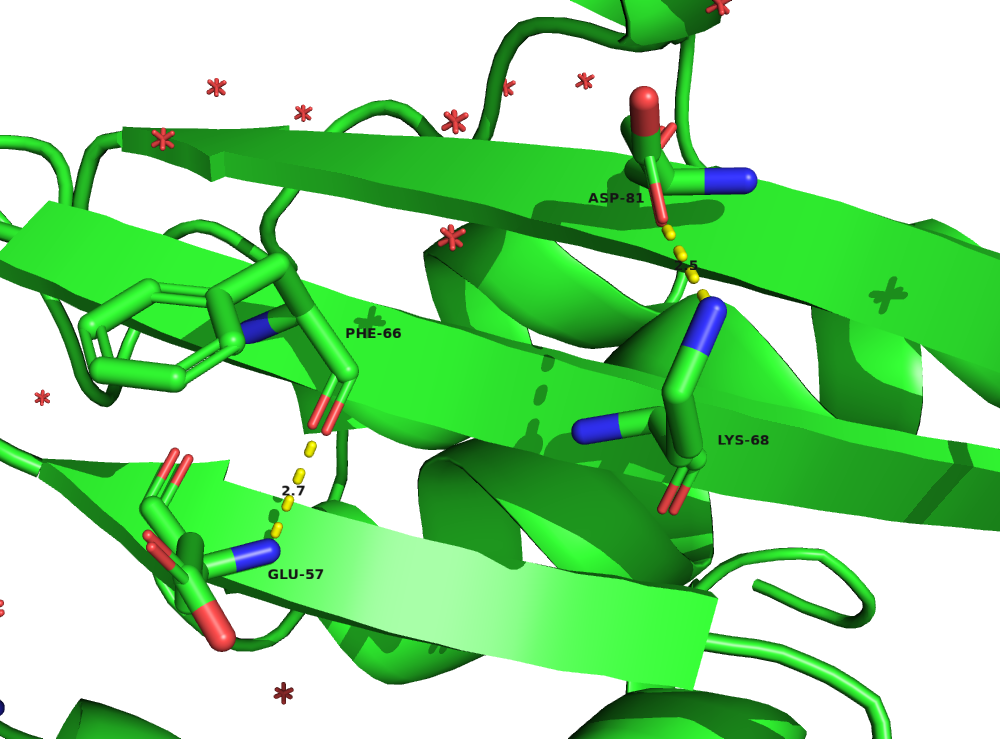
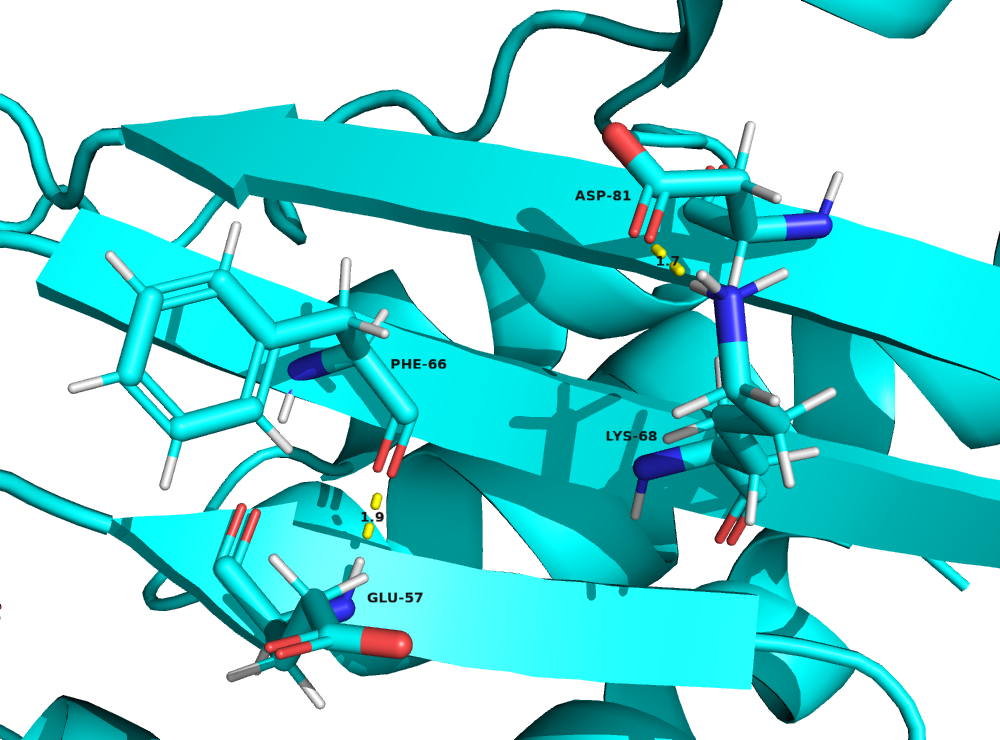
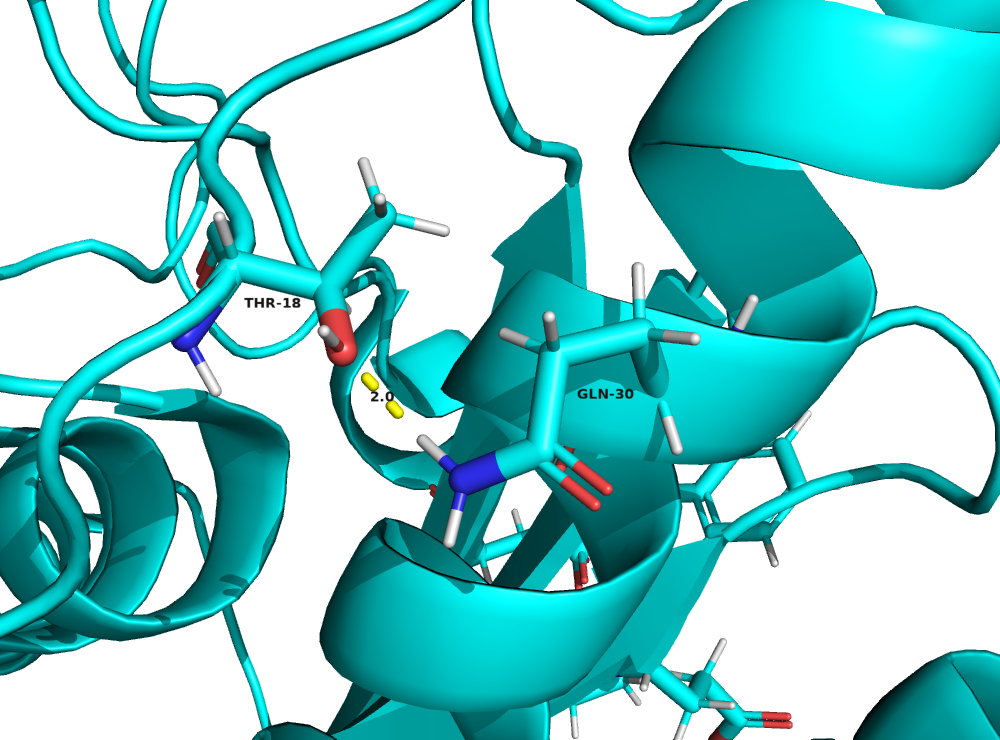
Для работы выбраны следующие водородные связи:
- между атомами остова в β-листе остатков GLU57 и PHE66
- между атомами боковых цепей в β-листе остатков LYS68 и ASP81
- между атомами на поверхности глобулы остатков GLN30 и THR18
distances = pd.DataFrame({"Донор" : [],
"Акцептор" : [],
"Расстояние в РСА" : [],
"% моделей ЯМР" : [],
"Минимальное расстояние в ЯМР" : [],
"Максимальное расстояние в ЯМР" : [],
"Медианное расстояние в ЯМР" : []})
distances.loc[0, "Донор"] = "GLU57"
distances.loc[0, "Акцептор"] = "PHE66"
aa1_xray = xray_7NW1.select('resnum 57 and name N and chain AAA')
aa2_xray = xray_7NW1.select('resnum 66 and name O and chain AAA')
distances.loc[0, "Расстояние в РСА"] = prody.calcDistance(aa1_xray, aa2_xray)
aa1_nmr = nmr_7OVC.select('resnum 57 and name N and chain A')
aa2_nmr = nmr_7OVC.select('resnum 66 and name O and chain A')
distances_0 = []
for glu, phe in zip(aa1_nmr.iterCoordsets(), aa2_nmr.iterCoordsets()):
distances_0.append(prody.calcDistance(glu,phe))
distances.loc[0, "Минимальное расстояние в ЯМР"] = min(distances_0)[0]
distances.loc[0, "Максимальное расстояние в ЯМР"] = max(distances_0)[0]
distances.loc[0, "Медианное расстояние в ЯМР"] = np.median(np.array(distances_0))
distances.loc[0, "% моделей ЯМР"] = len([i for i in distances_0 if i <= 3.5]) / 20 * 100
distances.loc[1, "Донор"] = "LYS68"
distances.loc[1, "Акцептор"] = "ASP81"
aa1_xray = xray_7NW1.select('resnum 68 and name NZ and chain AAA')
aa2_xray = xray_7NW1.select('resnum 81 and name OD1 and chain AAA')
distances.loc[1, "Расстояние в РСА"] = prody.calcDistance(aa1_xray, aa2_xray)
aa1_nmr = nmr_7OVC.select('resnum 68 and name NZ and chain A')
aa2_nmr = nmr_7OVC.select('resnum 81 and name OD1 and chain A')
distances_1 = []
for glu, phe in zip(aa1_nmr.iterCoordsets(), aa2_nmr.iterCoordsets()):
distances_1.append(prody.calcDistance(glu,phe))
distances.loc[1, "Минимальное расстояние в ЯМР"] = min(distances_1)[0]
distances.loc[1, "Максимальное расстояние в ЯМР"] = max(distances_1)[0]
distances.loc[1, "Медианное расстояние в ЯМР"] = np.median(np.array(distances_1))
distances.loc[1, "% моделей ЯМР"] = len([i for i in distances_1 if i <= 3.5]) / 20 * 100
distances.loc[2, "Донор"] = "GLN30"
distances.loc[2, "Акцептор"] = "ASP81"
aa1_xray = xray_7NW1.select('resnum 30 and name NE2 and chain AAA')
aa2_xray = xray_7NW1.select('resnum 18 and name OG1 and chain AAA')
distances.loc[2, "Расстояние в РСА"] = prody.calcDistance(aa1_xray,aa2_xray)
aa1_nmr = nmr_7OVC.select('resnum 30 and name NE2 and chain A')
aa2_nmr = nmr_7OVC.select('resnum 18 and name OG1 and chain A')
distances_2 = []
for glu, phe in zip(aa1_nmr.iterCoordsets(), aa2_nmr.iterCoordsets()):
distances_2.append(prody.calcDistance(glu,phe))
distances.loc[2, "Минимальное расстояние в ЯМР"] = min(distances_2)[0]
distances.loc[2, "Максимальное расстояние в ЯМР"] = max(distances_2)[0]
distances.loc[2, "Медианное расстояние в ЯМР"] = np.median(np.array(distances_2))
distances.loc[2, "% моделей ЯМР"] = len([i for i in distances_2 if i <= 3.5]) / 20 * 100
<ipython-input-4-c7c1e9ff7872>:9: FutureWarning: Setting an item of incompatible dtype is deprecated and will raise an error in a future version of pandas. Value 'GLU57' has dtype incompatible with float64, please explicitly cast to a compatible dtype first. distances.loc[0, "Донор"] = "GLU57" <ipython-input-4-c7c1e9ff7872>:10: FutureWarning: Setting an item of incompatible dtype is deprecated and will raise an error in a future version of pandas. Value 'PHE66' has dtype incompatible with float64, please explicitly cast to a compatible dtype first. distances.loc[0, "Акцептор"] = "PHE66"
distances
| Донор | Акцептор | Расстояние в РСА | % моделей ЯМР | Минимальное расстояние в ЯМР | Максимальное расстояние в ЯМР | Медианное расстояние в ЯМР | |
|---|---|---|---|---|---|---|---|
| 0 | GLU57 | PHE66 | 2.742513 | 100.0 | 2.780212 | 2.880255 | 2.858777 |
| 1 | LYS68 | ASP81 | 2.526808 | 20.0 | 2.715152 | 7.381178 | 5.483934 |
| 2 | GLN30 | ASP81 | 2.646262 | 85.0 | 2.842783 | 3.918057 | 2.969876 |
Можно заметить, что водородная связь между атомами остова, поддерживающая &beta-лист является наиболее стабильной: она представлена во всех моделях ЯМР и имеет малый разброс длины. В то время как связи между атомами боковых цепей уже менее стабильны. Удивительным образом связь между остатками ядра представлена в меньшем количестве структур ЯМР, чем связь между остатками на поверхности глобулы в неструктурированной части белка. По всей видимости это можно объяснить тем, что остаток лизина является длинным и его подвижность высока в любой части белка.
Задание 2¶
В данной части практикума сравнивалось значение B-факторов из модели РСА и RMSF модели ЯМР для проверки гипотезы о том, что ансамбль моделей в записи PDB, полученной методом ЯМР, можно считать отражением подвижности белка.
RMSFs = np.array([np.mean(prody.calcRMSF(res)) for res in nmr_7OVC["A"].iterResidues()][2:165])
xray_betas = []
for res in xray_7NW1.iterResidues():
mean_beta = np.mean(res.getBetas())
xray_betas.append(mean_beta)
xray_betas = np.array(xray_betas[0:163])
sns.regplot(x=RMSFs,
y = xray_betas,
color = "#7B018C")
plt.ylabel('В-фактор РСА')
plt.xlabel('RMSF')
plt.show()
reg = sm.OLS(xray_betas, RMSFs).fit()
reg.summary()
| Dep. Variable: | y | R-squared (uncentered): | 0.652 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared (uncentered): | 0.650 |
| Method: | Least Squares | F-statistic: | 303.9 |
| Date: | Wed, 18 Dec 2024 | Prob (F-statistic): | 5.30e-39 |
| Time: | 20:41:40 | Log-Likelihood: | -767.59 |
| No. Observations: | 163 | AIC: | 1537. |
| Df Residuals: | 162 | BIC: | 1540. |
| Df Model: | 1 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| x1 | 44.0863 | 2.529 | 17.434 | 0.000 | 39.093 | 49.080 |
| Omnibus: | 73.585 | Durbin-Watson: | 0.476 |
|---|---|---|---|
| Prob(Omnibus): | 0.000 | Jarque-Bera (JB): | 319.231 |
| Skew: | 1.667 | Prob(JB): | 4.78e-70 |
| Kurtosis: | 8.991 | Cond. No. | 1.00 |
Notes:
[1] R² is computed without centering (uncentered) since the model does not contain a constant.
[2] Standard Errors assume that the covariance matrix of the errors is correctly specified.
Формально между двумя параметрами имеется средняя положительная корреляция, однако остатки с большим значениями обеих метрик могут считаться выбросами в контексте регрессионного анализа, поэтому было решено отобрать только те остатки, для которых обе метрики не превышают 0,75-квантиль.
RMSFs_ = np.array([RMSF < np.quantile(RMSFs, 0.75) for RMSF in RMSFs])
xray_betas_ = np.array([xray_beta < np.quantile(xray_betas, 0.75) for xray_beta in xray_betas])
mask = RMSFs_ & xray_betas_
sns.regplot(x=RMSFs[mask],
y = xray_betas[mask],
color = "#7B018C")
plt.ylabel('В-фактор РСА')
plt.xlabel('RMSF')
plt.show()
reg = sm.OLS(xray_betas[mask], RMSFs[mask]).fit()
reg.summary()
| Dep. Variable: | y | R-squared (uncentered): | 0.918 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared (uncentered): | 0.917 |
| Method: | Least Squares | F-statistic: | 1093. |
| Date: | Wed, 18 Dec 2024 | Prob (F-statistic): | 5.86e-55 |
| Time: | 20:41:44 | Log-Likelihood: | -343.62 |
| No. Observations: | 99 | AIC: | 689.2 |
| Df Residuals: | 98 | BIC: | 691.8 |
| Df Model: | 1 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| x1 | 49.6999 | 1.503 | 33.065 | 0.000 | 46.717 | 52.683 |
| Omnibus: | 8.371 | Durbin-Watson: | 1.667 |
|---|---|---|---|
| Prob(Omnibus): | 0.015 | Jarque-Bera (JB): | 3.462 |
| Skew: | 0.136 | Prob(JB): | 0.177 |
| Kurtosis: | 2.125 | Cond. No. | 1.00 |
Notes:
[1] R² is computed without centering (uncentered) since the model does not contain a constant.
[2] Standard Errors assume that the covariance matrix of the errors is correctly specified.
В отфильтрованных данных всё ещё наблюдается корреляция при чём очень сильная. Таким образом ансамбль моделей в записи PDB, полученной методом ЯМР, можно считать отражением подвижности белка.
Задание 3¶
В данной части практикума сравнивалось значение B-факторов из модели РСА и RMSF модели ЯМР уже для всех атомов, а не для остатков.
xray_atoms_betas = []
for atom in xray_7NW1.iterAtoms():
if atom.getChid() == "AAA" and atom.getResnum() > 2 and atom.getResnum() < 165:
xray_atoms_betas.append(atom.getBeta())
xray_atoms_betas = np.array(xray_atoms_betas)
def calc_RMSF(coords):
return np.mean(np.linalg.norm((coords - np.mean(coords, axis=0)), axis=1) ** 2) ** 1/2
nmr_atoms_RMSFs = []
for atom in nmr_7OVC["A"].iterAtoms():
if atom.getElement() != "H" and atom.getResnum() > 2 and atom.getResnum() < 165:
nmr_atoms_RMSFs.append(calc_RMSF(atom.getCoordsets()))
nmr_atoms_RMSFs = np.array(nmr_atoms_RMSFs)
x_ = nmr_atoms_RMSFs ** 2
y_ = 3 * xray_atoms_betas / (8 * np.pi * np.pi)
sns.regplot(x = x_,
y = y_,
color = "#7B018C")
plt.ylabel('3 * В-фактор РСА / (8 * pi^2)')
plt.xlabel('RMSF^2')
plt.show()
reg = sm.OLS(y_, x_).fit()
reg.summary()
| Dep. Variable: | y | R-squared (uncentered): | 0.056 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared (uncentered): | 0.055 |
| Method: | Least Squares | F-statistic: | 79.54 |
| Date: | Wed, 18 Dec 2024 | Prob (F-statistic): | 1.52e-18 |
| Time: | 20:47:11 | Log-Likelihood: | -2600.6 |
| No. Observations: | 1337 | AIC: | 5203. |
| Df Residuals: | 1336 | BIC: | 5208. |
| Df Model: | 1 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| x1 | 0.0874 | 0.010 | 8.919 | 0.000 | 0.068 | 0.107 |
| Omnibus: | 600.254 | Durbin-Watson: | 0.067 |
|---|---|---|---|
| Prob(Omnibus): | 0.000 | Jarque-Bera (JB): | 4249.114 |
| Skew: | 1.948 | Prob(JB): | 0.00 |
| Kurtosis: | 10.817 | Cond. No. | 1.00 |
Notes:
[1] R² is computed without centering (uncentered) since the model does not contain a constant.
[2] Standard Errors assume that the covariance matrix of the errors is correctly specified.
Между двумя парметрами отсутствует ожидаемая зависимомть. Однако в данных явно есть выбросы, поэтому было опять отобрём только те атомы, для которых обе метрики не превышают 0,75-квантиль.
RMSFs_ = np.array([x < np.quantile(x_, 0.75) for x in x_])
xray_betas_ = np.array([y < np.quantile(y_, 0.75) for y in y_])
mask = RMSFs_ & xray_betas_
sns.regplot(x = x_[mask],
y = y_[mask],
color = "#7B018C")
plt.ylabel('3 * В-фактор РСА / (8 * pi^2)')
plt.xlabel('RMSF^2')
plt.show()
reg = sm.OLS(y_[mask], x_[mask]).fit()
reg.summary()
| Dep. Variable: | y | R-squared (uncentered): | 0.501 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared (uncentered): | 0.500 |
| Method: | Least Squares | F-statistic: | 841.9 |
| Date: | Wed, 18 Dec 2024 | Prob (F-statistic): | 8.99e-129 |
| Time: | 20:47:16 | Log-Likelihood: | -900.18 |
| No. Observations: | 841 | AIC: | 1802. |
| Df Residuals: | 840 | BIC: | 1807. |
| Df Model: | 1 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| x1 | 124.0151 | 4.274 | 29.015 | 0.000 | 115.626 | 132.404 |
| Omnibus: | 323.647 | Durbin-Watson: | 0.416 |
|---|---|---|---|
| Prob(Omnibus): | 0.000 | Jarque-Bera (JB): | 1160.127 |
| Skew: | -1.852 | Prob(JB): | 1.21e-252 |
| Kurtosis: | 7.403 | Cond. No. | 1.00 |
Notes:
[1] R² is computed without centering (uncentered) since the model does not contain a constant.
[2] Standard Errors assume that the covariance matrix of the errors is correctly specified.
В отфильтрованных данных можно наблюдать умеренную корреляцию, однако я бы не рискнул утверждать, что ансамбль моделей в записи PDB, полученной методом ЯМР, действительно можно принять за отражение подвижности белка, взяв для сравнения значения B-факторов из модели РСА.