Поиск в нуклеотидных банках по аннотации. Выравнивание геномов
Выбор геномов и получение DotPlot
Я решила выбрать геном патогенной бактерии Corynebacterium jeikeium. В NCBI ввела запрос (Corynebacterium jeikeium[Organism]) AND chromosome[Title]. Выбрала штамм DSM 7171 (полный геном – DSM 7171). Сначала попробовала сравнить геномы разных штаммов (их всего нашлось 2), но ничего интересного не получилось, поэтому решила выбрать бактрию того же рода, но другого вида – Corynebacterium glaucum DSM 44530 (полный геном – NZ_CP047358.1).
В BLAST ввела AC геномов и получила DotPlot. Сначала попробовала blastn:
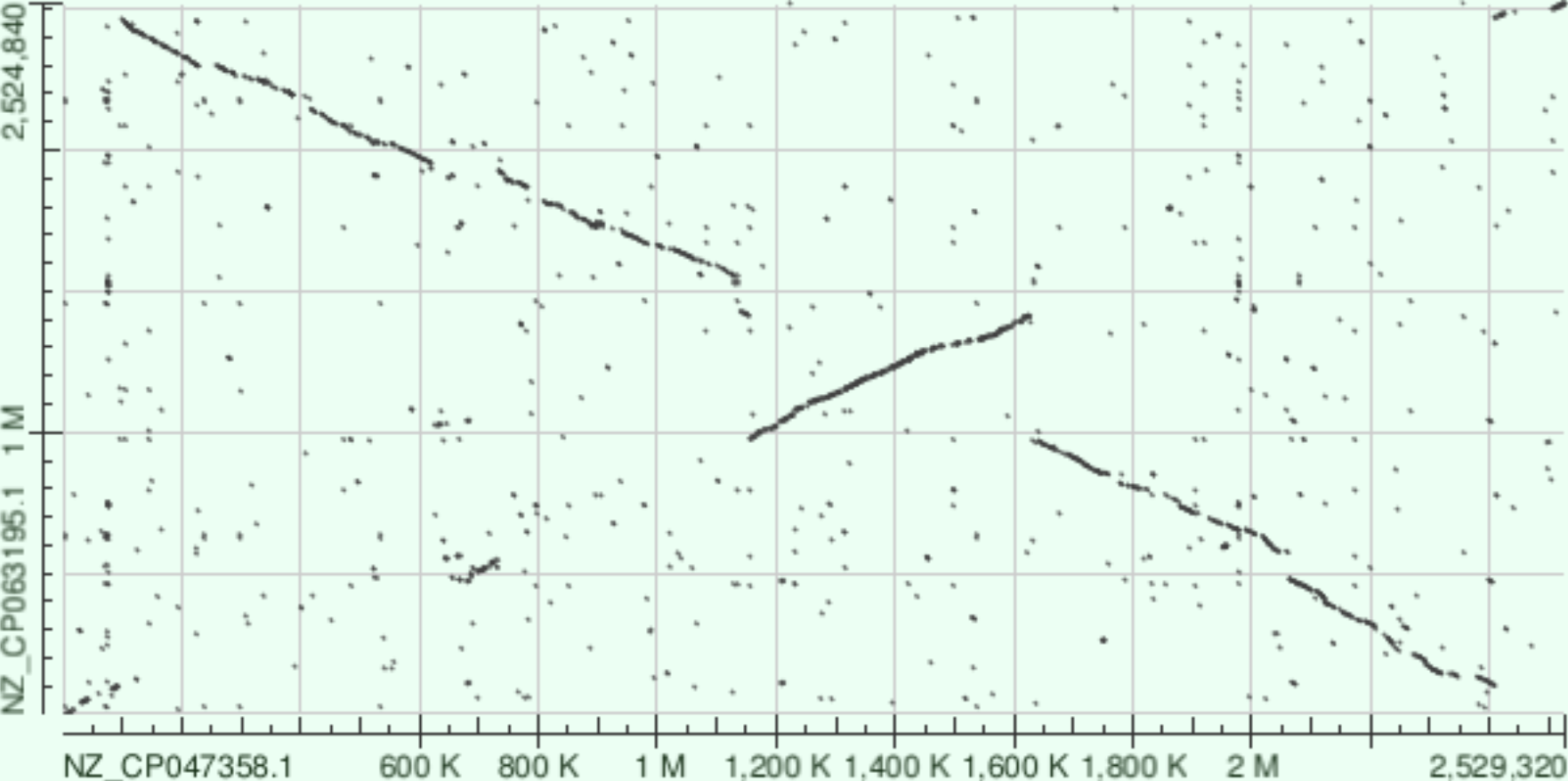
Значение word size я взяла 11, так как при значении 7 выбавалась ошибка.
Поиск megablast я сначала осуществила с word size 23, очертания DotPlot были такие же, как при blastn, но некоторые участки отсутствовали. Затем взяла word size 16 для более чёткого DotPlot, он почти полностью совпал с DotPlot, полученыым с помощью blastn.
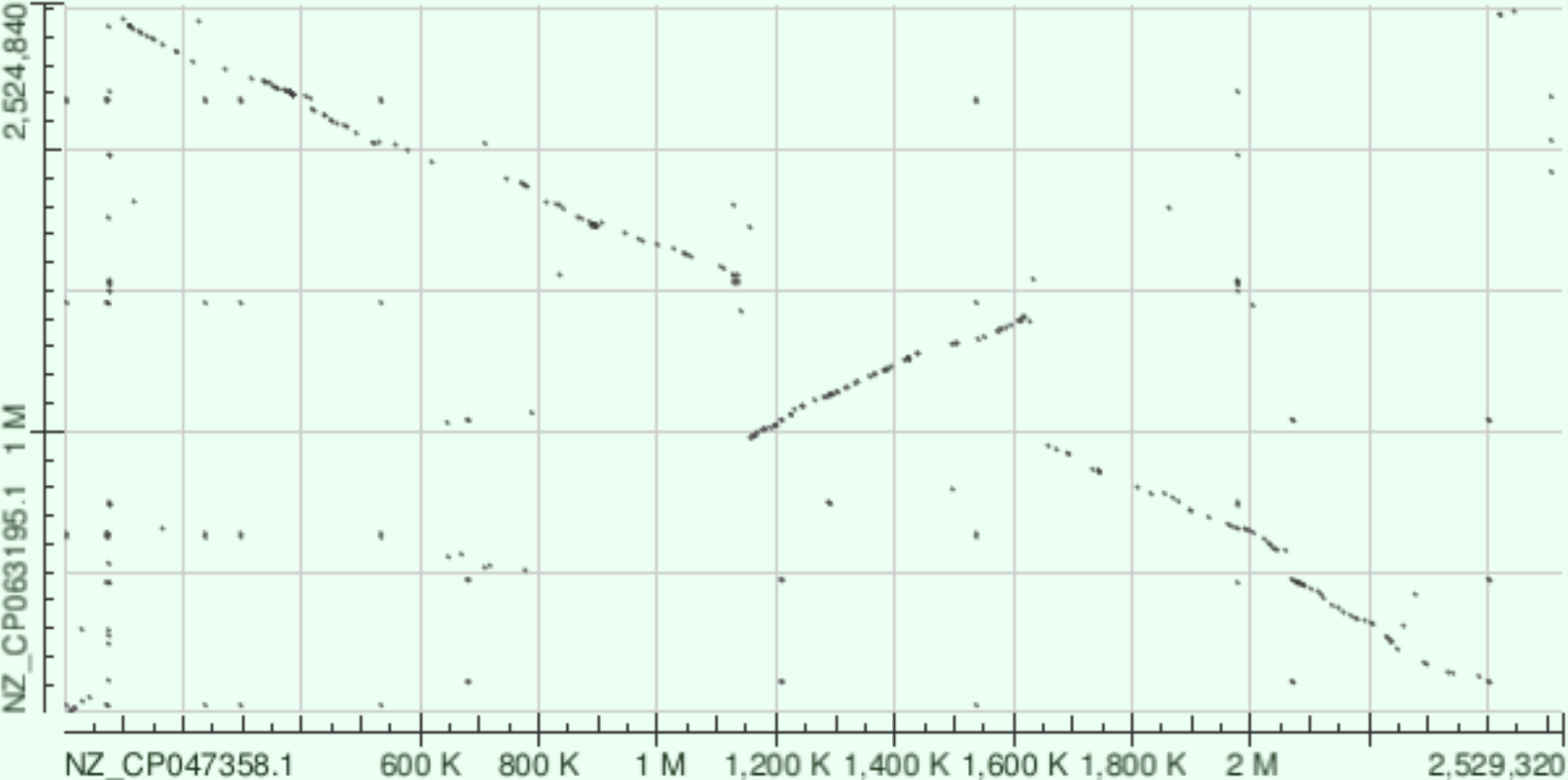
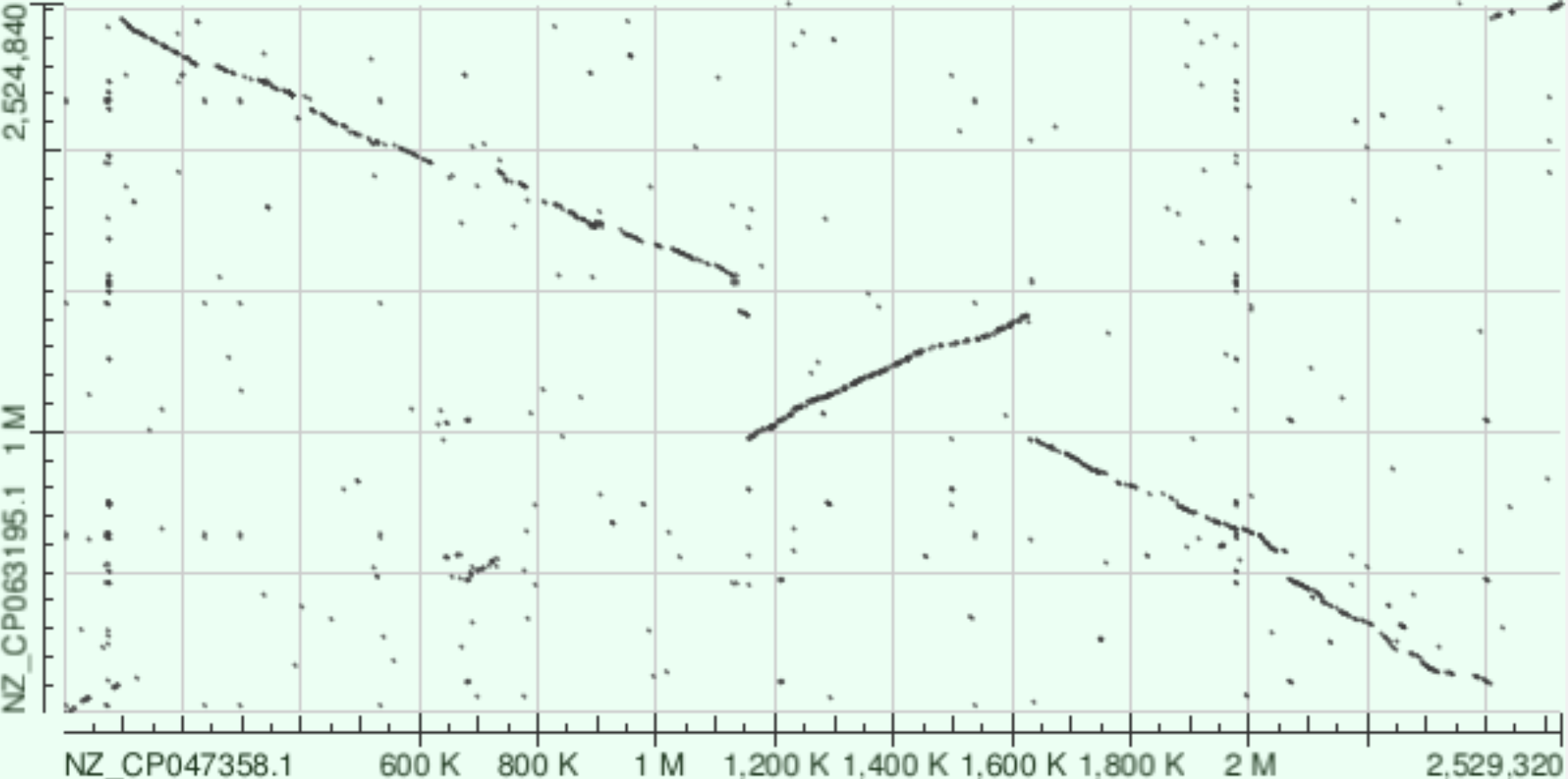
Анализ DotPlot
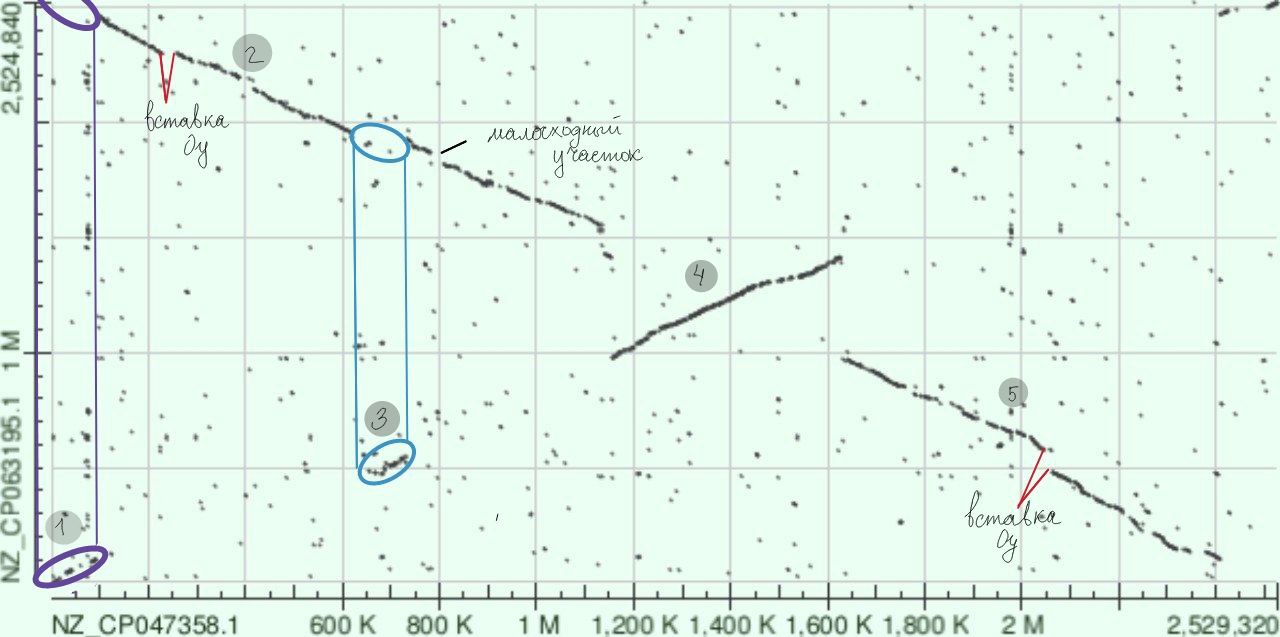
Первое, что стоит отметить – это то, что основная линия идёт по побочной диагонали, а это значит, что выбраны разные цепи.
По оси Oy расположен геном Corynebacterium jeikeium DSM 7171, а по оси Ox – Corynebacterium glaucum DSM 44530
Сразу видно большой гомологичный участок длиной примерно 1М нуклеотидов (Рис. 4 – 2), затем участок с инверсией с координатами 1150К-1600К (Рис. 4 – 4) и продолжение гомологичного участка длиной около 800К нуклеотидов.
Также в самом начале и дальше в середине 2 участка видны небольшие участки с релокацей и инверсией (Рис. 4 – 1 и 3).
Кроме этого, видны два участка (250К-300К и в районе 2М), где произошли небольшие вставки у Corynebacterium jeikeium DSM 7171.
Местами также заметны малосходные участки (например, в районе 800К)