Трансмембранные белки
Сравнение предсказаний трансмембранных участков в бета-листовом белке
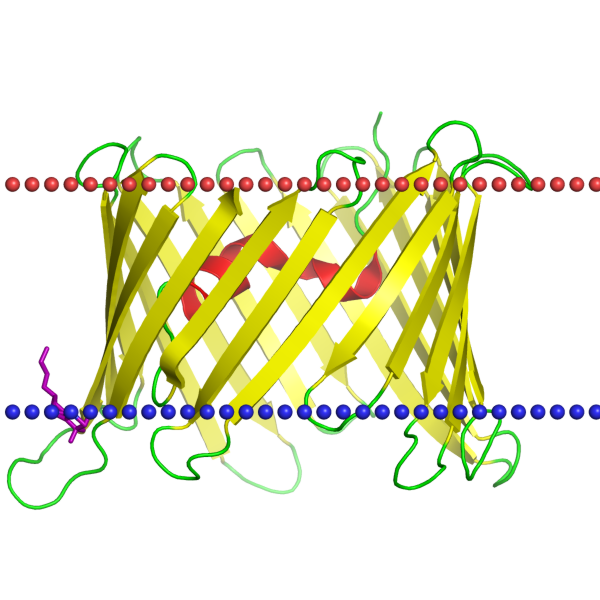
С помощью поиска по уровням классификации в базе данных OPM нашла белок, в трансмембранной части которого находятся β-листы.
Характеристики белка:
type: transmembrane (трансмембранные белки)
class: beta-barrel transmembrane (белки, в трансмембранной части которых находятся β-листы)
superfamily: mitochondrial and plastid porins (порины митохондрий и пластид)
family: voltage-dependent anion channel (VDAC) porin (потенциал-зависимые анионные порины)
name: VDAC-2 channel (потенциал-зависимый анионный канал 2)
pdb: 4bum
uniprot: Q8AWD0_DANRE
function: Белок формирует порообразующие потенциал-зависимые анионные каналы, которые считаются основным путем
диффузии метаболитов через внешнюю мембрану митохондрий. Считается также, что этот белок участвует в митохондриальном пути апоптоза
Координаты трансмембранных участков белка, приведенные в OPM
14-21, 28-33, 40-46, 55-62, 70-74, 81-87, 95-102, 111-119, 123-130, 137-145, 149-156, 166-173, 180-184, 191-197,
204-209, 218-226, 243-249, 257-263,
Запуск DeepTMHMM для последовательности белка
Из записи pdb (4bum) скачала fasta-файл с последовательностью и загрузила его в DeepTMHMM.
Предсказанные программой координаты трансмембранных участков:
41-48, 55-61, 69-75, 81-87, 96-103, 112-118, 125-131, 136-144,
149-157, 166-173, 180-184, 190-195, 204-209, 218-224, 232-236,
243-248, 257-262, 274-278
Графическое изображение результатов DeepTMHMM
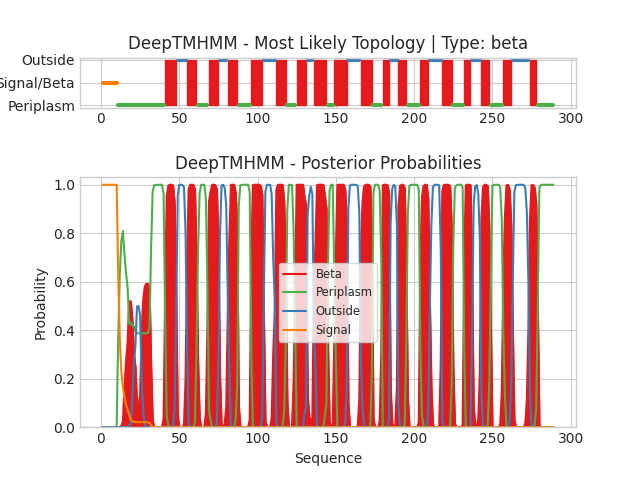
На данном изображении по горизонтали отложены позиции в последовательности белка. Сверху схематически показано положение всего белка относительно мембраны. Видно, что внутренние петли по размеру сходны со внешними петлями. Снизу по вертикали – вероятность пространственного положения данного участка белка. Красным цветом выделены трансмембранные участки, зелёным – участки с внутренней стороны (в периплазматическом пространстве), а синим – с внешней стороны.
Ссылка на файл с результатами в текстовом виде в формате .3line
Сравнение результатов, полученных двумя методами
| OPM | DeepTMHMM |
|---|---|
| 14-21 | – |
| 28-33 | – |
| 40-46 | 41-48 |
| 55-62 | 55-61 |
| 70-74 | 69-75 |
| 81-87 | 81-87 |
| 91-102 | 96-103 |
| 111-119 | 112-118 |
| 123-130 | 125-131 |
| 137-145 | 136-144 |
| 149-156 | 149-157 |
| 166-173 | 166-173 |
| 180-184 | 180-184 |
| 191-197 | 190-195 |
| 204-209 | 204-209 |
| 218-226 | 218-224 |
| – | 232-236 |
| 243-249 | 223-248 |
| 257-263 | 257-262 |
| 274-281 | 274-278 |
Из таблицы видно, что не результаты работы двух алгоритмов не совпадают. DeepTMHMM нашёл 18 трансмембранных участков, а OPM – 19. При этом DeepTMHMM пропустил два трансмембранных участка найденных OPM, но нашёл один участок, которого нет в OPM. Таким образом, предсказания перекрываются для 17 β-тяжей. Но полностью координаты совпадают только у 5, у остальных отличаются на пару единиц, причём в основном отличаются координаты именно конца участка, и β-тяжи с такими отличиями идут друг за другом, что наводит на мысль о том, что это просто результат погрешности работы программ. Так же стоит отметить, что на графическом изображении вывода DeepTMHMM на первых ~50 позициях белка вероятности положения довольно низкие, так что в этой области скорее стоит доверять предсказаниям OPM.
Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Описание белка
Для этого задания мне был выдан белок со следующими характеристиками:
Swiss-Prot AC: A4YWY9 (KUP3_BRASO)
name: Probable potassium transport system protein Kup 3 (вероятный белок системы транспорта калия Kup 3)
organism: Bradyrhizobium sp. (грамотрицательные)
functiom: Транспорт калия в клетку. Вероятно, осуществляет симпорт K+ и H+
Запуск DeepTMHMM
Из записи UniProt скачала последовательность в fasta формате и загрузила в DeepTMHMM.
Предсказанные программой координаты трансмембранных участков:
24-39, 61-77, 114-136, 153-168, 178-198, 223-243, 257-274, 294-319,
352-367, 376-396, 405-427, 433-448
Графическое изображение результатов DeepTMHMM
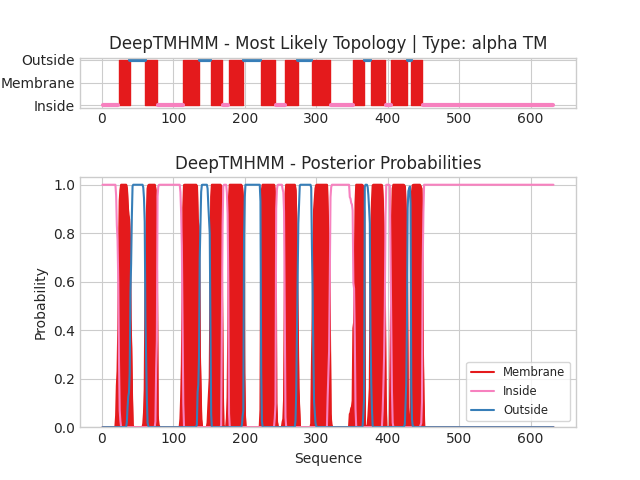
Изображение пояснено в первом задании. Здесь внешние и внутренние петли различны по размеру
Ссылка на файл с результатами в текстовом виде в формате .3line
Предсказание AlphaFold
В записи UniProt скачала предсказание AlphaFold трехмерной структуры выданного белка
Запуск PPM
Полученный файл загрузила в PPM 3.0 со следующими параметрами:
number of membranes: 1
type of membrane: Gram-negative bacteria inner membrane (из аннотации в UniProt)
allow curvature: no (по умолчанию)
topology (N-ter): in (по предсказанию DeepTMHMM N-конец внутри – inside 1 23)
Координаты предсказанных участков:
23-45, 61-84, 113-146, 149-168, 178-199, 224-244, 253-274, 293-319, 352-372, 373-394, 404-431, 432-449
Сравнение результатов, полученных двумя методами
| DeepTMHMM | PPM 3.0 |
|---|---|
| 24-39 | 23-45 |
| 61-77 | 61-84 |
| 114-136 | 113-146 |
| 153-168 | 149-168 |
| 178-198 | 178-199 |
| 223-243 | 224-244 |
| 257-274 | 253-274 |
| 294-319 | 293-219 |
| 352-367 | 352-372 |
| 376-396 | 373-394 |
| 405-427 | 404-431 |
| 433-448 | 432-449 |
Оба алгоритма нашли одинаковое количество трансмембранных участков – 12, но границы участков различаются сильнее, чем
для белка с