Поиск de novo сигналов в ДНК
Выбор бактерии и подготовка данных
Я решила выбрать бактерию Mycobacterium tuberculosis. Она вызывает туберкулёз у человека в 92% случаев.
- Taxon: Mycobacterium tuberculosis H37Rv
- NCBI RefSeq assembly: GCF_000195955.2
- Submitted GenBank assembly: GCA_000195955.2
Скачала два файла: FASTA (файл с последовательностью хромосомы) и GFF (аннотация). Загрузила их в Operon Mapper, чтобы получить список оперонов. По ключевым словам были отобраны гены домашнего хозяйства. В качестве отрицательного контроля были использованы случайные последовательности длиной 100 нуклеотидов (той же длины, что и промоторы).
Скрипт для получения выборок взяла у нашего старшего товарища Георгия Муравьёва
Получила 3 файла:
- housekeeping.fasta – материал обучения
- promotors.fasta – выборка для тестирования
- negative.fasta – негативный контроль (случайные последовательности длиной 100 нулеотидов)
Запуск MEME
Попробовала воспользоваться локальной версией на kodomo и сервисом MEME-suit.
Поиск мотивов на одной цепи:
meme housekeeping.fasta -dna -nmotifs 3 -minw 6 -maxw 50
Описание параметров:
- housekeeping.fasta – фаста файл со входными последовательностями (материал обучения)
- -dna – алфавит ДНК
- -nmotifs 3 – 3 мотива в выдаче
- -minw 6 – минимальная длина мотива
- -maxw 50 – максимальная длина мотива (default)
Выдача meme: текстовый формат, html-формат
MEME нашёл три мотива, LOGO и характеристики для которых представлены ниже:
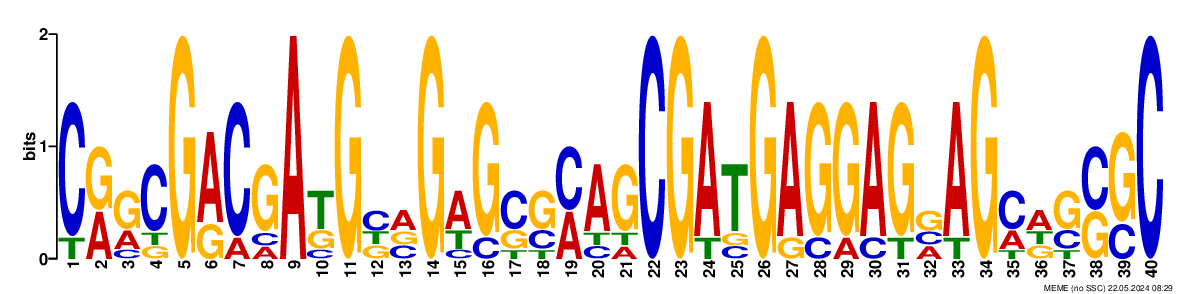


Меня, конечно, немного смущает выское GC содержание в первом мотиве, но у него наименьший и в целом адекватный e-value, поэтому сосредоточусь на нём.
Поиск сигнала в материале для тестирования с помощью FIMO
Я проверила первый мотив CRRCGACGAKGBVGWGSSMAGCGATGAGGAGVAGMDSSGC на материвле тестирования и выборке отрицательного контроля, запустив консольную версию FIMO на kodomo:
fimo -motif CRRCGACGAKGBVGWGSSMAGCGATGAGGAGVAGMDSSGC --norc --thresh 0.001 meme_out/meme.txt promotors.fasta
fimo -motif CRRCGACGAKGBVGWGSSMAGCGATGAGGAGVAGMDSSGC --norc --thresh 0.001 --o fimo2_out meme_out/meme.txt negative.fasta
Среди промоторов было найдено 2 315 последовательностей, подходящих под мотив с p-value меньше 0.001, а среди выборки отрицательного контроля – 263 последовательности.