BLAST
Для выполнения практикума была взята аминокислотная последовательность дУТФазы, выделенной из Amycolatopsis orientalis HCCB10007 (YP_008011580.1).
Задача: найти гомологов данного белка в базах данных последовательностей.
1. Поиск сходных последовательностей в базе данных Refseq_protein.
Для поиска гомологичных последовательностей на сайте NCBI был запущен BLASTP по базе данных refseq_protein. В дополнительных параметрах я установила максимальное количество находок 20000, чтобы увидеть все найденные гомологи. Все остальные параметры были оставлены по умолчанию. В результате было получено 8708 находок. Из них 7655 прокариотических белков, 813 эукариотических, 35 - архейных и 205 вирусных белка. Параметры для лучшей и худшей находок и для находки из середины списка указаны в таблице 1.
| Название белка | Организм | Длина выравнивания | bit score | % идентичных колонок | % сходных колонок | E-value | Выравнивание |
| deoxyuridine 5'-triphosphate nucleotidohydrolase | Amycolatopsis alba | 159 | 304 | 99 | 99 | 1e-102 | Выравнивание |
| deoxyuridine 5'-triphosphate nucleotidohydrolase | Bartonella bacilliformis | 149 | 103 | 43 | 56 | 4e-24 | Выравнивание |
| deoxycytidine triphosphate deaminase | Corynebacterium sputi | 62 | 36.2 | 39 | 53 | 9.5 | Выравнивание |
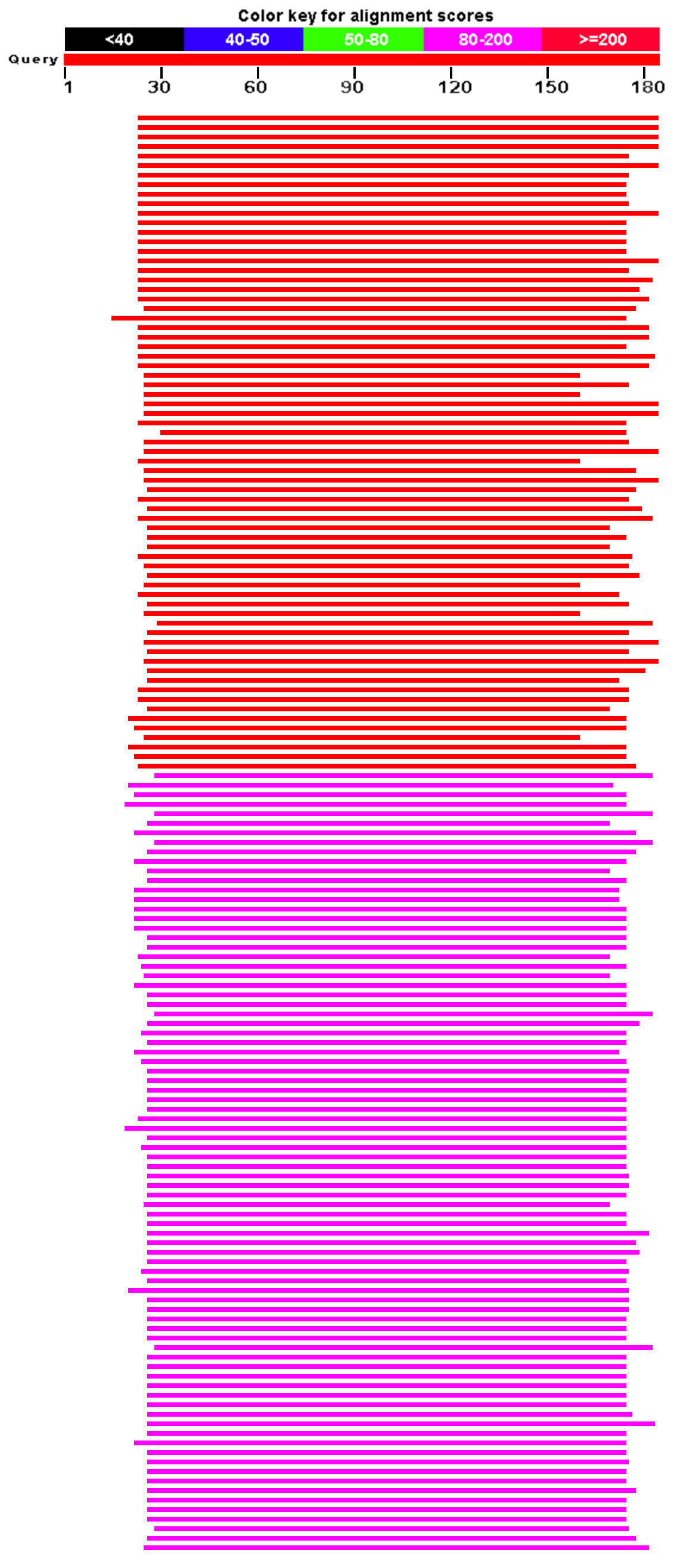
Рисунок 1. Графическое представление результатов поиска, содержащее 1000 находок, которые достоверно можно считать гомологами выбранного белка (увеличенное изображение - по клику).
2. Поиск сходных последовательностей среди белков из определенной таксономической группы.
Для выполнения этого задания я искала гомологичные последовательности среди бактерий рода Amycolatopsis. Для белка dUTP pyrophosphatase, выделенного из Amycolatopsis mediterranei (WP_013224478.1) я сравнила параметры при поиске по всем организмам и при поиске по некрупной таксономической группе. Все параметры, кроме E-value совпали (это логично, так как они характеризуют выравнивание). А вот E-value показывает, насколько случайна полученная находка, поэтому при поиске по меньшей базе данных это значение должно быть меньше. Так и есть: при поиске по всем организмам E-value данной находки составило 1e-78, а при поиске только по роду Amycolatopsis 3e-81.
3. Карта локального сходства для одной из найденных последовательностей.
Для последовательности deoxyuridine 5'-triphosphate nucleotidohydrolase из организма Loktanella hongkongensis было выполнено выравнивание с исходной последовательностью и получена карта локального сходства для этого выравнивания (рисунок 2).
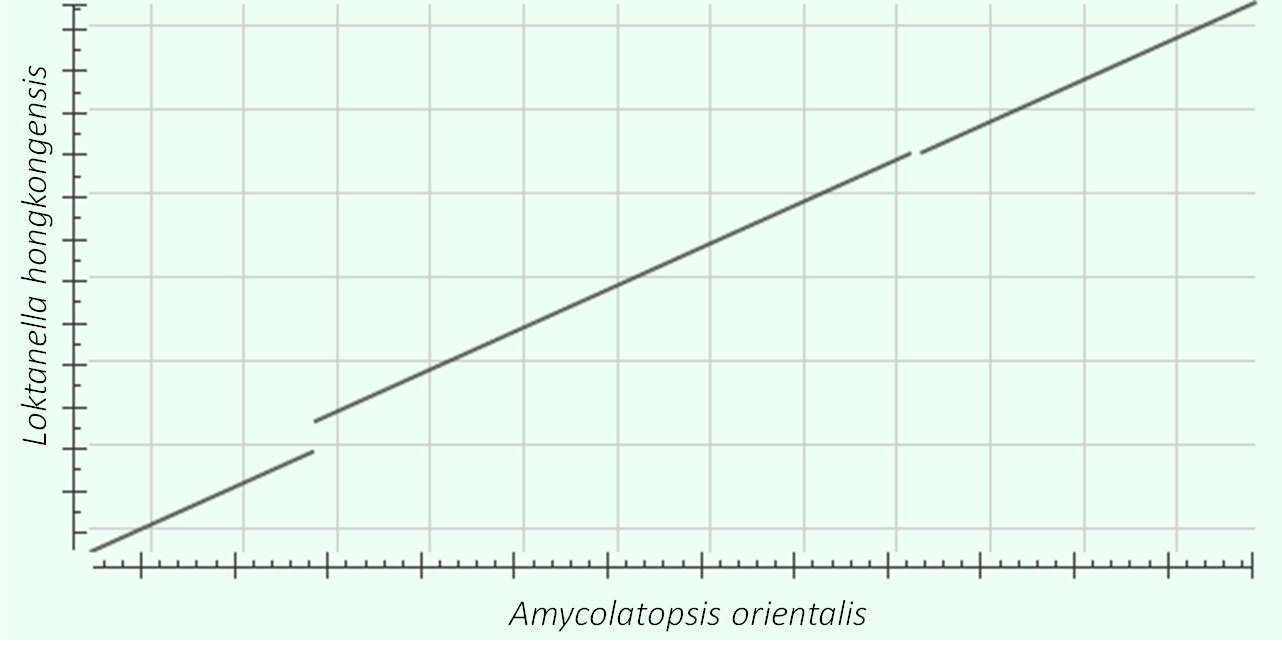
Рисунок 2. Карта локального сходства для двух выбранных последовательностей.
4. Использование BLAST для поиска в собственной базе данных.
Для выполнения этого задания была создана собственная база данных из последовательностей, входящих в выравнивание align_13.fasta. Все гэпы предварительно были удалены: database.fasta. Работа выполнялась на сервере kodomo:
makeblastdb -dbtype prot -in database.fasta -out mydatabase blastp -db mydatabase -query sequence.fastaПрограмма выдала только одну находку. Полученное выравнивание представлено на рисунке 3, а параметры для данной находки представлены в таблице 2.
Параметры выравнивания:
Матрица: BLOSUM62
Штраф за открытие гэпа: 11
Штраф за продолжение гэпа: 1
Neighboring words threshold: 11

Рисунок 3. Выравнивание последовательности выбранного белка с белком ALIAD из новой базы данных.
| Находка | Длина выравнивания | bit score | % идентичных колонок | % сходных колонок | E-value |
| ALIAD | 84 | 16.2 | 26 | 38 | 3.6 |
© Наталья Ланина
e-mail: n.lanina@fbb.msu.ru
последний раз обновлялось: 19.2.16