Комплексы ДНК-белок
Задание 1. Поиск ДНК-белковых контактов в заданной структуре
Упр. 1.
Для программы JMol был создан скрипт. Данный скрипт содержит определения следующих множеств:
- множество атомов кислорода 2'-дезоксирибозы (set1);
- множество атомов кислорода в остатке фосфорной кислоты (set2);
- множество атомов азота в азотистых основаниях (set3).
Вызов данного скрипта в JMol дает последовательное изображение всей структуры, только ДНК в проволочной модели, той же модели, но с выделенными шариками множеством атомов set1, затем set2 и set3.
Упр. 2.
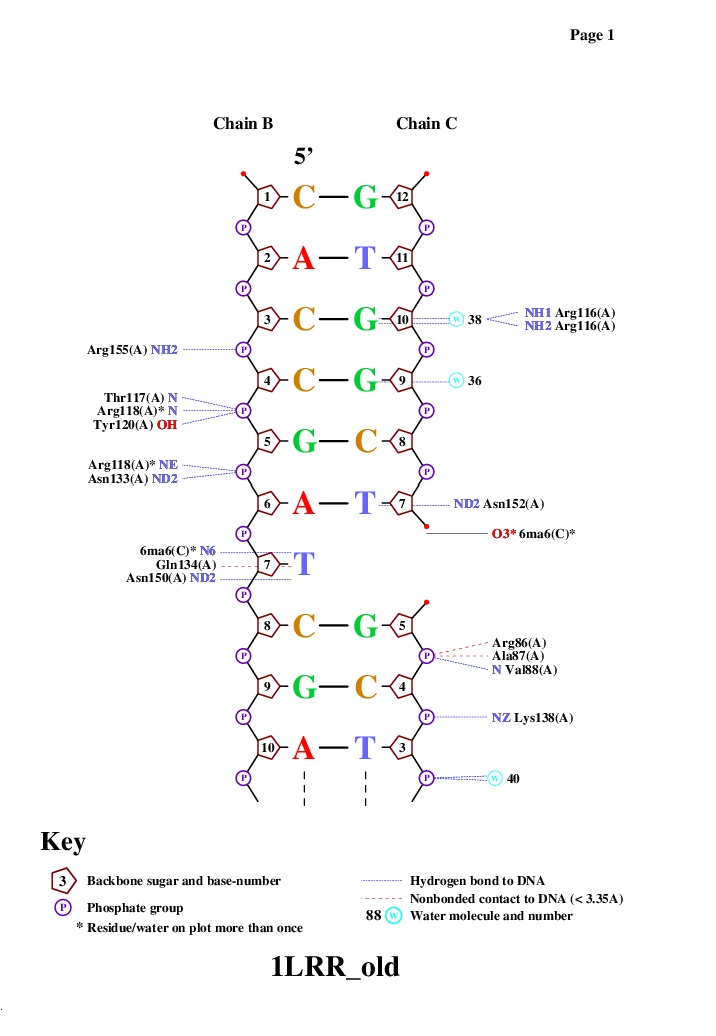
Опишем ДНК-белковые контакты в структуре 1LRR.
Будем считать полярными атомы кислорода и азота, а неполярными атомы углерода, фосфора и серы.
Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5Å. Аналогично, неполярным контактом будем считать парунеполярных атомов на расстоянии меньше 4.5Å.
Для подсчета контактов разного типа был создан скрипт. Полученные результаты представлены в таблице:
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 2 | 12 | 14 |
| остатками фосфорной кислоты | 26 | 30 | 56 |
| остатками азотистых оснований со стороны большой бороздки | 4 | 8 | 12 |
| остатками азотистых оснований со стороны малой бороздки | 1 | 0 | 1 |
Упр. 3.
С помощью программы nucplot была получена популярная схема ДНК-белковых контактов.
Упр. 4.
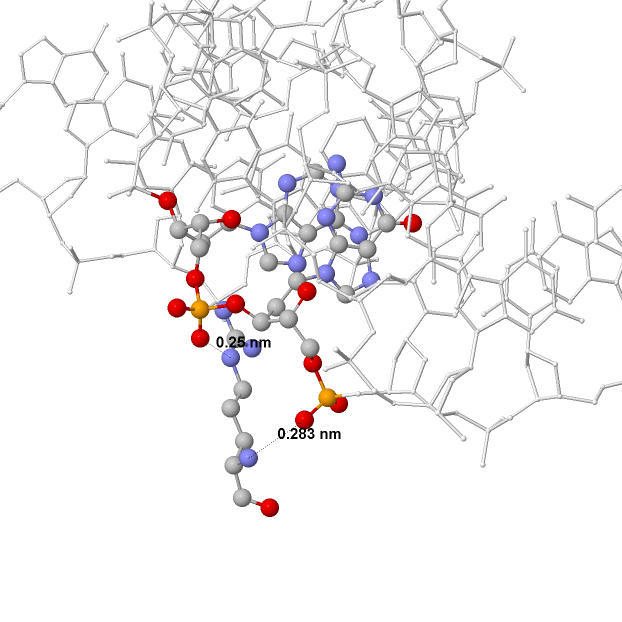
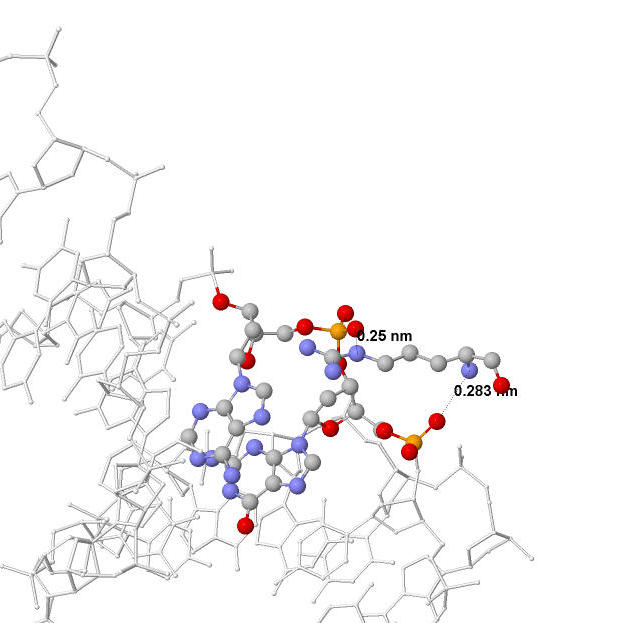
Из схемы видно, что наибольшее количество контактов у Arg118 - с 5 и 6 нуклеотидами. Вероятно, эта аминокислота является наиболее важной для распознавания последовательности ДНК. На рисунках, приведенных ниже, показано взаимодействие данной аминокислоты с ДНК. Обозначены расстояния между аминокислотным остатком и ДНК.
Задание 2. Предсказание вторичной структуры заданной тРНК
Упр. 1. Предсказание вторичной структуры тРНК путем поиска инвертированных повторов
Для тРНК 1QTQ с помощью программы einverted была получена информация об инвертированных участках тРНК.
При параметрах, установленных по умолчанию, инвертированные участки не были найдены. Поэтому использовались следующие параметры:
gap penalty 10 minimum score threshold 25 match score 5 mismatch score -2
Были найдены D-стебель и акцепторный стебель, 3 пары антикодонового стебля и пара T-стебля.
Упр. 2. Предсказание вторичной структуры тРНК по алгоритму Зукера
С помощью программы mfold было получено предсказание вторичной структуры тРНК по алгоритму Зукера. Ниже представлено полученное изображение вторичной структуры.
В таблице представлено сравненние полученных результатов:
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-902-907-3' 5'-966-970-3' (7 пар) | 7 из 7 | 7 из 7 |
| D-стебель | 5'-910-912-3' 5'-923-925-3' (3 пары) | 2 из 3 | 3 из 3 |
| T-стебель | 5'-949-953-3' 5'-926-933-3' (8 пар) | 1 из 8 | 6 из 8 |
| Антикодоновый стебель | 5'-937-944-3' 5'-961-965-3'(5 пар) | 3 из 5 | 3 из 5 |
| Общее число канонических пар нуклеотидов | 23 | 13 | 19 |
© Наталья Ланина
e-mail: n.lanina@fbb.msu.ru