Филогенетическое дерево. Реконструкция филогении. Укоренение и бутстрэп.
В отчете представлено выполнение некоторых заданий практикумов 1-3. Цель этих занятий состояла в том, чтобы научиться работать с филогенетическими деревьями, попробовать реконструировать филогенетическое дерево на основании белковых последовательностей и научиться переукоренять деревья.
Отобранные бактерии.
Из предложенного списка я выбрала следующие 8 видов бактерий:
таксономический сервис NCBI. Все бактерии относятся к грамположительным (Bacteria; Firmicutes), поэтому для упрощения в таблице указана систематика, начиная с класса.На основании данного нам правильного филогенетического дерева, было построено филогенетическое дерево для отобранных организмов.
Cкобочная формула дерева.
(CLOB1,((LACDA,(ENTFA, STRPN)),(((BACAN, GEOKA),LISMO),STAES)));Для получения изображения дерева данная скобочная формула была сохранена в отдельном файле .tre, а затем открыта в программе MEGA6.
Изображение дерева.
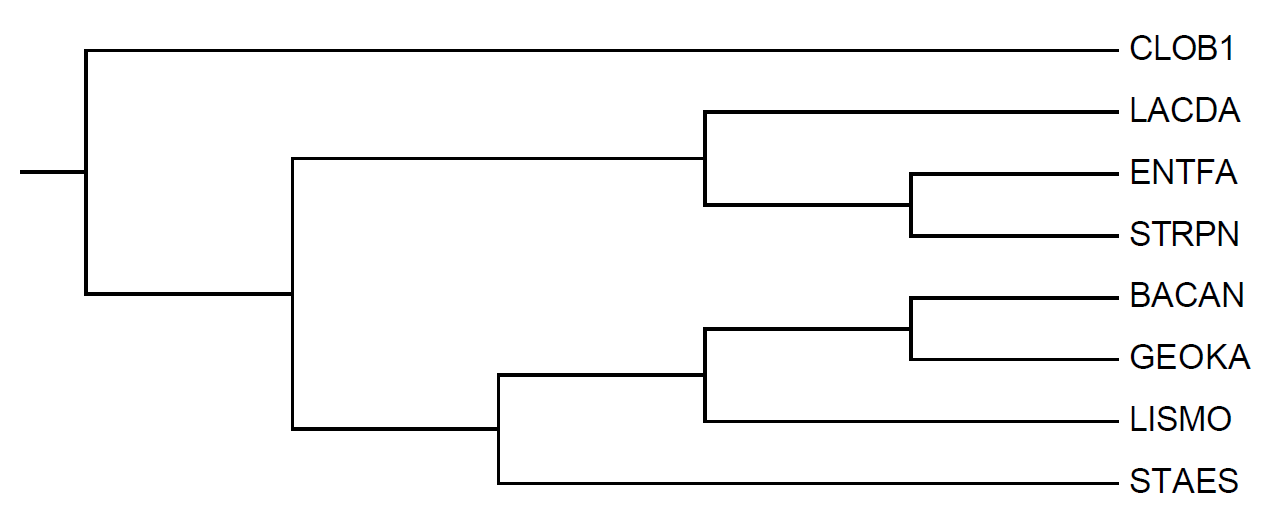
Рисунок 1. Филогенетическое дерево для восьми выбранных видов бактерий.
Ветви дерева.
Полученное дерево содержит 5 нетривиальных ветвей (ветвь нетривиальная, если она разбивает множество листьев на подмножества, в каждом из которых более одного элемента):
- {LACDA, ENTFA, STRPN} против {CLOB1, BACAN, GEOKA, LISMO, STAES}
- {ENTFA, STRPN} против {CLOB1, LACDA, BACAN, GEOKA, LISMO, STAES}
- {BACAN, GEOKA, LISMO, STAES} против {CLOB1, LACDA, ENTFA, STRPN}
- {BACAN, GEOKA, LISMO} против {CLOB1, LACDA, ENTFA, STRPN, STAES}
- {BACAN, GEOKA} против {CLOB1, LACDA, ENTFA, STRPN, LISMO, STAES}
- Первая ветвь из списка соответствует порядку Lactobacillales;
- Третья ветвь из списка соответствует порядку Bacillales;
- Пятая ветвь из списка соответствует семейству Bacillaceae;
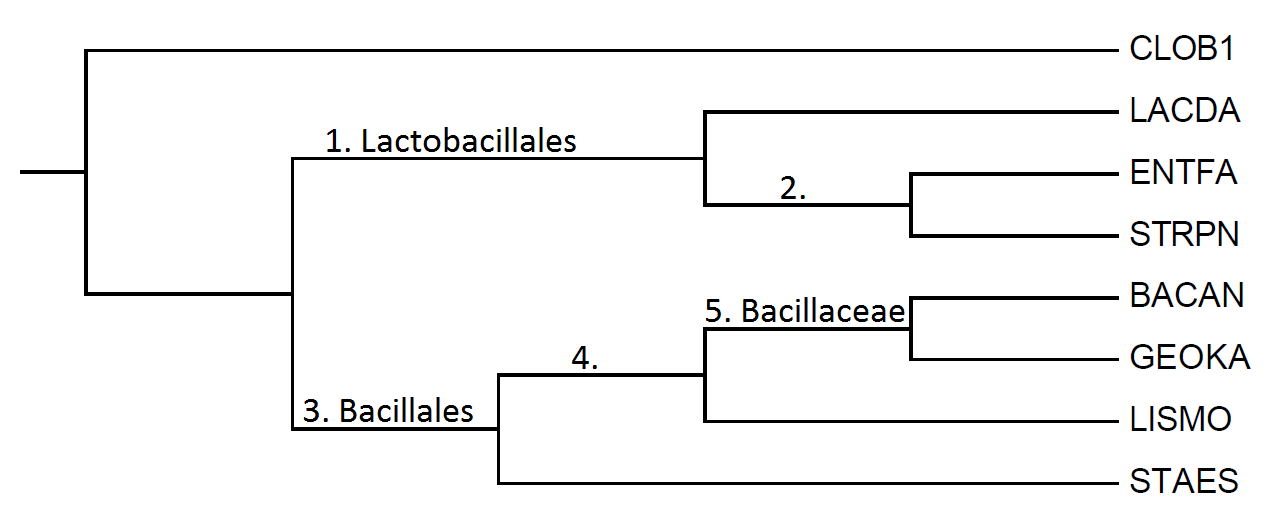
Рисунок 2. Нетривиальные ветви для полученного филогенетического дерева. Номера ветвей соответствуют номеру в списке. Таксоны, если они есть, указаны рядом с номером.
Реконструкция филогенетического дерева.
Для выбранных организмов на основании белковой последовательности фактора элонгации трансляции G было реконструировано филогенетическое дерево.Для этого с помощью программы JalView были получены необходимые последовательности из базы данных Uniprot для которых было построено выравнивание с помощью Muscle. Реконструкция деревом проводилась методом Neighbor Joining Using % Identity. Изображение полученного дерева представлено на рисунке 3. Можно заметить, что реконструированное дерево отличается от правильного, избраженного на рисунке 2. Так у полученного дерева отсутствуют нетривиальные ветви 1 и 3, но вместо них есть ветви {LACDA, CLOB1} против {STAES, STRPN, ENTFA, LISMO, GEOKA, BACAN} и {LACDA, STAES, CLOB1} против {STRPN, ENTFA, LISMO, GEOKA, BACAN}.
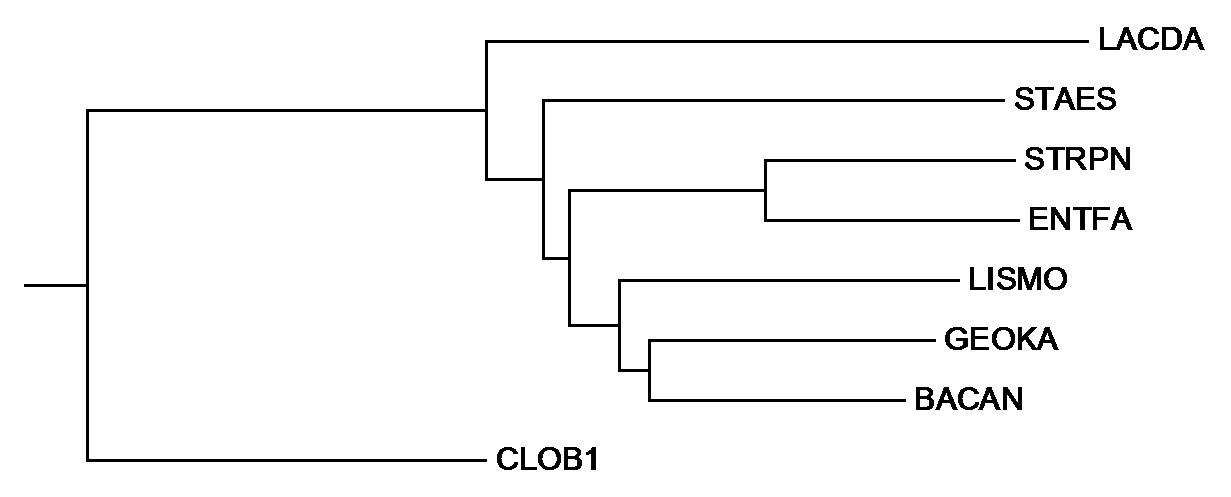
Рисунок 3. Филогенетическое дерево, полученное в результате реконструкции по белковой последовательности фактора элонгации трансляции G методом Neighbor Joining Using % Identity.
Укоренение в среднюю точку.
Для укоренения в среднюю точку была использованна программа retree пакета PHYLIP. Изображение полученного дерева представлено на рисунке 4, оно не отличается от изображения дерева до укоренения. Укоренение произошло в ту же ветвь, что и у правильного дерева - {CLOB1} против {LACDA, STAES, STRPN, ENTFA, LISMO, GEOKA, BACAN}.
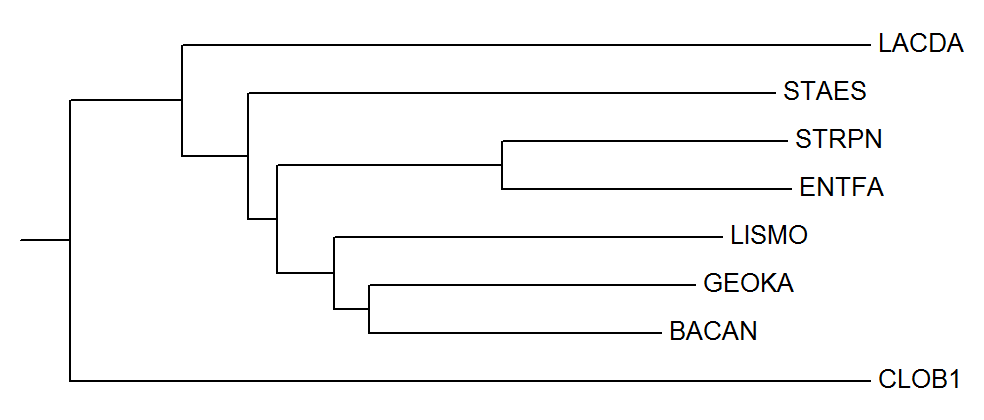
Рисунок 4. Филогенетическое дерево, укорененное в среднюю точку.
© Наталья Ланина
e-mail: n.lanina@fbb.msu.ru
последний раз обновлялось: 18.1.16