Демонстрация навыков.
Быстрый переход к:
d1. Определение вторичной структуры.
d2. Совмещение структур.
d3. Нахождение гидрофобных кластеров.
d4. Построение поверхности.
d5. Сравнение доменов SCOP и Pfam.
d1: Определение вторичной структуры.
Для выполнения задания была взята структура 4D7S. На рисунке 1.1 представлена разметка данной структуры, исходя из аннотации в .pdb-файле.
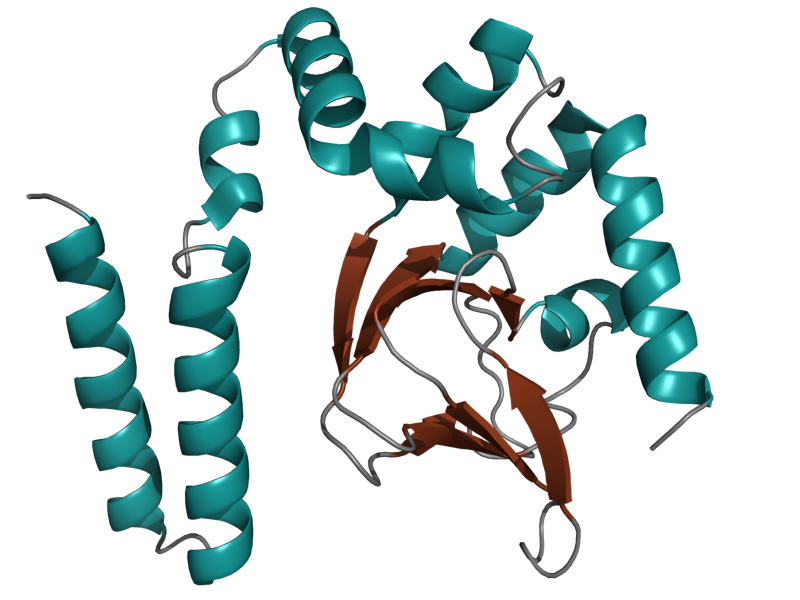
Рисунок 1.1. Разметка вторичной структуры 4D7S в .pdb-файле. Голубым цветом обозначены α-спирали, коричневым - β-тяжи.
| № | Аннотация PDB | Разметка DSSP |
| α-спираль | ||
| 1 | 228-246 | 229-245 |
| 2 | 249-267 | 250-266 |
| 3 | 271-277 | 272-276 |
| 4 | 280-292 | 281-291 |
| 5 | 292-299 | 293-298 |
| 6 | 306-314 | 307-313 |
| 7 | 315-317 | |
| 8 | 368-373 | 369-372 |
| 9 | 393-404 | 394-403 |
| 10 | 404-420 | 405-419 |
| β-тяж | ||
| 1 | 318-322 | 318-322 |
| 2 | 327-329 | 327-329 |
| 3 | 338-343 | 338-343 |
| 4 | 346-349 | 346-349 |
| 5 | 356-360 | 356-360 |
| 6 | 365-366 | 365-366 |
| 7 | 380-383 | 380-383 |
| 8 | 387-392 | 387-392 |
С помощью сервиса SheeP была построена карта бета-листа для данной структуры.
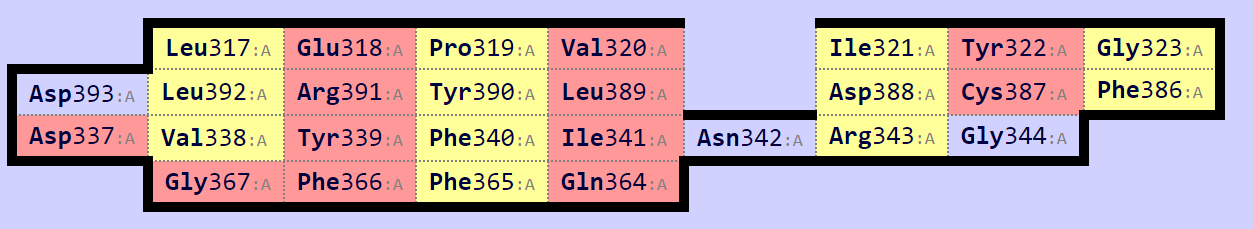
Рисунок 1.2. Карта β-листа для структуры 4D7S.
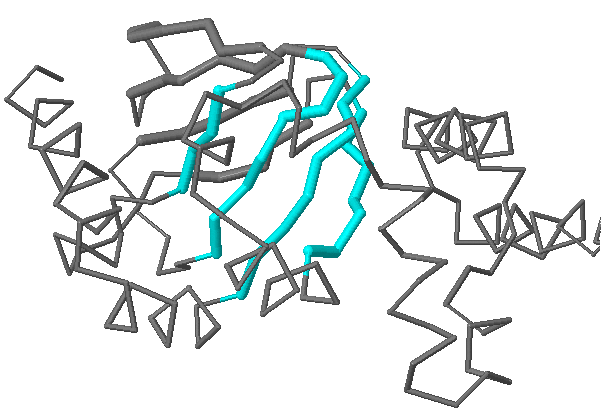
Рисунок 1.3. β-лист структуры 4D7S, состоящий из четырех тяжей, выделен голубым цветом.
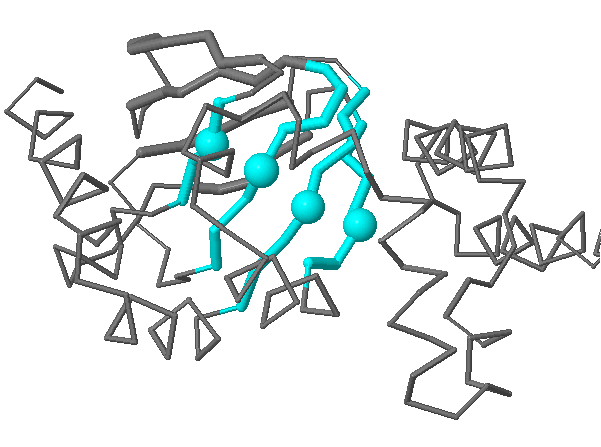
Рисунок 1.4. β-лист структуры 4D7S, Cα-атомы аминокислот, образующих один из хребтов, обозначены шариками.
d2: Совмещение структур.
Совмещение структуры 4IP7 и структурных гомологов
С помощью сервиса PDBeFold были выбраны 4 структурных гомолога для цепи A структуры 4IP7: 2G50:B, 4HYV:A, 4QG9:D, 4FXJ:A. Для данных структур были загружены выравнивание последовательностей по совмещению структур (попарное и множественное) и само совмещение структур (рисунок 2.1).
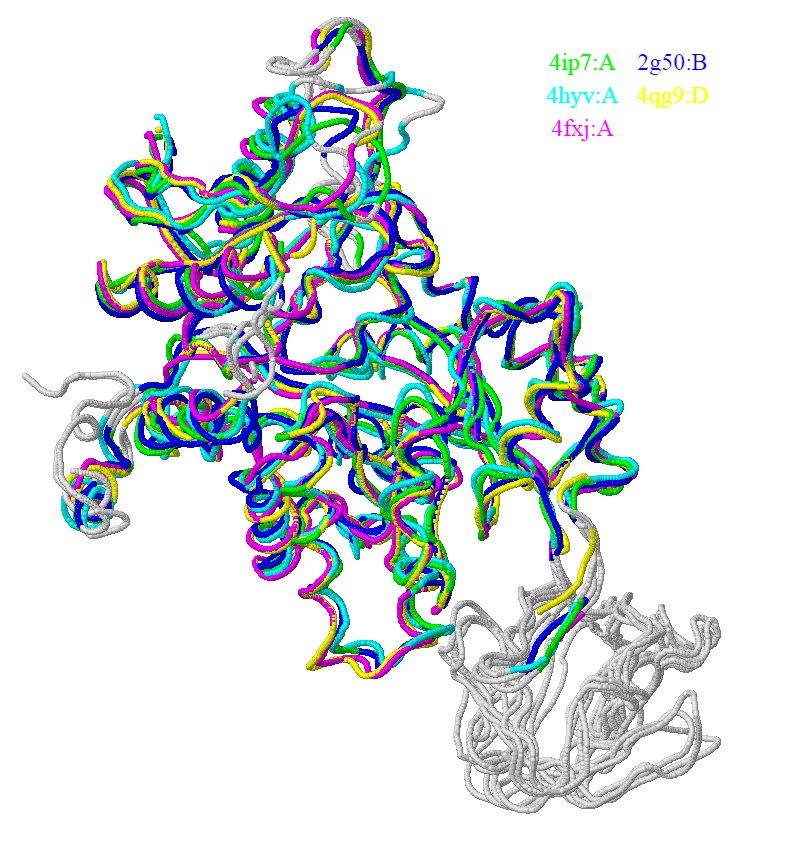
Рисунок 2.1. Совмещение структур 4IP7:A и его структурных гомологов, полученной с помощью сервиса PDBeFold.
Рисунок 2.3. Несовпадающие участки выравниваний.
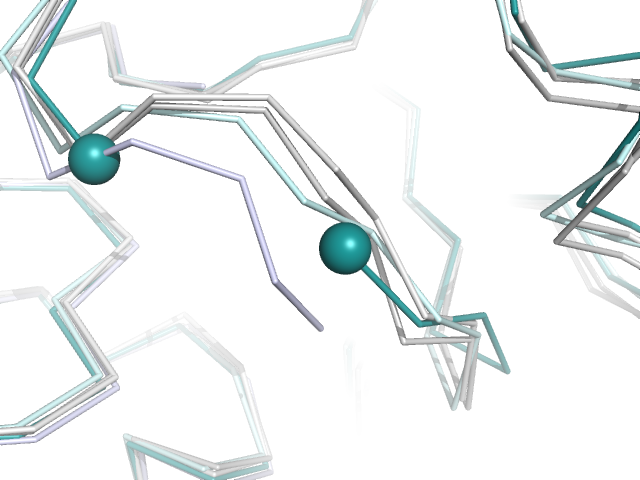
Рисунок 2.4. Разрыв в структуре 4IP7 (синий цвет). Шариками обозначены C-α- атомы остатков, расположенных на границах разрыва. Можно заметить, что рядом в одной из гомологичных структур тоже есть разрыв.
Совмещение по заданному выравниванию
Для выполнения данного заданиы были выбраны структуры константного домена человеческого Т-клеточного рецептора из цепи α (1oga, region d: 118-202) и из цепи β (1oga, region e: 119 - 245) (рисунок 2.5). Были получены файлы: alpha.pdb и beta.pdb.
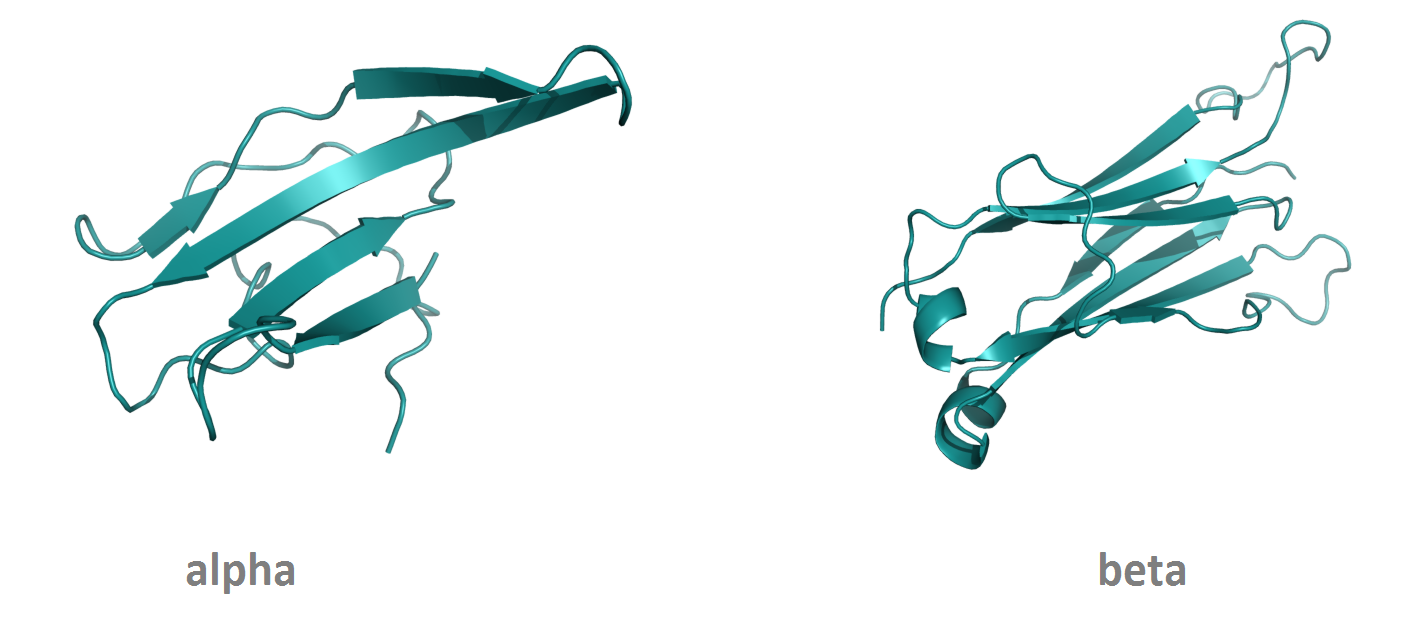
Рисунок 2.5. Домены человеческого Т-клеточного рецептора из цепи α и из цепи β.
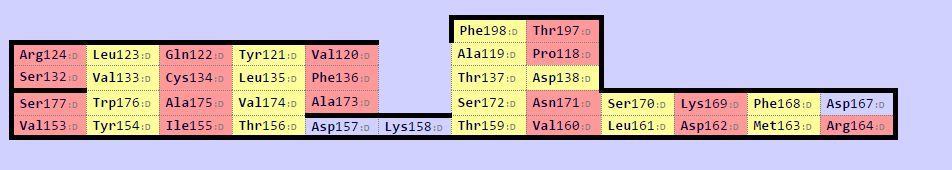
Рисунок 2.6. Карта бета-листа для домена из цепи α (map0).
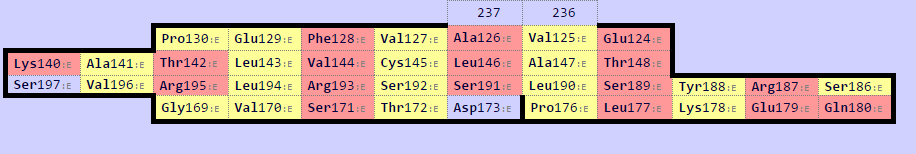
Рисунок 2.7. Карта бета-листа для домена из цепи β (map0).
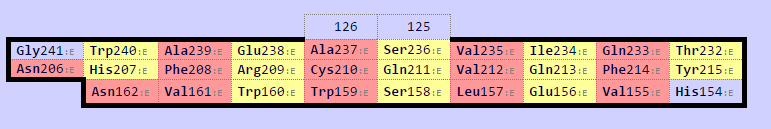
Рисунок 2.8. Карта бета-листа для домена из цепи β (map1).
select alph, alpha and name CA and resi (122+133+134+135+155+175) select bet, beta and name CA and resi (127+144+145+146+172+192) pair_fit alph, bet
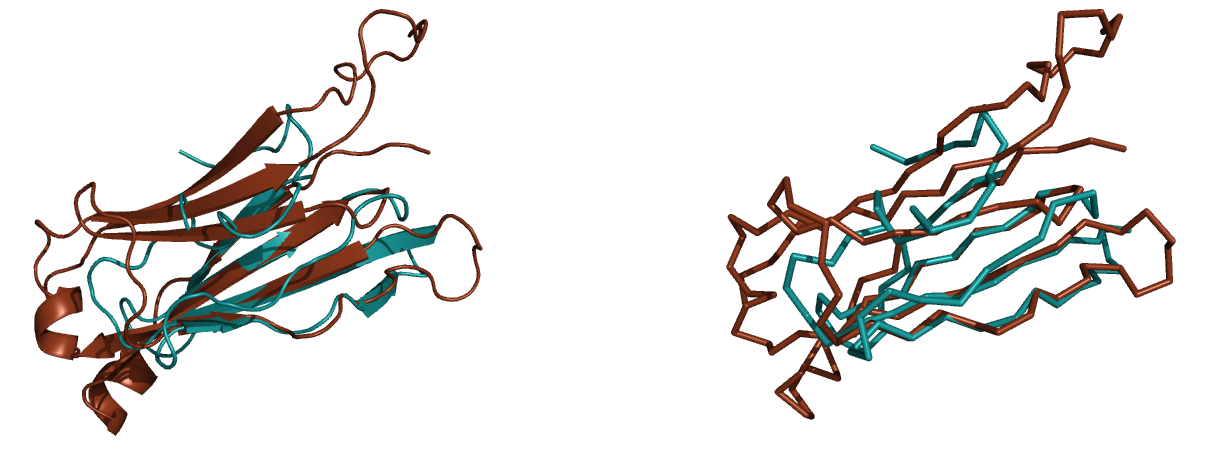
Рисунок 2.9. Совмещение доменов T-клеточного рецептора. Общий ход полипептидной цепи совпадает.
d3: Нахождение гидрофобных кластеров.
Поиск гидрофобных кластеров в структуре 4IP7 проводился с помощью сервиса CluD. Так как структура представляет собой гомотетрамер, поиск кластеров сначала проводился только для цепи A. При значениях 5.4 для порога расстояния и 10 для минимального размера кластера было найдено 8 кластеров, изображенных на рисунке 3.1:
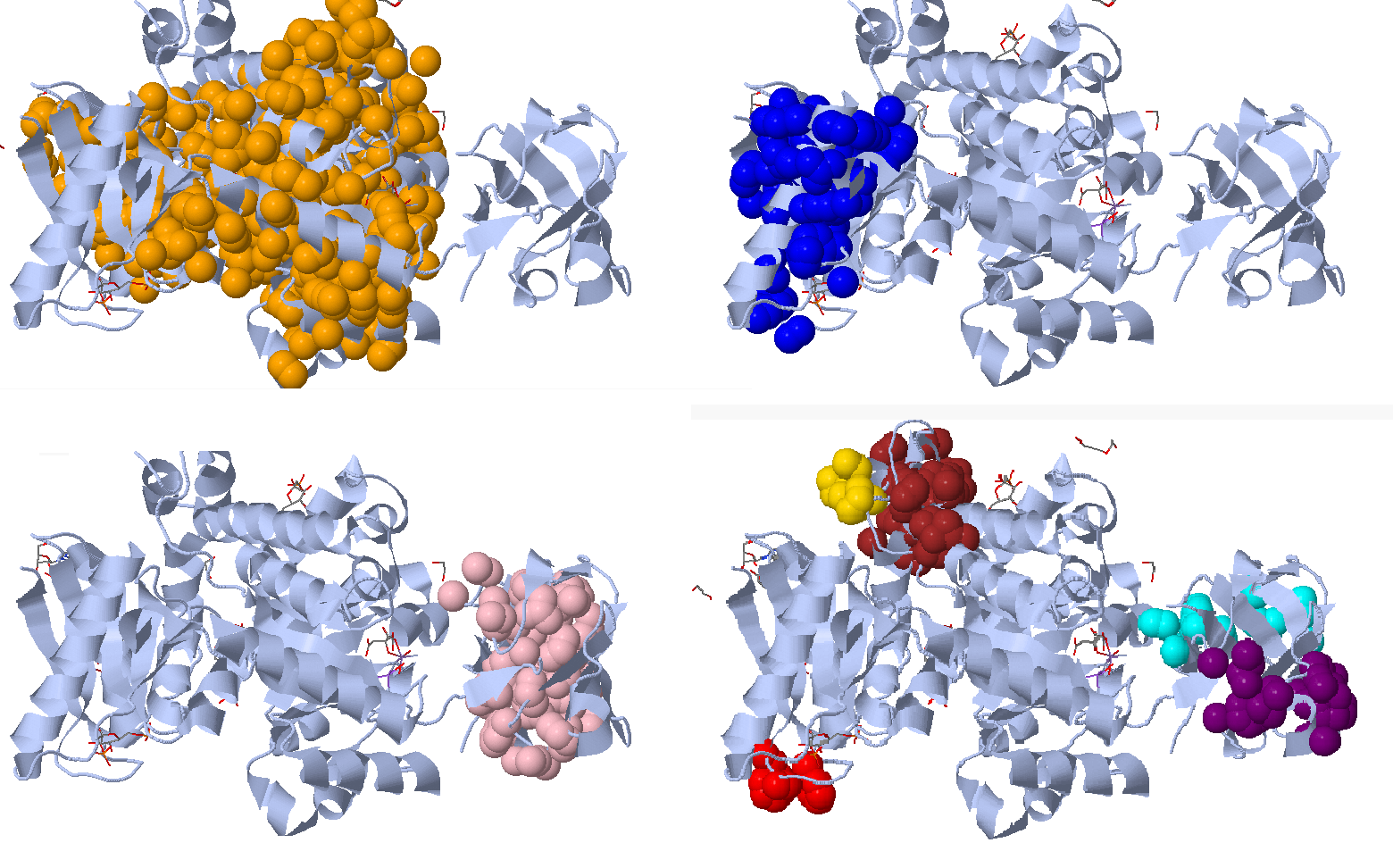
Рисунок 3.1. Найденные кластеры. 3 наиболее крупных кластера (больше 100 аминоксилотных остатков) изображены на отдельных структурах, остальные 5 изображены на одной структуре.
Можно заметить, что некоторые домены оказались разбиты на несколько кластеров, например β-бочонок, представляющий отдельный домен, был разделен программой на 3 отдельных кластера (рисунок 3.2).
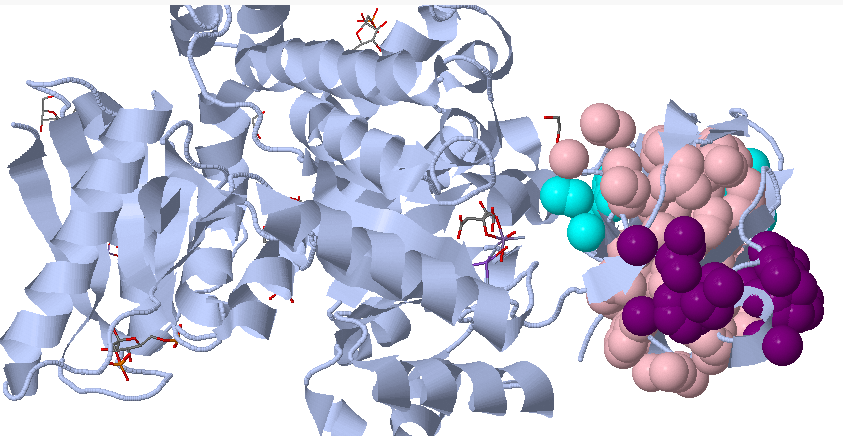
Рисунок 3.2. Структурный домен разбит на 3 отдельных кластера.
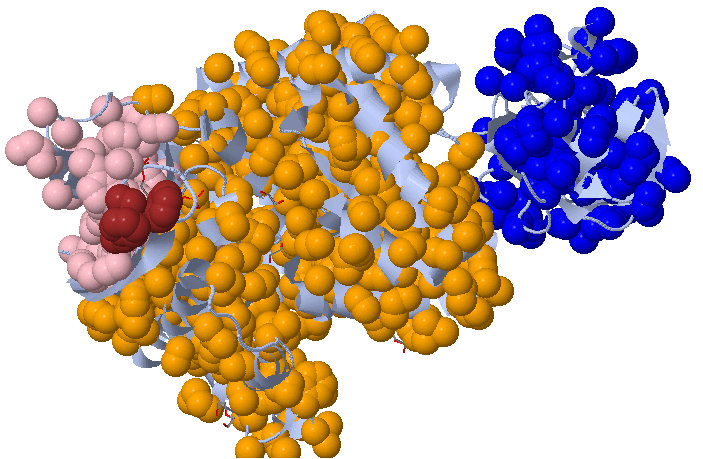
Рисунок 3.3. 4 кластера, полученные при новых параметрах поиска.
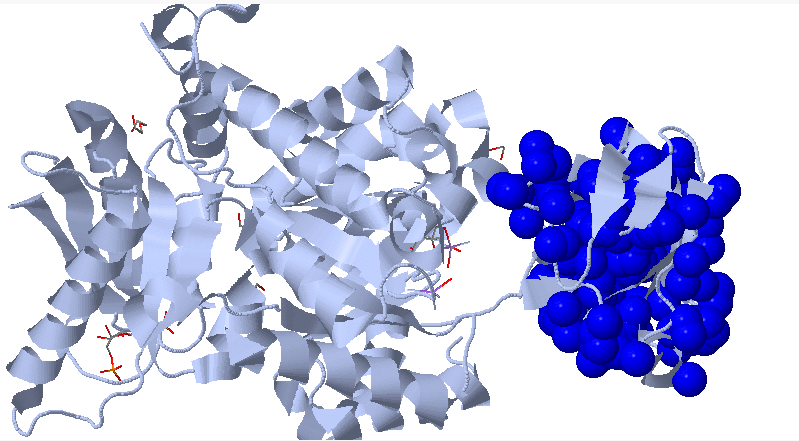
Рисунок 3.4. Один гидрофобный кластер соответствует домену белка.
Для анализа гидрофобных взаимодействий между цепями для той же структуры производился поиск гидрофобных кластеров сразу во всех цепях белка. Был установлен порог расстояния 4.5 и минимальный размер кластера 10. В результате было выявлено 2 симметричных гидрофобных кластера, общих для цепей A и D и для цепей B и C (рисунок 3.5).
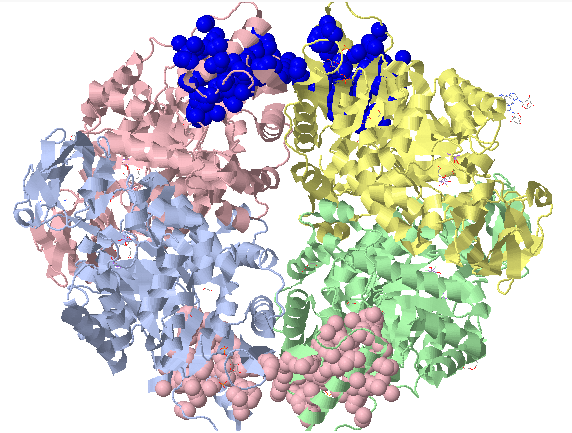
Рисунок 3.5. Гидрофобные кластеры, общие для цепей A и D и для цепей B и C.
d4: Построение поверхности.
Для комплекса димера пуринового репрессора с ДНК (биологическая единица 2PUE, рисунок 4.1) в программе PyMol были созданы изображения контакта мономеров белка между собой (рисунки 4.2 и 4.3) и контакта белка с ДНК (рисунки 4.4-4.7).
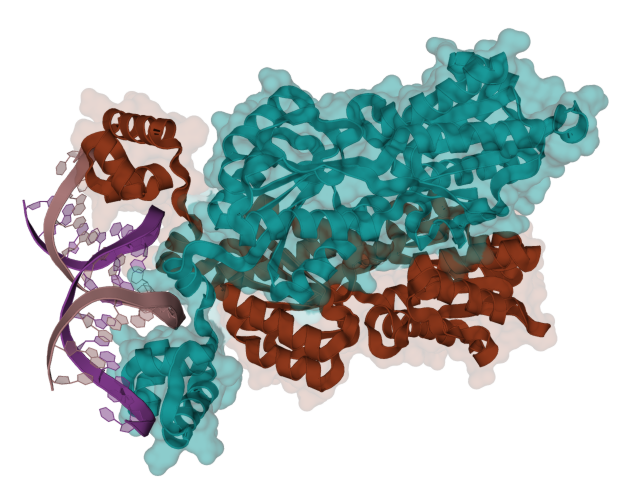
Рисунок 4.1. Биологическая единица 2PUE. (Белочек прильнул к ДНКашечке)
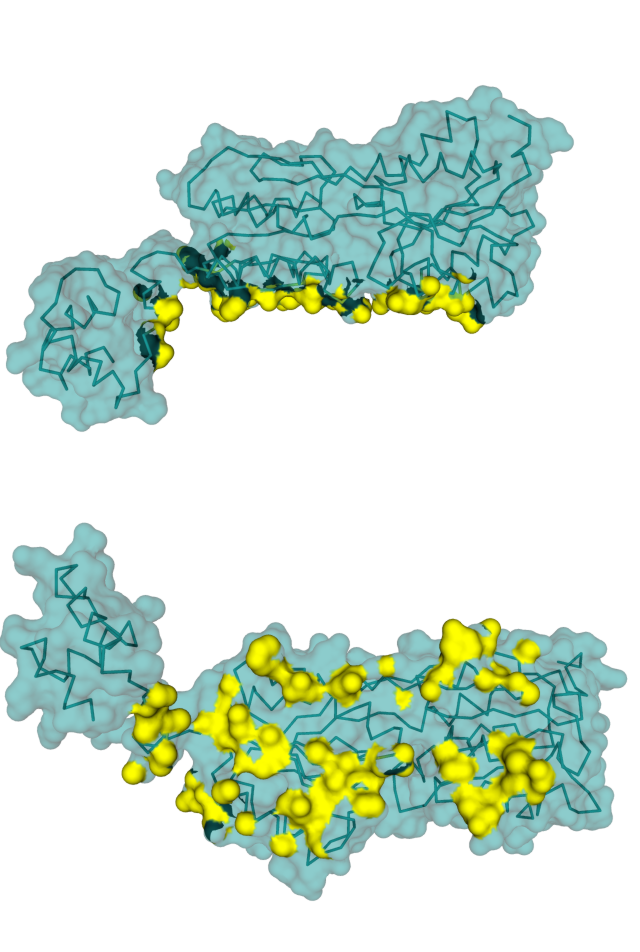
Рисунок 4.2. Поверхность контакта (обозначена желтым цветом) одной из субъединиц белка со второй субъединицей.
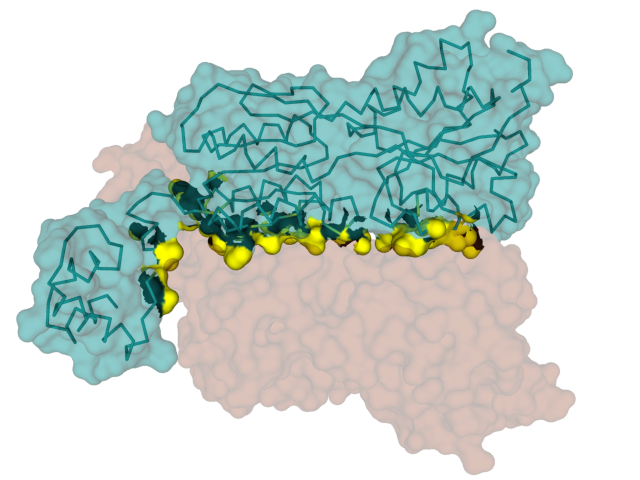
Рисунок 4.3. Две субъединицы белка 2PUE и поверхность контакта между ними (обозначена желтым цветом).
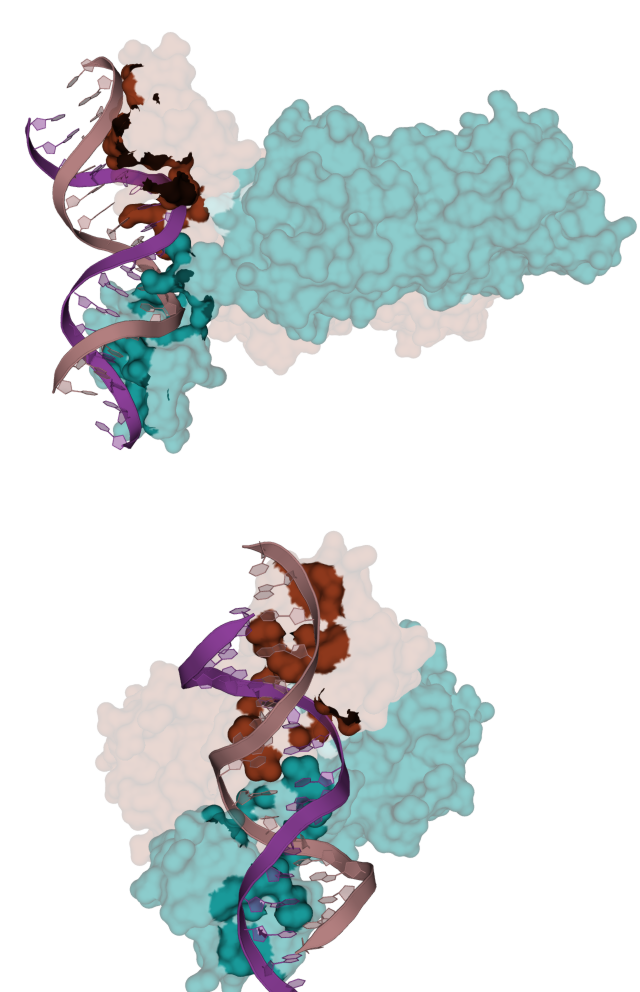
Рисунок 4.4. Контакт димера пуринового репрессора с ДНК. На рисунке показана белковая поверхность контакта.
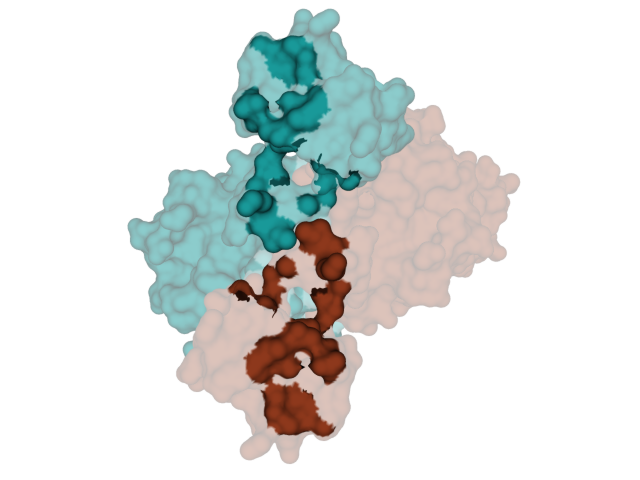
Рисунок 4.5. Белковая поверхность контакта с ДНК.
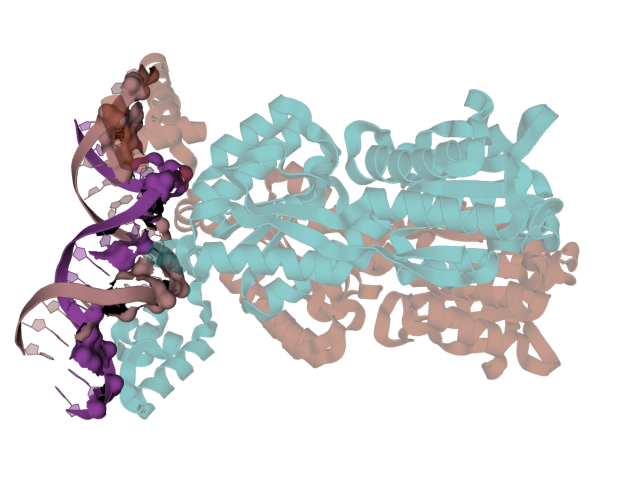
Рисунок 4.6. Контакт ДНК с димером пуринового репрессора. На рисунке показана поверхность ДНК, контактирующая с белком.
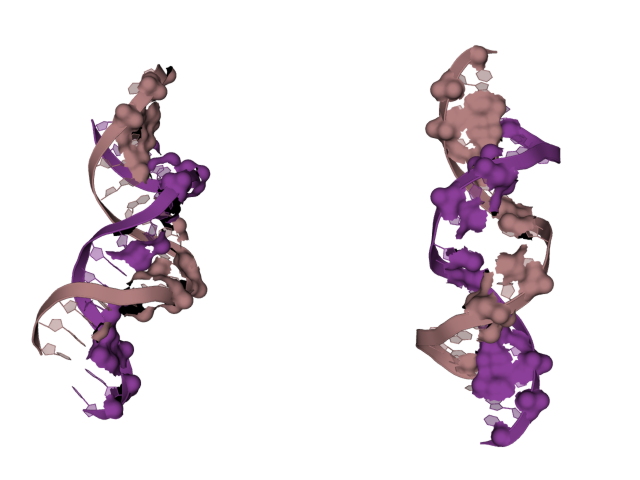
Рисунок 4.7. Поверхность ДНК, контактирующая с белком.
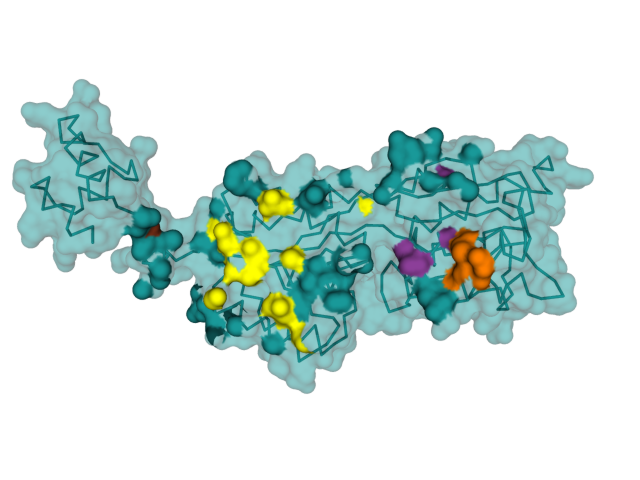
Рисунок 4.8. Поверхность контакта субъединиц белка. Каждый цвет соответствует гидрофобному кластеру, найденному Clud.
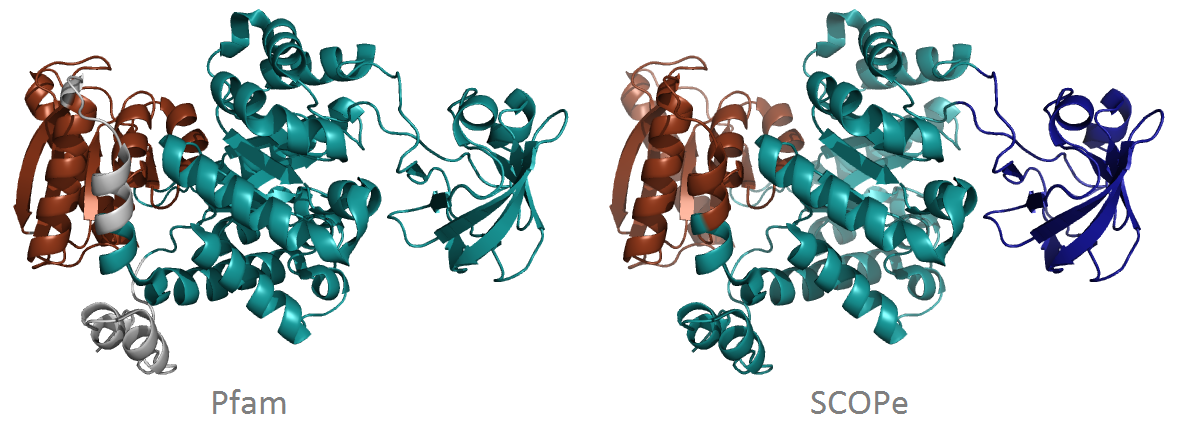
d5: Сравнение доменов SCOP и Pfam.
Для структуры 2VGB был произведен поиск доменов в Pfam и SCOPe. Структура 2VGB - пируваткиназа эритроцитов человека, гомотетрамер, сотоящий из четырех одинаковых субъединиц (A, B, C и D). Указанные ниже домены найдены для цепи A, и совпадают с доменами для остальных цепей.
Домены, найденные Pfam: домен PK с координатами 85-438 и домен PK_C с координатами 452-572.
Домены, найденные SCOPe: домен d2vgba1 с координатами 160-261, домен d2vgba2 с координатами 57-159 и 262-439 и домен d2vgba3 с координатами 440-573.
© Наталья Ланина
e-mail: n.lanina@fbb.msu.ru
последний раз обновлялось: 6.3.15