Молекулярная динамика биологических молекул в GROMACS.
Цель данного занятия - ознакомится с возможностями моделирования молекулярной динамики. Для этого будем использовать пакет молекулярной динамики Gromacs.
Моделирование самосборки липидного бислоя.
0. Настройка соединения с суперкомпьютером.
rm ~/.ssh mkdir ~/.ssh cp /home/preps/golovin/skif/* ~/.ssh cat /home/preps/golovin/skif/config >> ~/.ssh/config chmod 600 ~/.ssh/chemprac
1.Исходные файлы.
Для работы нам даны следующие файлы:
- файл дополнительной топологии для липида DPPC, dppc.itp;
- параметры для липидов lipid.itp;
- координаты одного липида dppc.gro;
- файл-заготовка тополгии системы b.top;
- файл праметров для минимизации энергии em.mdp;
- файл праметров для "утряски" воды pr.mdp;
- файл праметров для молекулярной динамики md.mdp;
2. Подготовка системы.
Сначала на основе одного липида создаем ячейку с 64 липидами:
genconf -f dppc.gro -o b_64.gro -nbox 4 4 4Преобразовываем dppc.gro и b_64.gro в .pdb-файлы:
editconf -f dppc.gro -o dppc.pdb editconf -f b_64.gro -o b_64.pdbПолученные структуры изображены на рисунках 1,2.
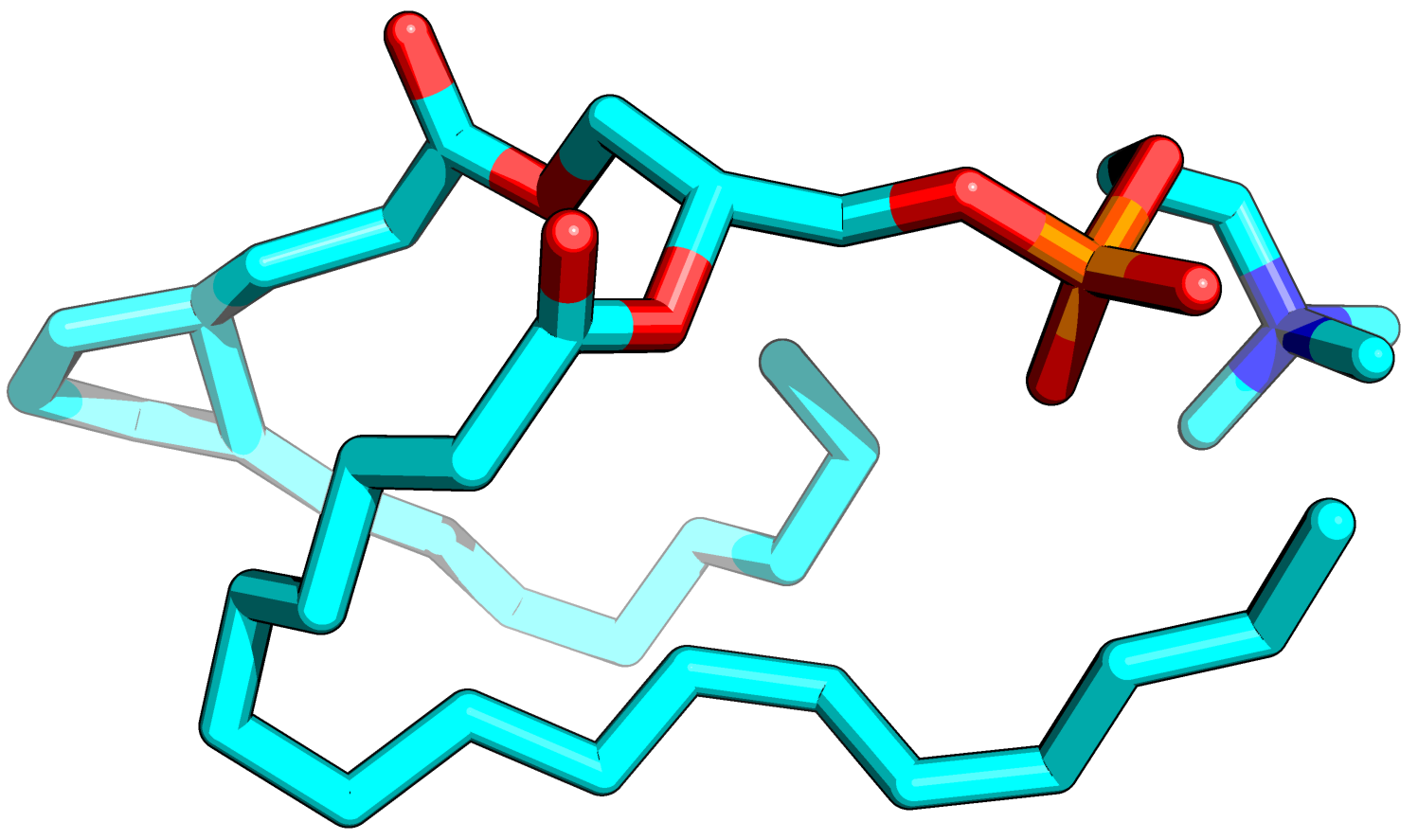
Рисунок 1. Структура одной молекулы липида.
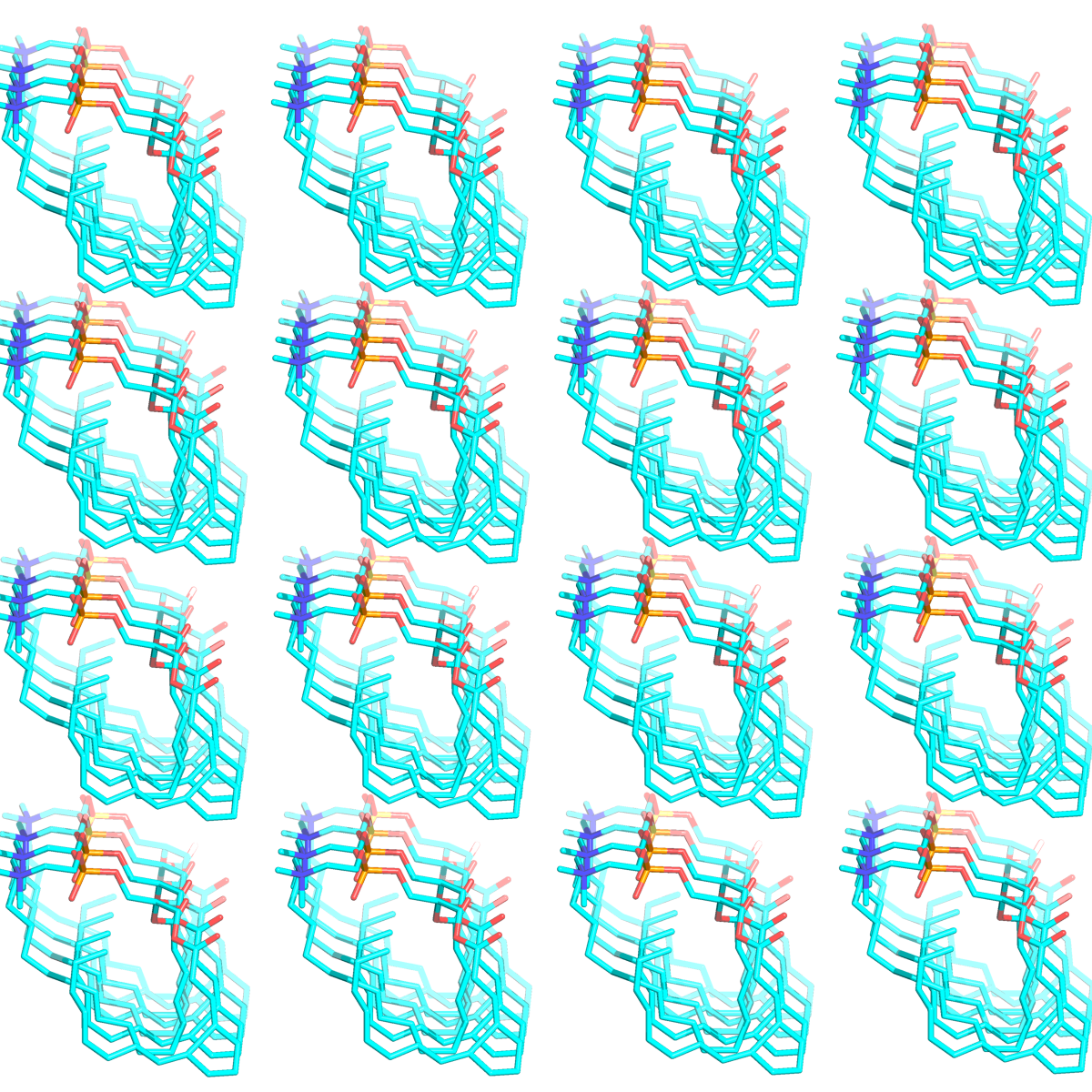
Рисунок 2. Ячейка, содержащая 64 молекулы липида.
[ molecules ] ; name number DPPC 64Делаем небольшой отступ в ячейке от липидов, что бы добавить примерно 2500 молекул воды:
editconf -f b_64.gro -o b_ec -d 0.5Проводим оптимизацию геометрии системы, чтобы удалить "плохие" контакты молекул:
grompp -f em -c b_ec -p b -o b_em -maxwarn 2 mdrun -deffnm b_em -vНачальное значение максимальной силы: 4.37970e+05
Конечное значение максимальной силы: 6.16887e+02
Потенциальная энергия: 3.2059077e+03
Максимальное значение силы: 6.1937860e+02 на атоме 915
Норма силы: 1.7839124e+02
Более подробную информацию можно найти в файле force.txt.
Добавляем в ячейку молекулы воды типа spc:
genbox -cp b_em -p b -cs spc216 -o b_sПроводим "утряску" воды:
grompp -f em -c b_s -p b -o b_empr -maxwarn 1 mdrun -deffnm b_empr -v grompp -f pr -c b_empr -p b -o b_pr -maxwarn 1 mdrun -deffnm b_pr -vПреобразовываем b_pr.gro и b_s.gro в .pdb-файлы:
editconf -f b_pr.gro -o b_pr.pdb editconf -f b_s.gro -o b_s.pdbПолученные структуры представлены на рисунках 3-5.

Рисунок 3. Ячейка с липидами после добавления воды. Приведено два изображения ячейки, с выделенными молекулами воды (слева) и с выделенными молекулами липида (справа).

Рисунок 4. Ячейка с липидами после добавления воды и ее "утряски". Приведено два изображения ячейки, с выделенными молекулами воды (слева) и с выделенными молекулами липида (справа).
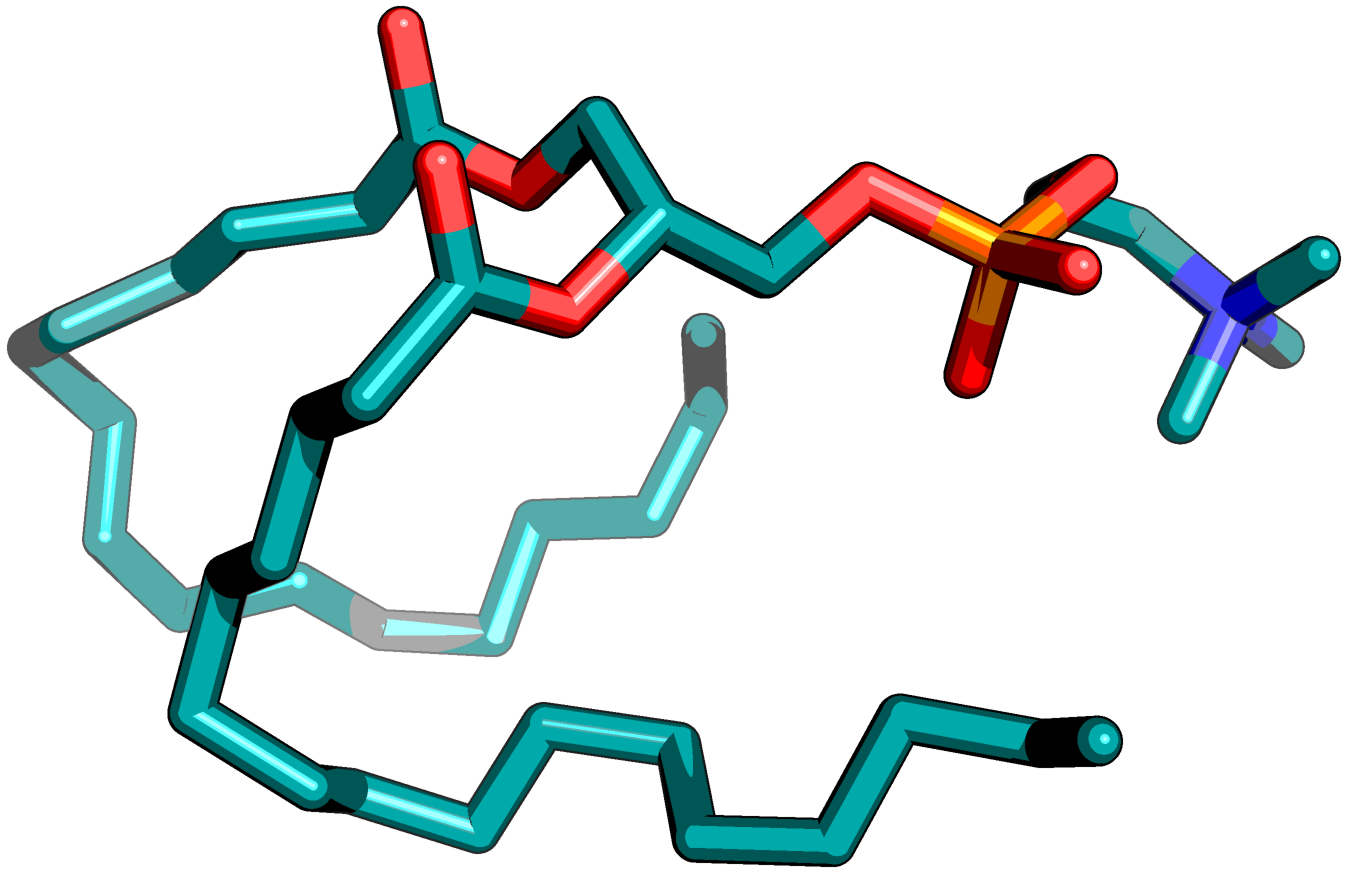
Рисунок 5. Молекула липида после "утряски".
3. Запуск задачи на суперкомпьтере.
Копируем файлы на суперкомпьютер (работа проводилась в директории Lanina):
cd .. scp -r ./Lanina skif:_scratch/fbbЗапускаем тестовое моделирование на суперкомпьтере:
ssh skif cd _scratch/fbb/Lanina cp /home/users/golovin/progs/share/gromacs/top/residuetypes.dat . grompp -f md -c b_pr -p b -o b_md -maxwarn 1 sbatch -n 4 -e error.log -o output.log -t 5 -p test impi /mnt/msu/ users/fbbstudent/gmx-4.6.7-intel-impi-mkl-single/bin/mdrun_mpi -deffnm b_md -vНомер задачи 1043338. Программа не выдает ошибок, но и не создает файл mdrun_mpi.out-1043338 (который должен содержать ход счета).
Запускаем основное моделирование на суперкомпьтере:
sbatch -n 16 -e error.log -o output.log -t 500 -p regular4 impi /mnt/msu/ users/fbbstudent/gmx-4.6.7-intel-impi-mkl-single/bin/mdrun_mpi-deffnm b_md -vНомер задачи 1044443.
Все файлы, использованные и полученные при выполнении практикума, можно посмотреть в папке.
© Наталья Ланина
e-mail: n.lanina@fbb.msu.ru
последний раз обновлялось: 2.4.15