Гомологичное моделирование комплекса белка с лигандом.
Для выполнения работы был выбран лизоцим Ostrea edulis LYS_OSTED
1. Подготовка файла выравнивания для программы MODELLER.
С помощью Clustal строим выравнивание для структуры 1lmp и выбранного белка, сохраняем полученное выравнивание в формате .pir.
Далее модифицируем файол с выравниванием следующим образом: переименовали последовательности P1;seq и P1;1lmp для моделируемого белка и белка-образца соответсвтенно. После имени последовательности моделируемого белка добавляем строчку (описывает входные параметры последовательности для modeller):
sequence:ХХХХХ::::::: 0.00: 0.00После имени последовательности белка-образца добавляем строчку (описывает, какой файл содержит структуру белка с этой последовательностью, номера первой и последней аминокислот в структуре, идентификатор цепи...):
structureX:1lmp_now.ent:1 :A: 132 :A:undefined:undefined:-1.00:-1.00В конце каждой последовательности добавляем символы: /.
Получили файл ali.pir.
2. Подготовка файла со структурой для программы MODELLER.
Удаляем всю воду из структуры (в текстовом редакторе). Затем всем атомам лиганда присваиваем один и тот же номер "остатка" (MODELLER считает, что один лиганд = один остаток) и модифицируем имена атомов каждого остатка, добавив в конец буквы A, B, C. Смысл операции в том, что атомы остатка 130 имели индекс А, атомы остатка 131 имели индекс В и т.д. Сохраняем новый файл как 1lmp_now.ent.
3. Создание управляющего скрипта.
Дана заготовка скрипта:
from modeller.automodel import *
class mymodel(automodel):
def special_restraints(self, aln):
rsr = self.restraints
for ids in (('OD1:98:A', 'O6A:131:A'),
('N:65:A', 'O7B:132:A'),
('OD2:73:A', 'O1C:133:A')):
atoms = [self.atoms[i] for i in ids]
rsr.add(forms.upper_bound(group=physical.upper_distance,
feature=features.distance(*atoms), mean=3.5, stdev=0.1))
env = environ()
env.io.hetatm = True
a = mymodel(env, alnfile='test1.ali', knowns=('1lmp'), sequence='seq')
a.starting_model = 1
a.ending_model = 5
a.make()
Необходимо модифицировать заготовку: редактируем строчки, в которых указано, какие водородные связи белка с лигандом должны быть в построенной модели. Для этого определяем, какие водородные связи между белком и лигандом присутствуют в образце. В моделируемом белке 137 аминокислот, значит номер "остатка" лиганда 138, а номера остатков в белке надо определяем согласно выравниванию. Получили скрипт lis_osted.py.Запускаем исполнение скрипта:
mod9v7 lys_osted.py &Получили пять моделей:
модель 1
модель 2
модель 3
модель 4
модель 5
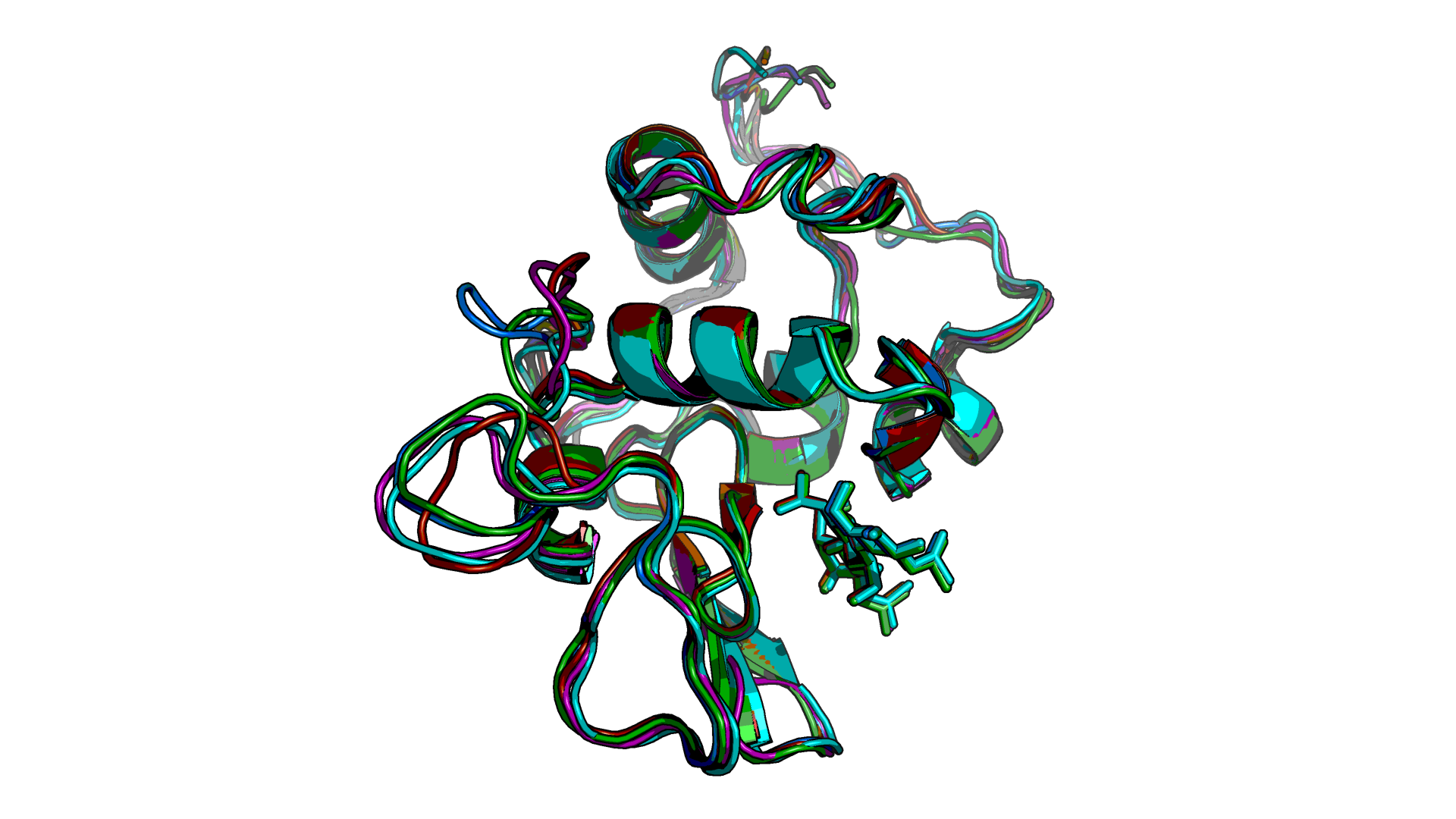
4. Сравнение моделей.
Все модели очень похожи друг на друга (рисунок 1), поэтому визульно оценить их качество нельзя. Есть некоторые расхождения в петлях, но визуально выбрать лучшую модель невозможно. Для сравнения проверим некоторые характеристики сервером WHATIF:| Критерий | Модель1 | Модель2 | Модель3 | Модель4 | Модель5 |
| Карта Рамачандрана | -1.643 | -0.998 | -1.230 | -1.499 | -1.105 |
| Omega | 181.620+/-4.964 | 181.280+/-4.553 | 181.750+/-4.255 | 181.150+/-6.062 | 181.300+/-4.049 |
| Packing quality control Z-score | -5.31 | -5.17 | -5.32 | -5.49 | -5.03 |
© Наталья Ланина
e-mail: n.lanina@fbb.msu.ru
последний раз обновлялось: 21.5.15