Работа с PyMol.
1. Введение мутации в белок
Мутация вводилась в зону контакта белка 1LMP и лиганда (рисунок 1).
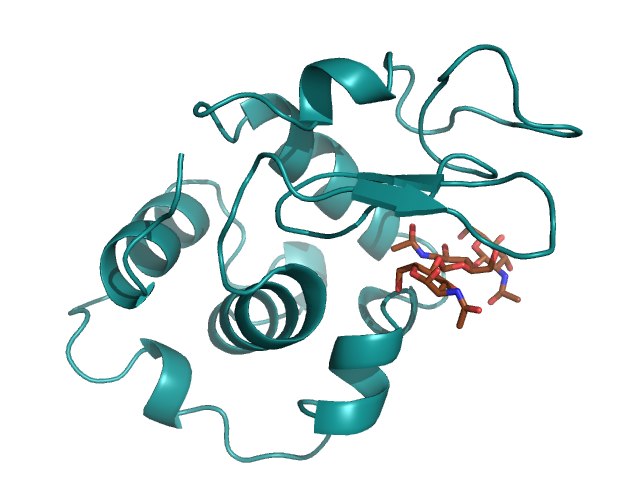
Рисунок 1. Структура белка 1LMP (изображена голубым цветом) с лигандом (изображен коричневым цветом).
Оценка зоны контакта
Для того, чтобы выбрать остаток, взаимодействуюзий с лигандами, были найдены атомы азота и кислорода, расстояние между которыми не превышает 3.5 Å (длину водородной связи):
remove resn HOH select ligand, 1LMP and resn nag+ndg select cont, 1LMP and not name C* within 3.5 of (ligand and not name C*) select lcont, ligand and not name C* within 3.5 of (1LMP and not name C*) select rcont, byres cont distance bond, cont, lcont, 3.5На рисунке 2 показаны остатки, обеспечивающие взаимодействия белка с лигандом.
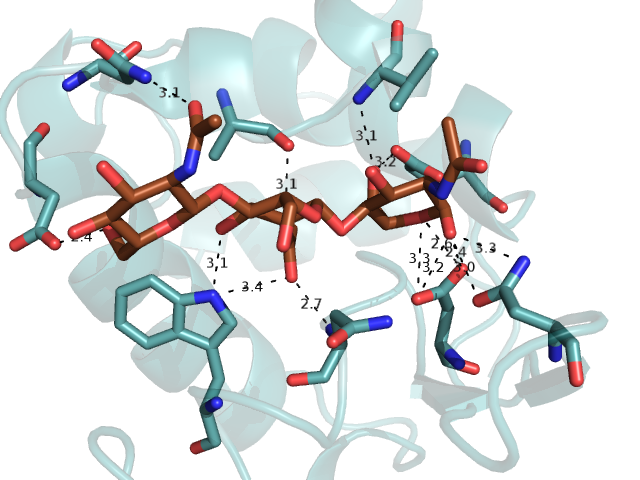
Рисунок 2. Водородные связи между атомами азота и кислорода белка и лиганда.
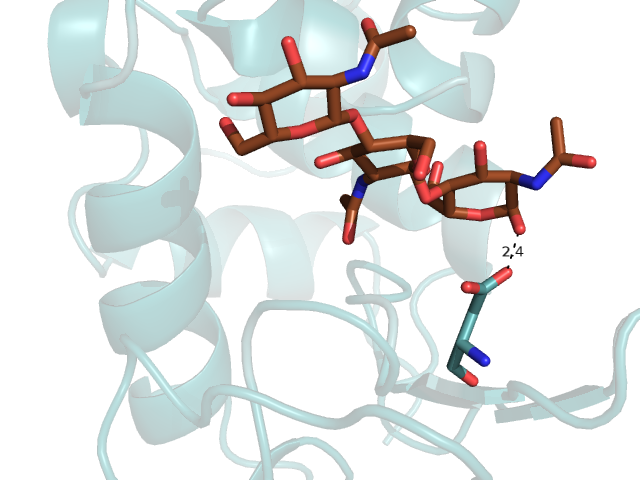
Рисунок 3. Остаток Asp 52, взаимодействующий с лигандом.
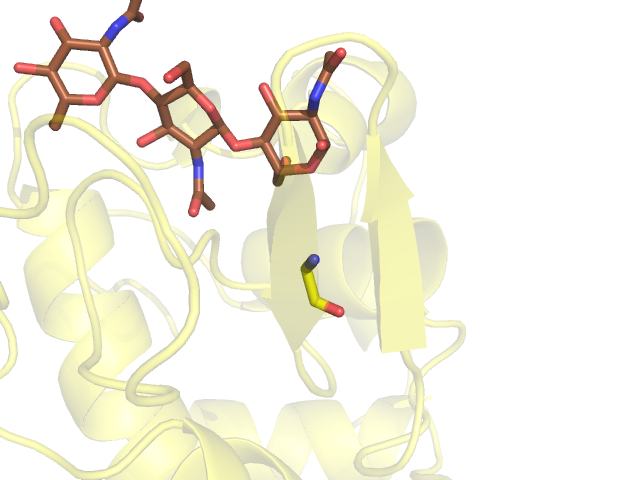
Рисунок 4. Остаток Gly 52 в новой структуре, не взаимодействующий с лигандом.
2. Создание анимационного ролика
Для создания ролика открываем структуры 1LMP и 1LMP_mut. Сначала переносим структуру 1LMP влево и выбираем остатки Gly52 и Asp52: <
select gly52, 1LMP and resi 52 select asp52, 1LMP_mut and resi 52 translate [-50,0,0], 1LMPДалее создаем ролик, при этом используем следующие команды:
mset 1x420 frame 1 mview store mview store, object=1LMP mview store, object=1LMP_mut #фокусируемся на остатке Gly 52 frame 30 zoom gly52 mview store mview store, object=1LMP mview store, object=1LMP_mut ... #совмещаем белки frame 220 translate [25,0,0], object=1LMP translate [-25,0,0], object=1LMP mview store mview store, object=1LMP mview store, object=1LMP_mut ... mview interpolate, object=1LMP mview reinterpolate set ray_trace_frames,1 mpng 1LMP_mutЕсли возникли проблемы с воспроизведением видео, его можно посмотреть на YouTube.
3. Присоединение флуоресцентной метки
Присоединим к белку 1LMP флуоресцентную метку TAMRA через сложноэфирную связь. Сначала скачаем метку (рисунок 5).
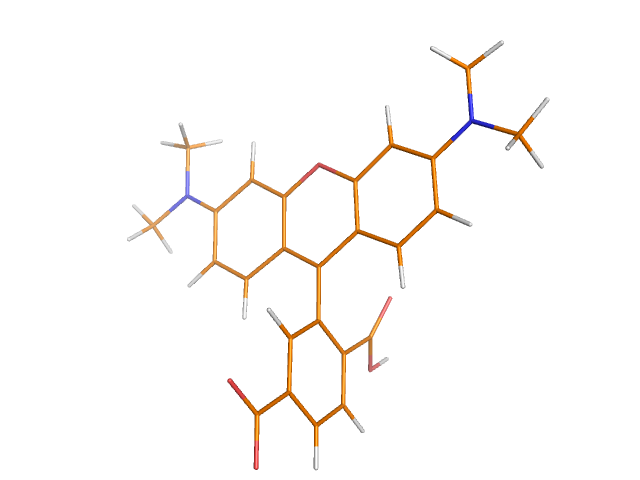
Рисунок 5. Изображение структуры флуоресцентной метки TAMRA.
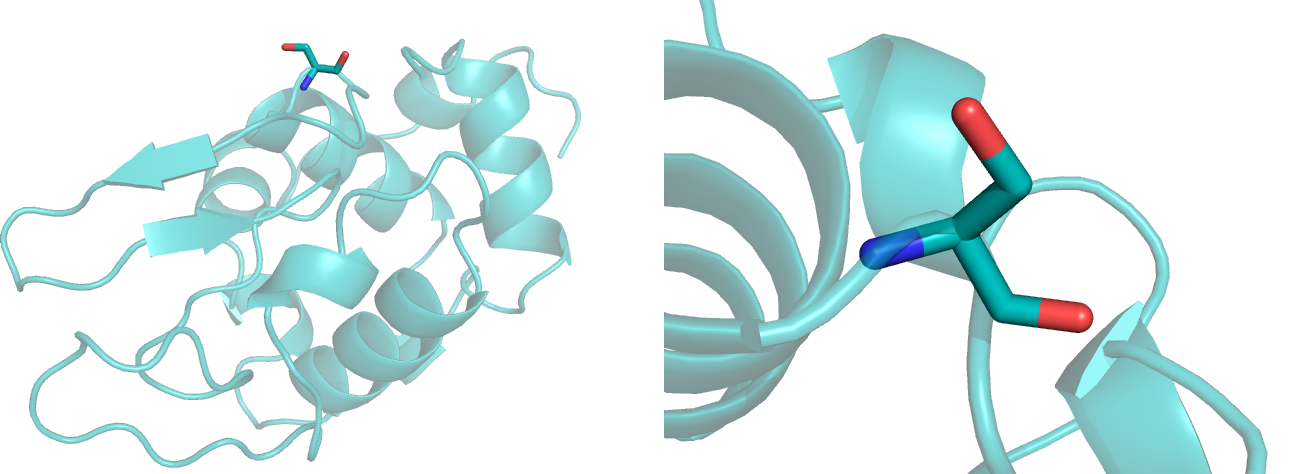
Рисунок 6. Выбранный для присоединения метки аминокислотный остаток Ser 37.
select sero, 1LMP and resn 37 and name OG fuse sero, tamracСначала получаем связь с торсионным углом равным 64, при этом метка перекрывается с белком. Вручную поворачиваем метку так, чтобы перекрытий не возникало. Конечный торсионный угол составляет 14,5. Изображение полученного соединения белка с флуоресцентной меткой приведено на рисунке 7.
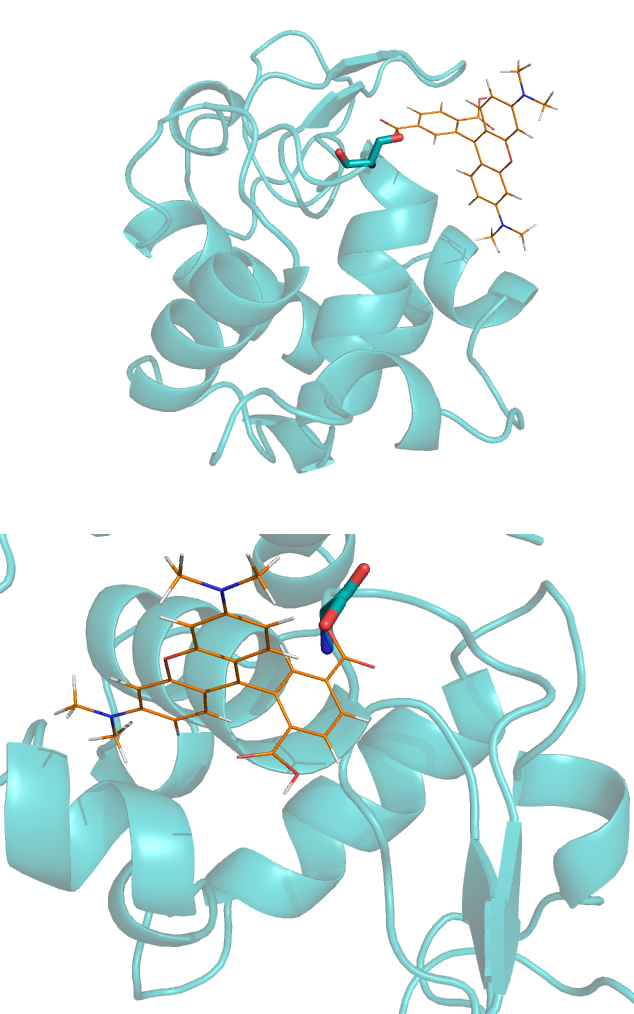
Рисунок 7. Выбранный для присоединения метки аминокислотный остаток Ser 37.
4. Построение поли-аланиновой альфа-спирали
Для построения полиаланиновой спирали был использован скрипт helix.py:
cmd.reinitialize()
phi = '-65'
psi = '-45'
cmd.fragment('ALA')
n=100
for i in range(2,n+1):
cmd.do('edit i. %i and n. c' % i)
cmd.do('editor.attach_amino_acid("pk1","ALA")')
for i in range(2,100):
cmd.set_dihedral("i. %i & n. c" % i,
"i. %i & n. n" % (i+1),
"i. %i & n. ca" % (i+1),
"i. %i & n. c" % (i+1), phi)
cmd.set_dihedral("i. %i and n. n" % i,
"i. %i and n. ca" % i,
"i. %i and n. c" % i,
"i. %i and n. n" % (i+1), psi)
cmd.remove('n. *h*')
cmd.save('helix.pdb')
Значения торсионных углов φ и ψ были приняты равными -65 и -45 соответственно. На рисунке 8 показана карта Рамачандрана для трипептида AlaAlaAla, взятая с сайта http://www.iop.vast.ac.vn/theor/conferences/smp/1st/kaminuma/UCSFComputerGraphicsLab/AAA.html
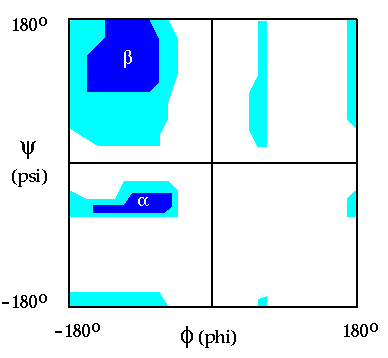
Рисунок 8. Карта Рамачандрана для трипептида AlaAlaAla.

Рисунок 9. Изображение структуры полиаланиновой спирали.
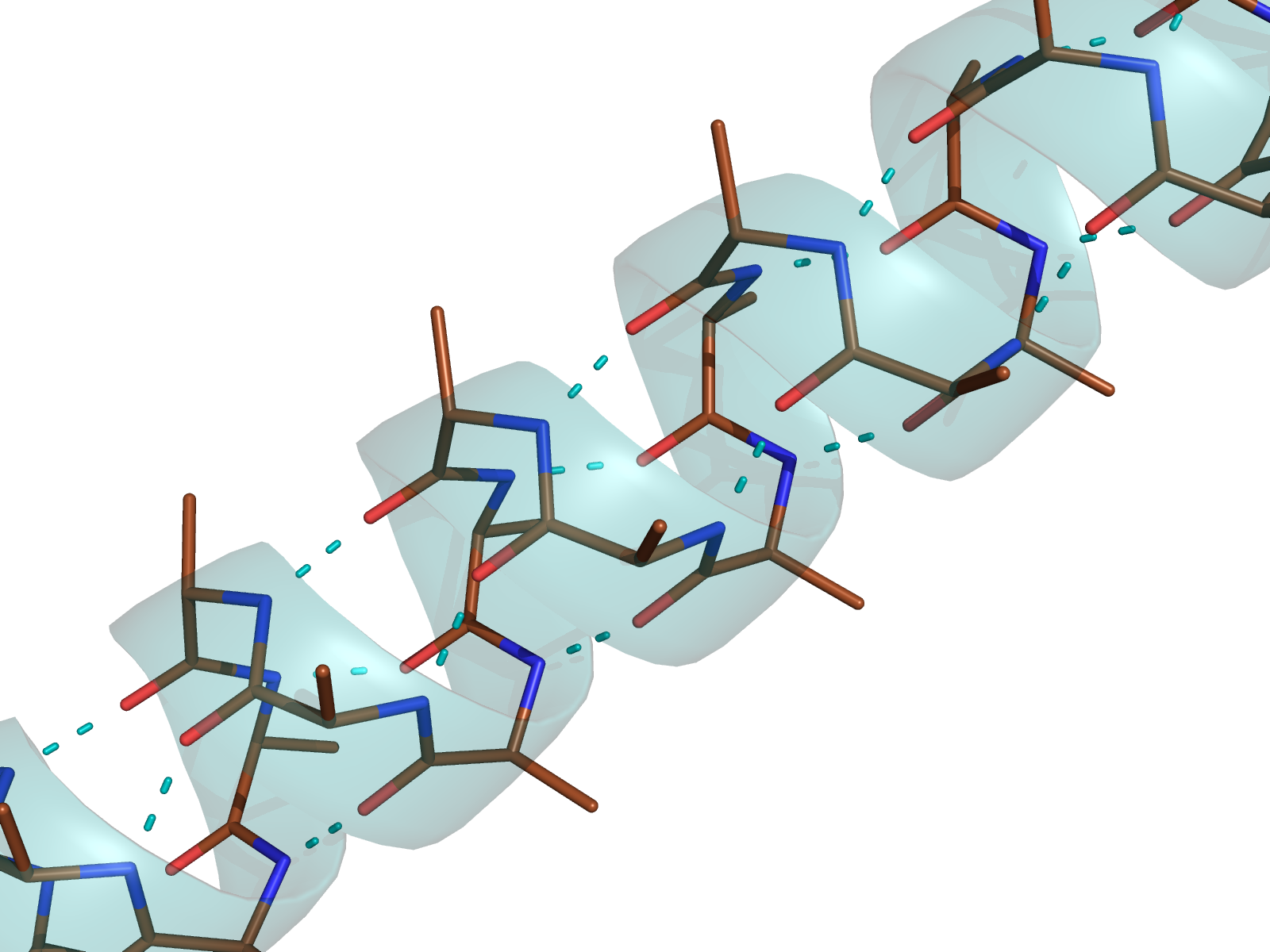
Рисунок 10. Изображение структуры полиаланиновой спирали. Голубым пунктиром показаны водородные связи, обеспечивающиес структуру спирали. Также в формате cartoons показан ход спирали.

Рисунок 11. Поверхность полиаланиновой спирали. Голубым цветом показаны атомы углерода, синим - атомы азота, красным - атомы кислорода.
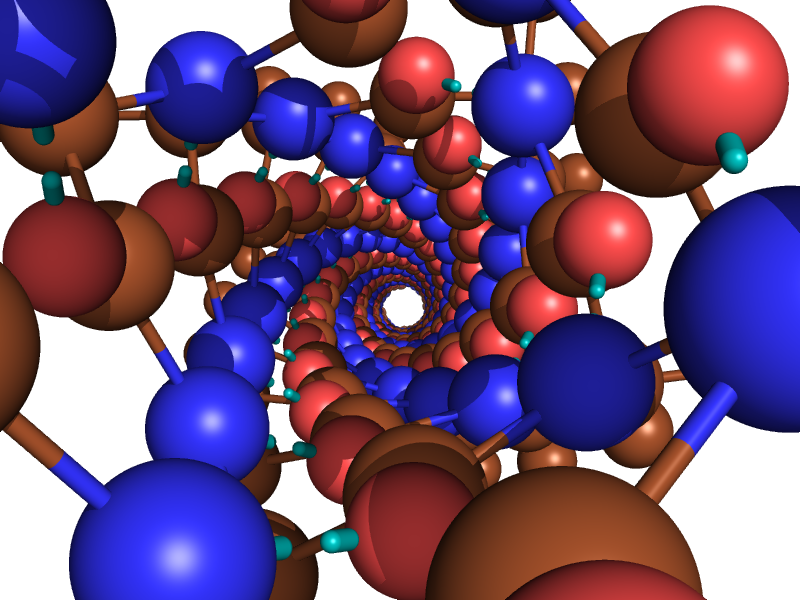
Рисунок 12. Структура спирали изнутри. Коричневым цветом показаны атомы углерода, синим - атомы азота, красным - атомы кислорода.
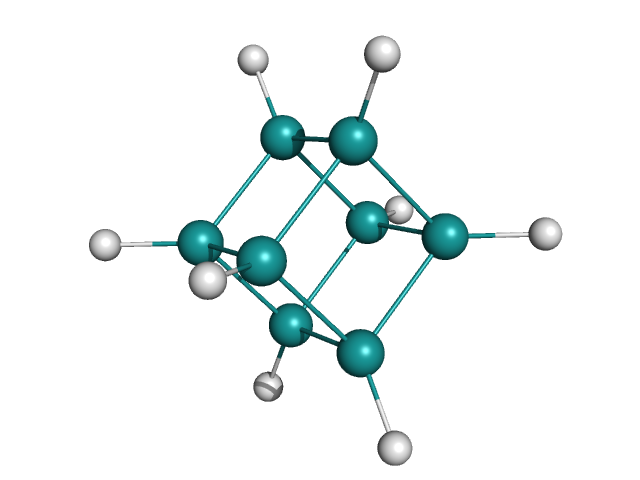
5. Построение структуры кубана
С помощью программы obgen была построена структура кубана.
$ echo C12C3C4C1C5C2C3C45 cubane > cubane.smi $ obgen cubane.smi > cubane.molПолученный файл cubane.mol был открыт в программе PyMol. Полученное изображение структуры показано на рисунке 13.
© Наталья Ланина
e-mail: n.lanina@fbb.msu.ru
последний раз обновлялось: 28.3.15