NGS: de-novo сборка генома
Сборка генома и его анализ
Архив с чтениями был получен с помощи команды wget.
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/009/SRR4240359/SRR4240359.fastq.gz
Далее был создан файл с адаптерами для их дальнейшего удаления.
cat /mnt/scratch/NGS/adapters/* > adapters.fa
Потом из чтений сначала удалялись адаптеры, а потом чтения с качеством ниже 20 и длиной меньше 32.
-
TrimmomaticSE -phred33 SRR4240359.fastq.gz SRR4240359_trimmed.fastq.gz ILLUMINACLIP:adapters.fa:2:7:7 -threads 15 -trimlog trimmomatic.log -
TrimmomaticSE -phred33 SRR4240359_trimmed.fastq.gz SRR4240359_filtered_trimmed.fastq.gz TRAILING:20 MINLEN:32 -threads 15 -trimlog trimmomatic.log
Изначально было 13 557 938 чтений. В результате первой программы осталось 13 502 066 последовательностей (99.59%). Потом фильтрация уменьшила число до 12 184 080 чтений (89.87% от изначального числа). Размер файла сократился сначала с 445 MB до 443 MB, а потом после фильтрации до 385 MB.
Поскольку чтения теперь не короче 30 нуклеотидов, минимальной длиной k-мера для построения графа выберем 31. Для сборки будем использовать пакет программ velvet, а конкретно для этого программу velveth (-short указывает на короткие непарные чтения, а velvet — название папки).
velveth velvet 31 -short -fastq.gz SRR4240359_filtered_trimmed.fastq.gz
Теперь воспользуемся программой velvetg для сборки генома.
velvetg velvet
Из Log файла внутри папки velvet можно узнать, что N50 = 70 607. В файле stats.txt найдем три самых длинных контига и их покрытие (Табл. 1).
| ID | Длина | Покрытие |
|---|---|---|
| 11 | 125 674 | 44.55 |
| 1 | 108 447 | 42.01 |
| 14 | 71 403 | 39.41 |
Все контиги с аномально большими или аномально малыми покрытиями имееют длину меньше k (в нашем случае 31). Такие контиги не попадут в файл contigs.fa, потому что в него попадают только контиги длиной больше или равной максимальной длине k-меров. При помощи grep удалось узнать, что таких 285.
grep '>' contigs.fa | wc -l
Анализ контигов с помощью megablast
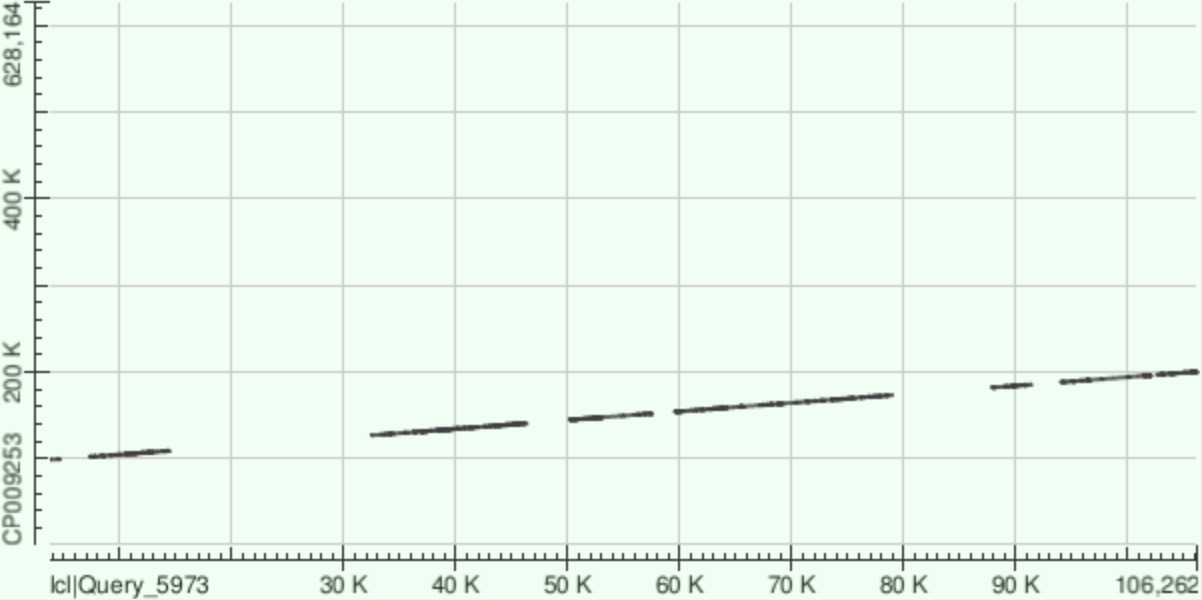
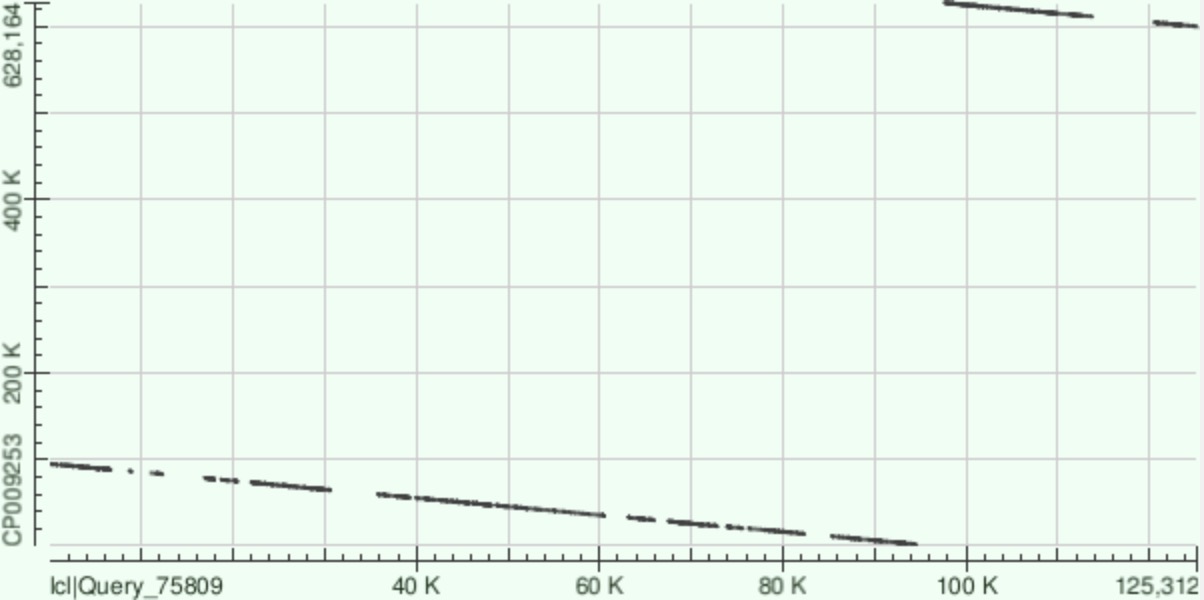
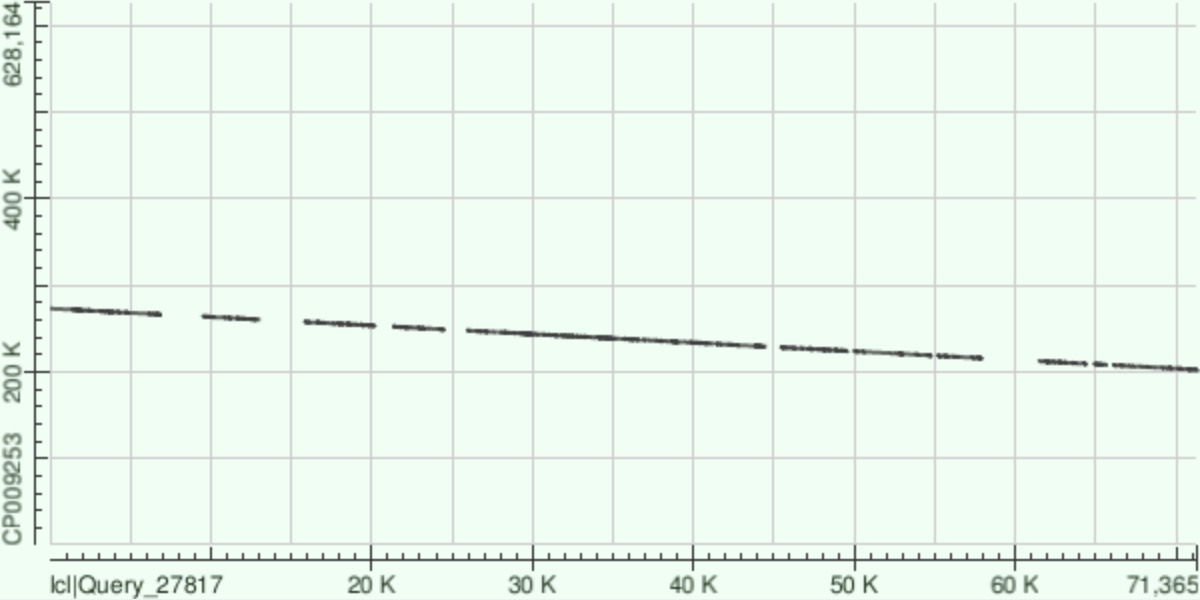
Три самых больших контига были картированы на хромосому Buchnera aphidicola (всегда по оси Y, GenBank/EMBL AC — CP009253): контиг 1 (рис. 1), контиг 11 (рис. 2) и контиг 14 (рис. 3). Под каждым дотплотом находится таблица со сводной статистикой по выравниванию (коорднаты выровненного участка, количество совпавших на нем нуклеотидов и количество гэпов).