Нуклеотидный BLAST
Задание 1. BLAST на фрагменте ДНК
Я выбрал окрестность ABC транспортера С с длиной в 1523 нуклеотида и координатами 19 909 201 – 19 918 461 на комплементарной цепи первой хромосомы. В итоге была взята окрестность 19 918 981 – 19 909 035 (длина 9 947 пар оснований, рис. 1). Полученный FASTA файл можно посмотреть по ссылке.
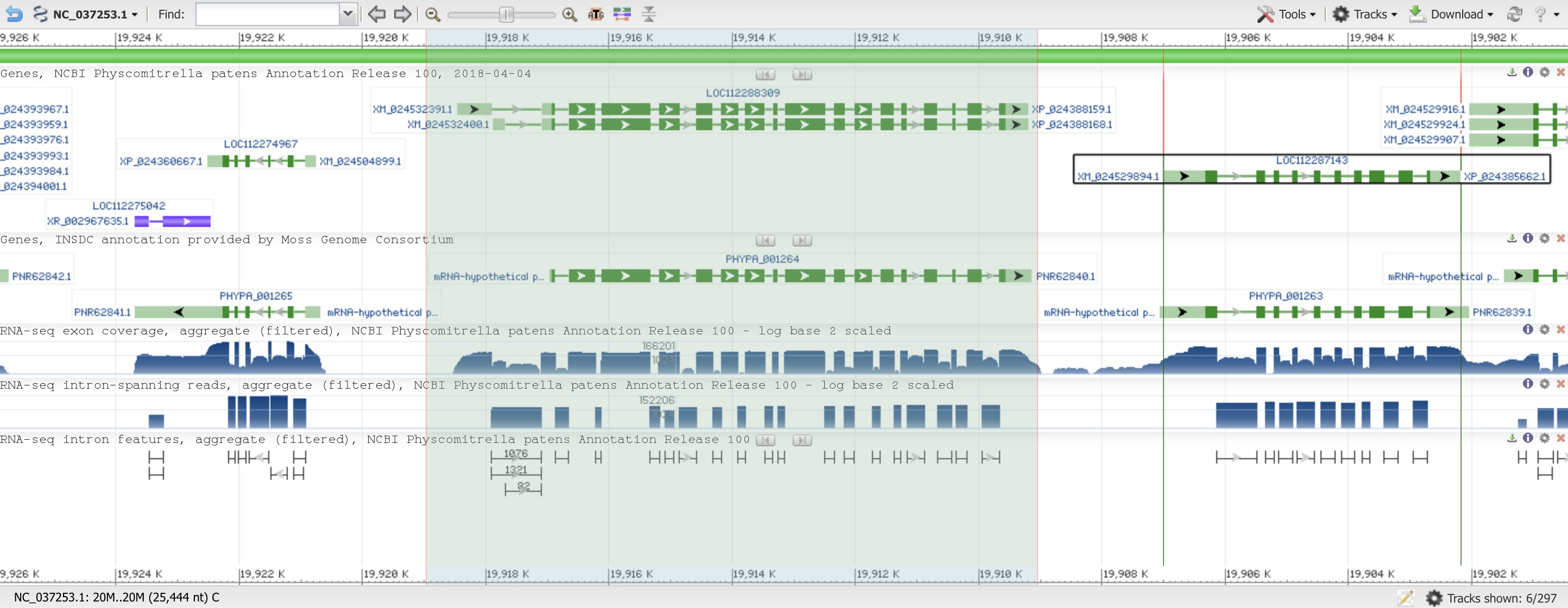
Выбранный мной организм по своей сущности мох, и с учетом этого факта я провел поиск последовательностей по выбранной мной с использованием разных алгоритмов BLAST: blastn, megablast, blastx, tblastx (табл. 1). Искал по всем семенным растениям, так как среди них довольно много собранных геномов и много просто аннотированных последовательностей ДНК.
| Алгоритм | Таксон | Длина слова | Кол-во находок | Находки | Использование алгоритма |
|---|---|---|---|---|---|
| blastn | Семенные растения | 28 | 3 | Agrostemma githago (хромосомы 8 и 18) и Sphenostylis stenocarpa | Используется для определения принадлежности к тому или иному таксону, а также для сравнения геномов не близкородственных организмов. |
| megablast | 28 | 3 | Выдача полностью совпала с выдачей blastn | Осуществляет быстрый поиск среди очень похожих последовательностей. | |
| blastx | 3 | 5000 | Поиск выдал аналогичные ABC транспортеры С у самых разных цветковых растений. Идентичность практически всех находок 45±5%. Присутствуют самые разные семейства цветковых и голосеменных. | Определяет кодирующие последовательности и ведет поиск по ним. | |
| tblastx | Я пытался запускать эту программу с самыми щадящими CPU параметрами, но все равно вылезала ошибка, связанная со слишком большим использованием CPU. При запуске на локальном компьютере получил несовместимость аргументов remote и taxidlist, а качать базу данных себе на ноутбук не хотелось :( | Может использоваться для предсказания генов в последовательностях. | |||
Задание 2. Гены рибосомальных РНК
Сначала была создана база данных на основе генома моего чудного мха. Для выполнения следующей задачи был выбран blastn, поскольку проводится поиск по нуклеотидным последовательностям, а гены рРНК как раз не являются белок-кодирующими.
Ниже указаны команды в порядке использования: makeblastdb использовалась для создания базы данных, из опций только файл на вход и указание типа базы данных.
-
makeblastdb -in GCA_000002425.2_Phypa_V3_genomic.fna -dbtype nucl
Для реализации алгоритма blastn использовалась одноименная программа. При помощи аргумента -evalue удалялись плохие находки, а -outfmt для приятного вывода в табличку. Файлы с последовательностями рРНК кишечной палочки были получены из соответсвующего файла.
-
blastn -task blastn -evalue 0.05 -query 16S_rRNA.txt -db GCA_000002425.2_Phypa_V3_genomic.fna -out blastn_16S_rRNA.txt -outfmt 7 -
blastn -task blastn -evalue 0.05 -query 23S_rRNA.txt -db GCA_000002425.2_Phypa_V3_genomic.fna -out blastn_23S_rRNA.txt -outfmt 7
16S рРНК входит в состав малой субъединицы, 23S рРНК в состав большой субъединицы прокариотической рибосомы, а прокариотические рибосомы в клетке мха могут находиться разве что в митохондриальном и пластидном геномах.
Как и ожидалось, в топе находок при сортировке по E-Value от меньшего митохондриальный (AB251495.1) и пластидный (AP005672.1) геномы.
Но стоит отметить, что в записи генома митохондриальный и хлоропластный геномы не аннотированы в принципе, да и мхи, к сожалению, не самый популярный объект для аннотации геномных сборок. Как результат, искать какие-либо закономерности в голомологии вряд ли имеет смысл.