Филогенетическая реконструкция
Построение дерева
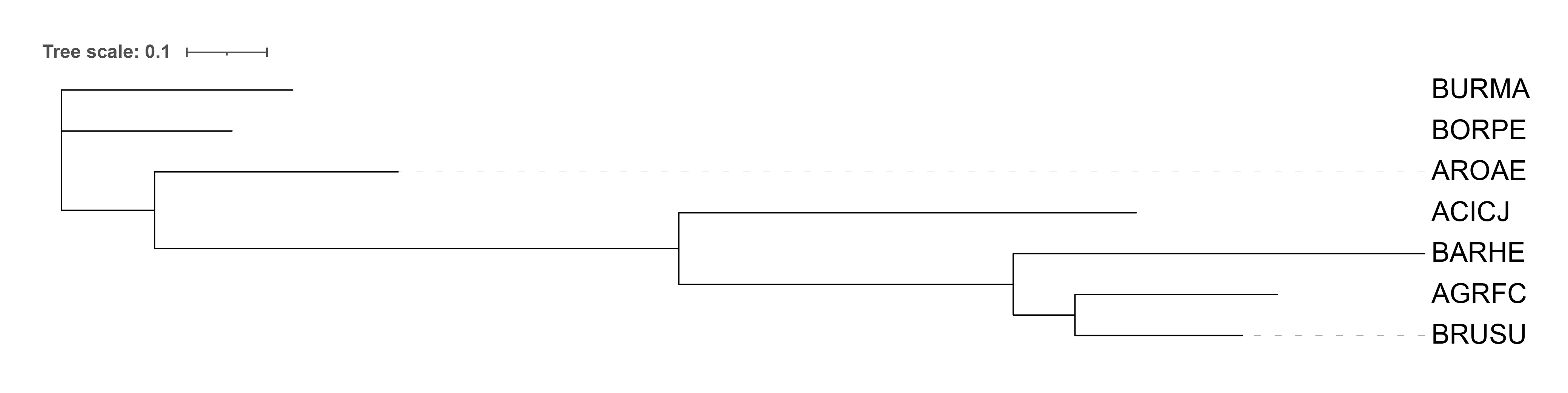
Я поступил критически лениво и выбрал первые семь видов из представленной таблички.
Ниже по ним построено древо, его скобочковая форма и нетривиальные ветви указаны под картинкой.
| Название | Мнемоника |
|---|---|
| Acidiphilium cryptum | ACICJ |
| Agrobacterium fabrum | AGRFC |
| Aromatoleum aromaticum | AROAE |
| Bartonella henselae | BARHE |
| Bordetella pertussis | BORPE |
| Brucella suis | BRUSU |
| Burkholderia mallei | BURMA |
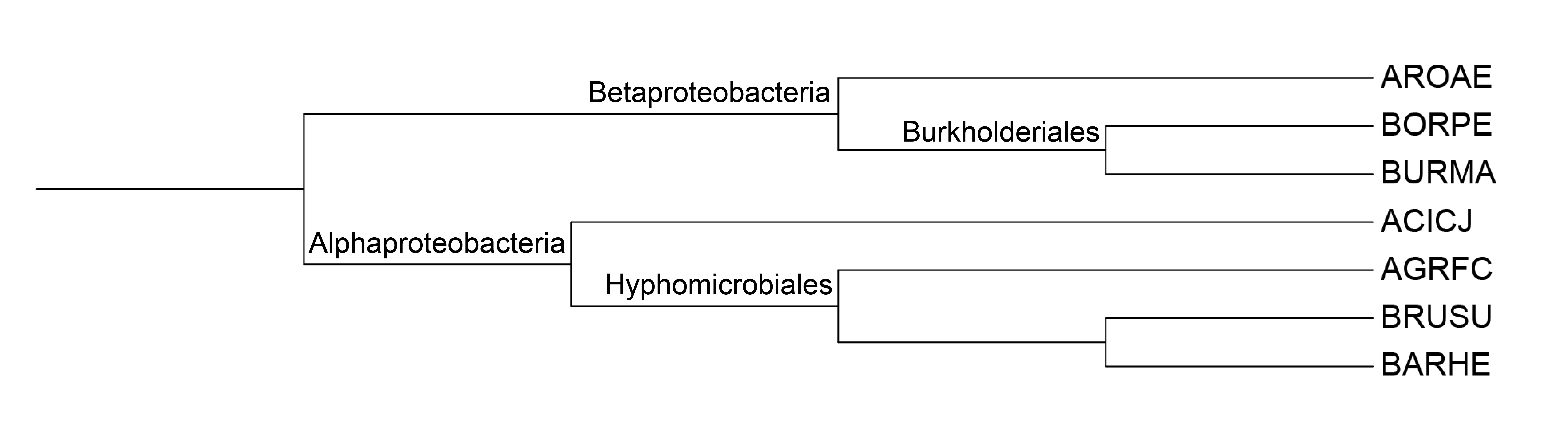
Нетривиальные ветви дерева
Я постарался выделить всевозможные нетривиальные ветви у данного дерева, перечислил их ниже.
- {BARHE, BRUSU} vs {AGRFC, ACICJ, BURMA, BORPE, AROAE}
- {BARHE, BRUSU, AGRFC} vs {ACICJ, BURMA, BORPE, AROAE}
- {BARHE, BRUSU, AGRFC, ACICJ} vs {BURMA, BORPE, AROAE}
- {BARHE, BRUSU, AGRFC, ACICJ, AROAE} vs {BURMA, BORPE}
Дерево также было дополнено названиями таксонов в соотвествии с данными из NCBI taxonomy.
- Hyphomicrobiales (BARHE, BRUSU, AGRFC)
- Alphaproteobacteria (BARHE, BRUSU, AGRFC, ACICJ)
- Burkholderiales (BURMA, BORPE)
- Betaproteobacteria (BURMA, BORPE, AROAE)
Методы филогенетической реконстукции
Не стал изменять традциям прошлого практикума и выбрал первую же мнемонику функций белков из списка — DAPE. При помощи JalView и базы данных UniProt был получен fasta-файл с последовательностями сукцинилдиаминопимелат-десукцинилазы выбранных бактерий.
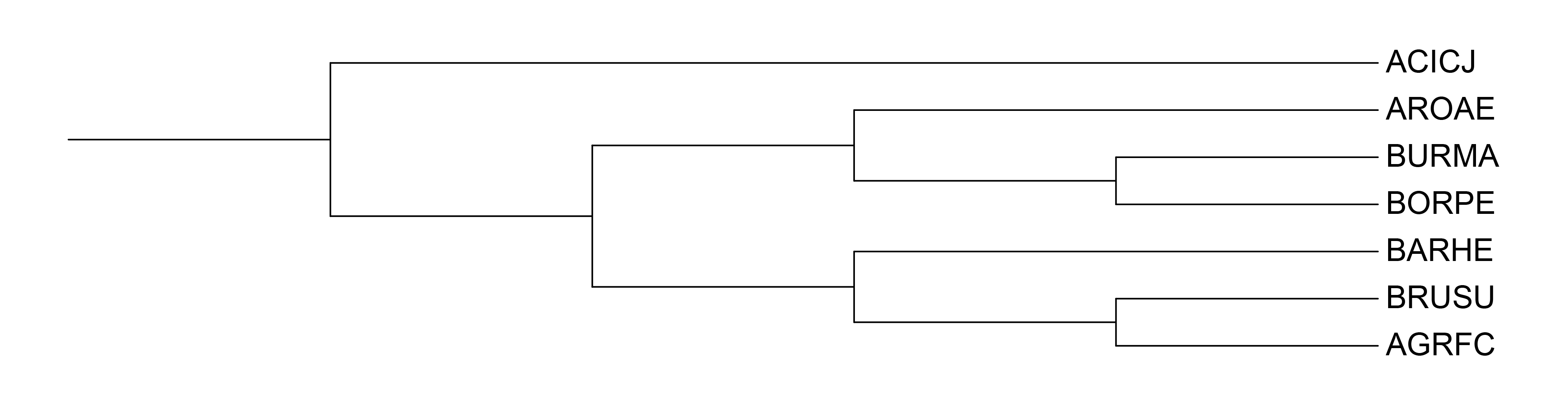
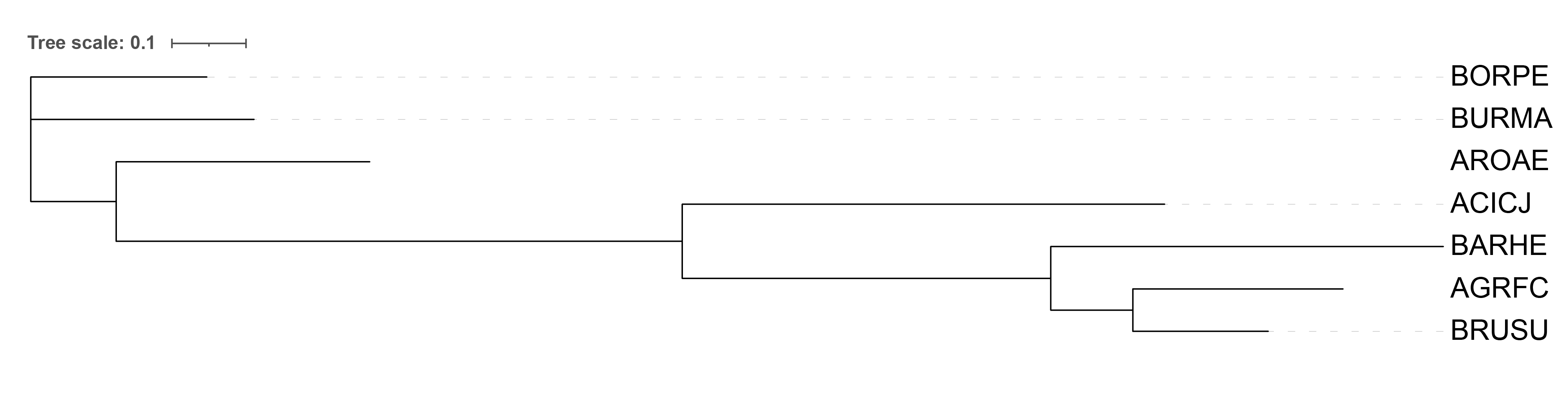
Филогенетическая реконструкция производилась на сайте NGPhylogeny.fr тремя алгоритмами: FastME (Distance-based inference of phylogenetic trees, рис. 2, 3), TNT (Phylogenetic analysis under parsimony, рис. 4, 5) и PhyML (Phylogeny software based on the maximum-likelihood, рис. 6) при выравнивании методом MAFFT. Файлы с деревьями можно найти по соответствующим ссылкам: FastME, TNT и PhyML.
Результат метода TNT отличается от двух други и все они втроем отличаются от исходного дерева в практикуме. Не могу однозачно интерпетировать этот результат без детального анализа, но выдвину несколько гипотез. Анализировать длину ветвей не берусь :(
- Возможно, выбранный белок не самый репрезентативный для данных организмов.
- Мы сравнивали аминокислотные последовательности, что не всегда наилучший выбор. Возможно, следовало бы анализировать нуклеотидные.
- Я не очень понимаю, на основании чего построено дерево в описании первого практикума, так что не могу сравнивать его с остальными. Зато про построенные мной знаю, что наилучшим (с теоретической точки зрения) является метод максимального правдоподобия, на котором основан алгоритм PhyML. И поскольку с его результатами совпала выдача алгоритма FastME, этому результату можно доверять в большей степени.
Укоренение с использованием внешней группы
Далее мы сравнивали разные варианты укоренения: укоренение в среднюю точку и при помощи внешней группы (рис. 7). Внешней группой был выбран Helicobacter pylori (HELPY). Новый файл с fasta-последовательностями доступен по ссылке, для построения дерева использовался алгоритм PhyML.
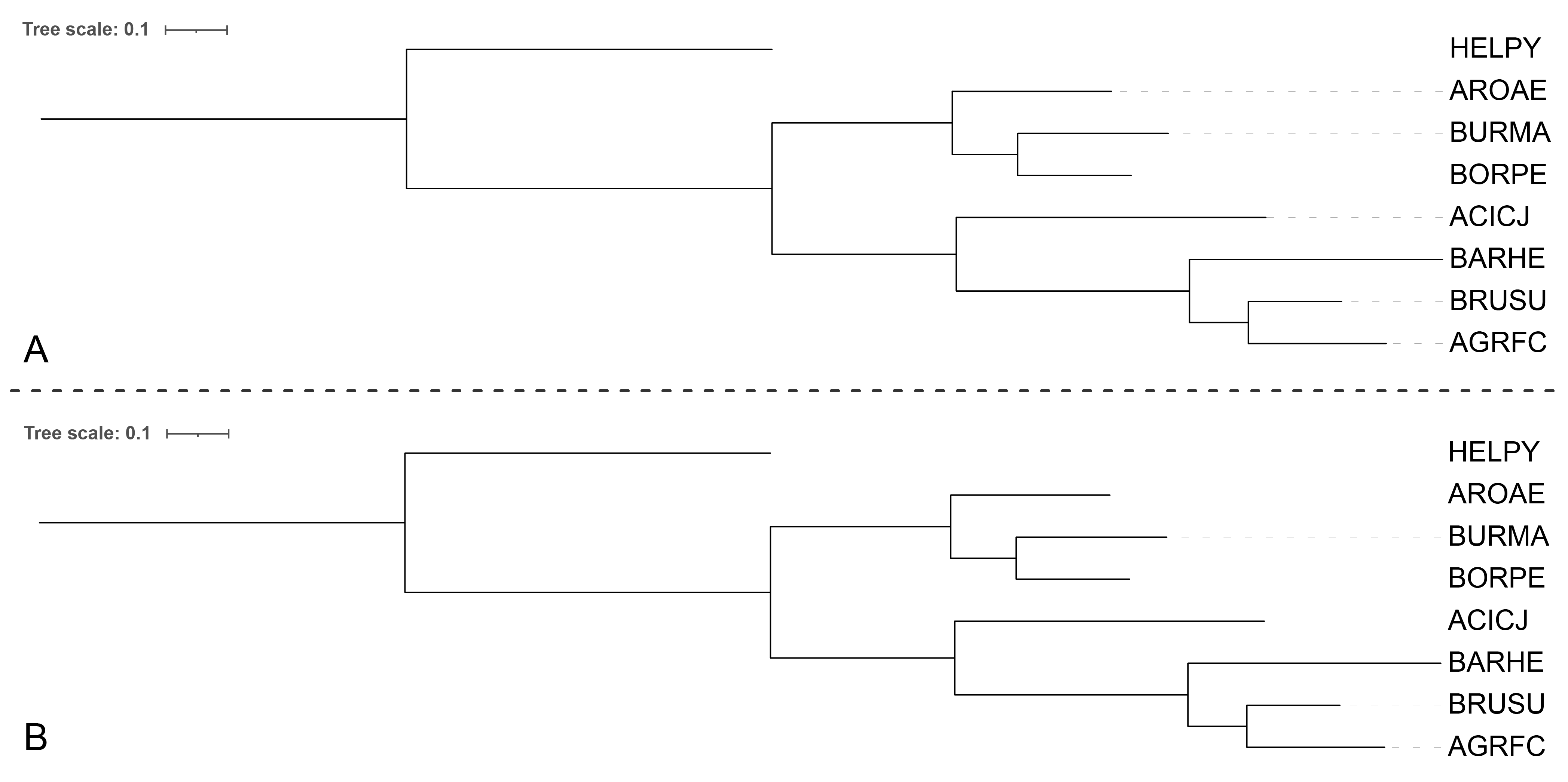
К счастью, деревья совсем не отличаются. Также они не отличаются от дерева на рис. 6 за исключением того, что на нем нет Helicobacter pylori. Но не исключено, что мне повезло, и в спорных ситуациях больше следует доверять дереву с укоренением относительно внешней группы.
Bootstrap анализ
Затем дерево было улучшено путем проведения bootstrap анализа со 100 репликами в методе FastME (рис. 8). Для этого использовался тот же набор последовательностей с Helicobacter pylori.
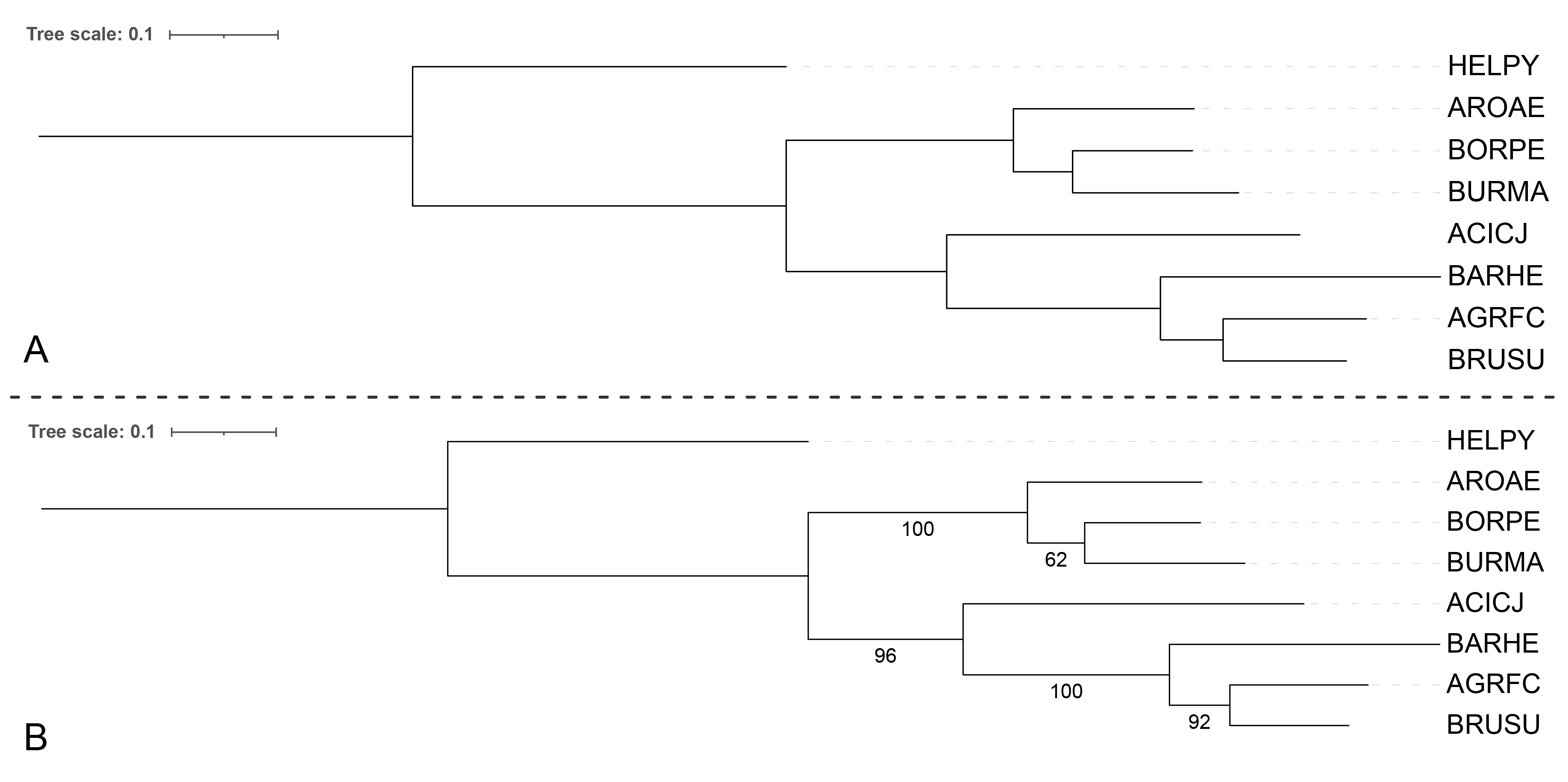
По топологии деревья не отличаются (что неудивительно, так как строились деревья одним методом с одинаковыми гиперпараметрам). Также если посчитать за правильное дерево из рис. 1, то можно подчеркнуть следующие утверждения.
- У неправильных групп поддержка bootstrap гарантированно не равна 100.
- У правильных ветвей поддержка может быть равна 100, но не всегда.
Построение дерева по нуклеотидным последовательностям
Ранее уже упоминалось, что построенное дерево может быть неправильным из-за того, что мы строили его именно по аминокислотным последовательностям. Чтобы исправить это, было решено построить дерево по 16S rRNA выбранных бактерий (включая Helicobacter pylori). Это дерево так же не факт, что отражает правду, для полной коллекции можно было бы построить еще одно по какой-нибудь некодирующей ДНК последовательности (например, ITS), но задание этого не требует.
Последовательности были взяты из базы данных NCBI и сохранены в fasta-файл. Дерево по ним строилось при помощи FastME с выравнением методом MAFFT, также проводился bootstrap анализ с 1000 реплик (рис. 9).
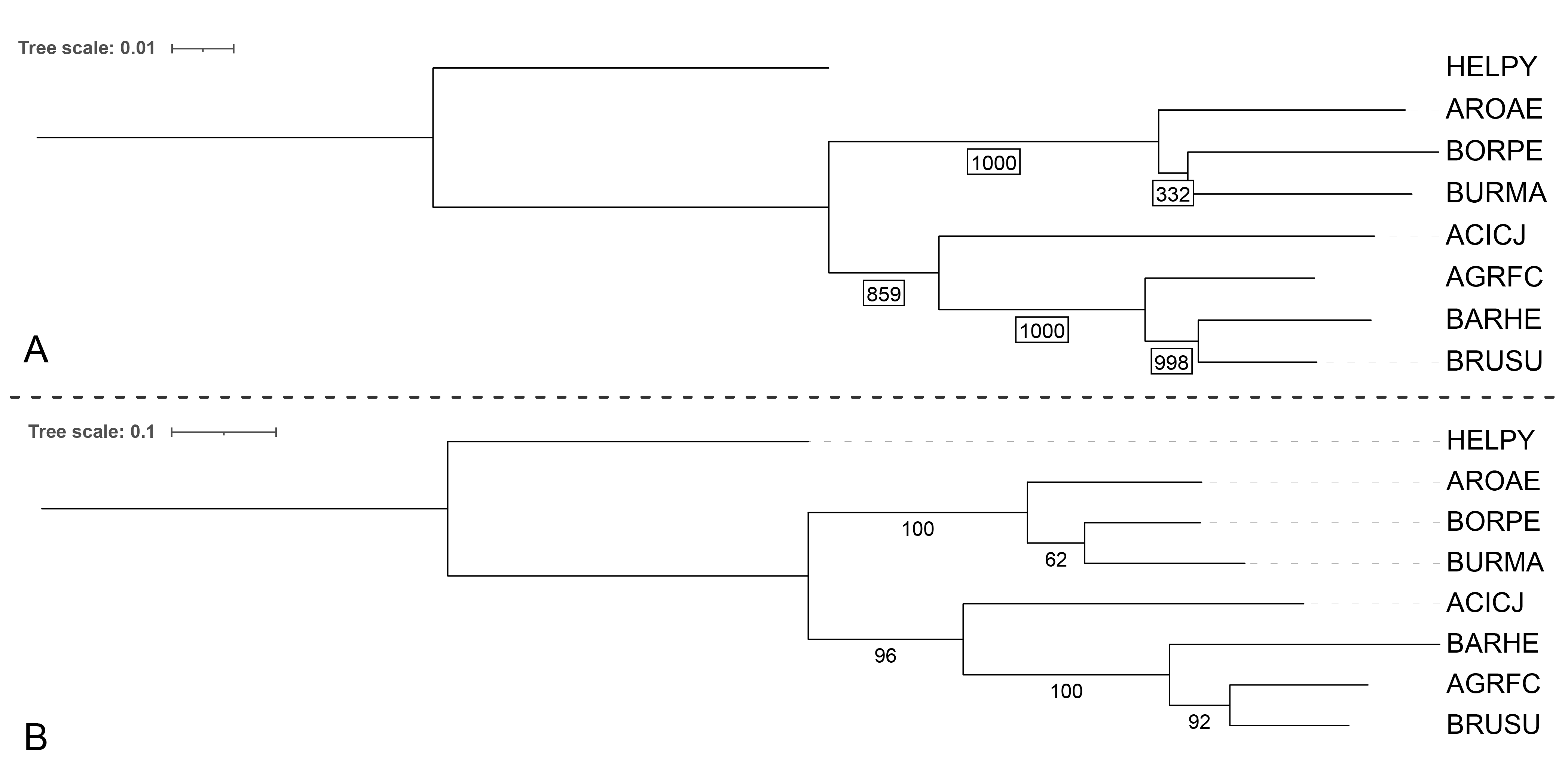
Как и ожидалось, это дерево не просто оказалось самым близким к правдивому (рис. 1), но и в принципе совпало с ним! Хотя стоит отметить, что поддержка bootstrap некоторых ветвей не самая высокая