Изучение ортологичных и паралогичных белков
Составление списка гомологичных белков, включающих паралоги
Поиск гомологов белка CLPX_ECOLI осуществлялся при помощи указанных ниже команд. Файлы с последовательности взяты из диреткории /P/y22/term4/Proteomes. Результат можно посмотреть по ссылке.
cat ACICJ.fasta AGRFC.fasta AROAE.fasta BARHE.fasta BORPE.fasta BRUSU.fasta BURMA.fasta > ~/term4/pr4/proteomes.fasta
makeblastdb -dbtype prot -in proteomes.fasta -out prot
blastp -query clpx_ecoli.fasta -num_threads 4 -db prot -evalue 0.001 -out blast_results.txt
Реконструкция и визуализация
После выравнивания последовательностей методом MAFFT в программе NGPhylogeny.fr методом FastMe строилось филогенетическое дерево со следующими параметрами.
- Gamma distributed rates across sites — NO
- Starting tree — BIONJ
- No refinement
- 100 bootstrap реплик
Остальные параметры были оставлены по умолчанию. Результирующее дерево представлено на рис. 1 (его Newick формула доступна по ссылке).
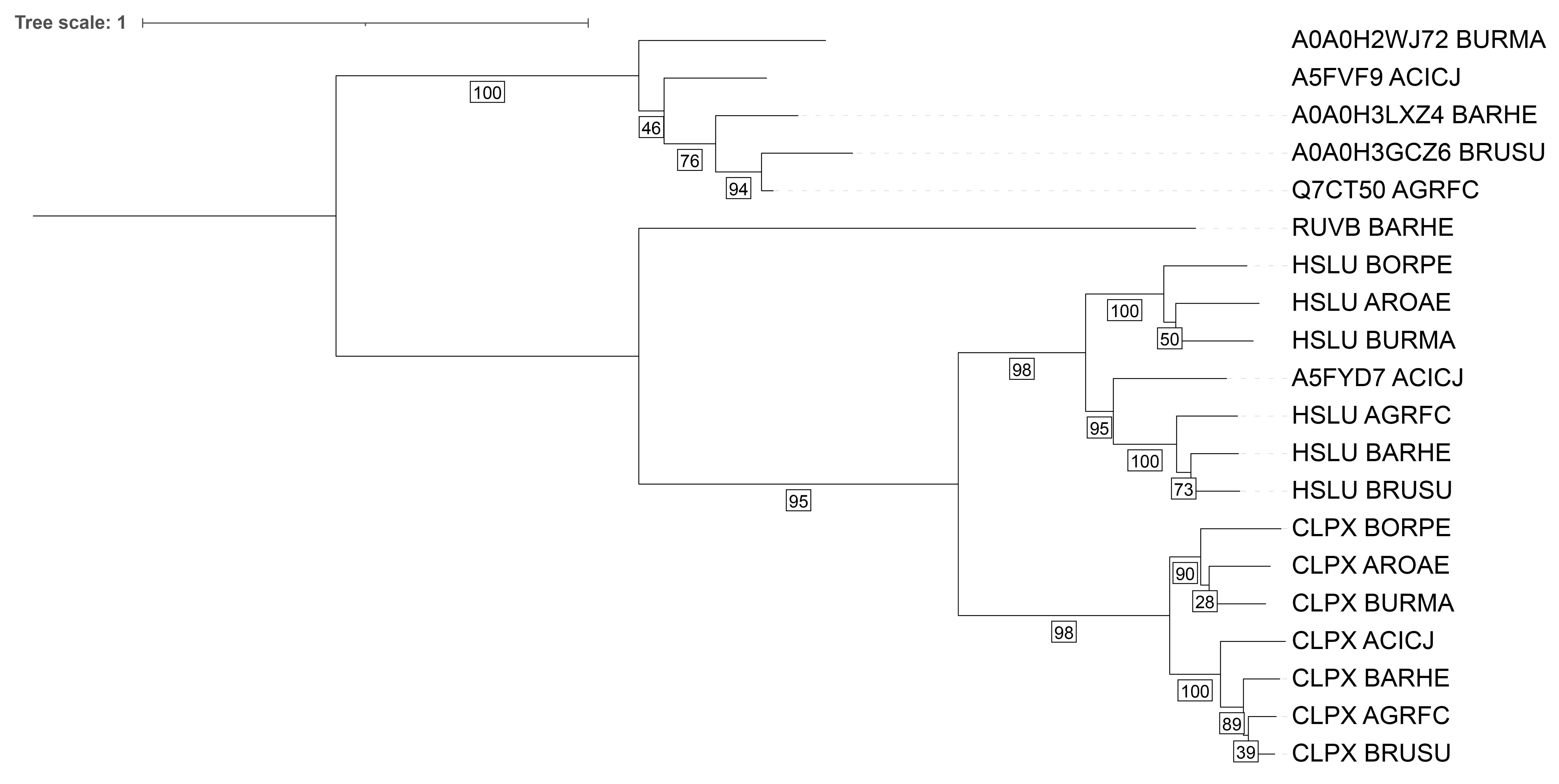
На дереве были найдены пары ортологов и паралогов (табл. 1), а сами клады были покрашены в соответствии с ортологичными группами (каждая имеет поддержку bootstrap не менее 98, рис. 2).
| Тип | № | Белки |
|---|---|---|
| Ортологи | I | HSLU AROAE и HSLU BURMA |
| II | HSLU BARHE и HSLU BRUSU | |
| III | CLPX AGRFC и CLPX BRUSU | |
| Паралоги | I | HSLU BURMA и CLPX BURMA |
| II | RUVB BARHE и HSLU BARHE | |
| III | A0A0H3GCZ6 BRUSU и HSLU BRUSU |
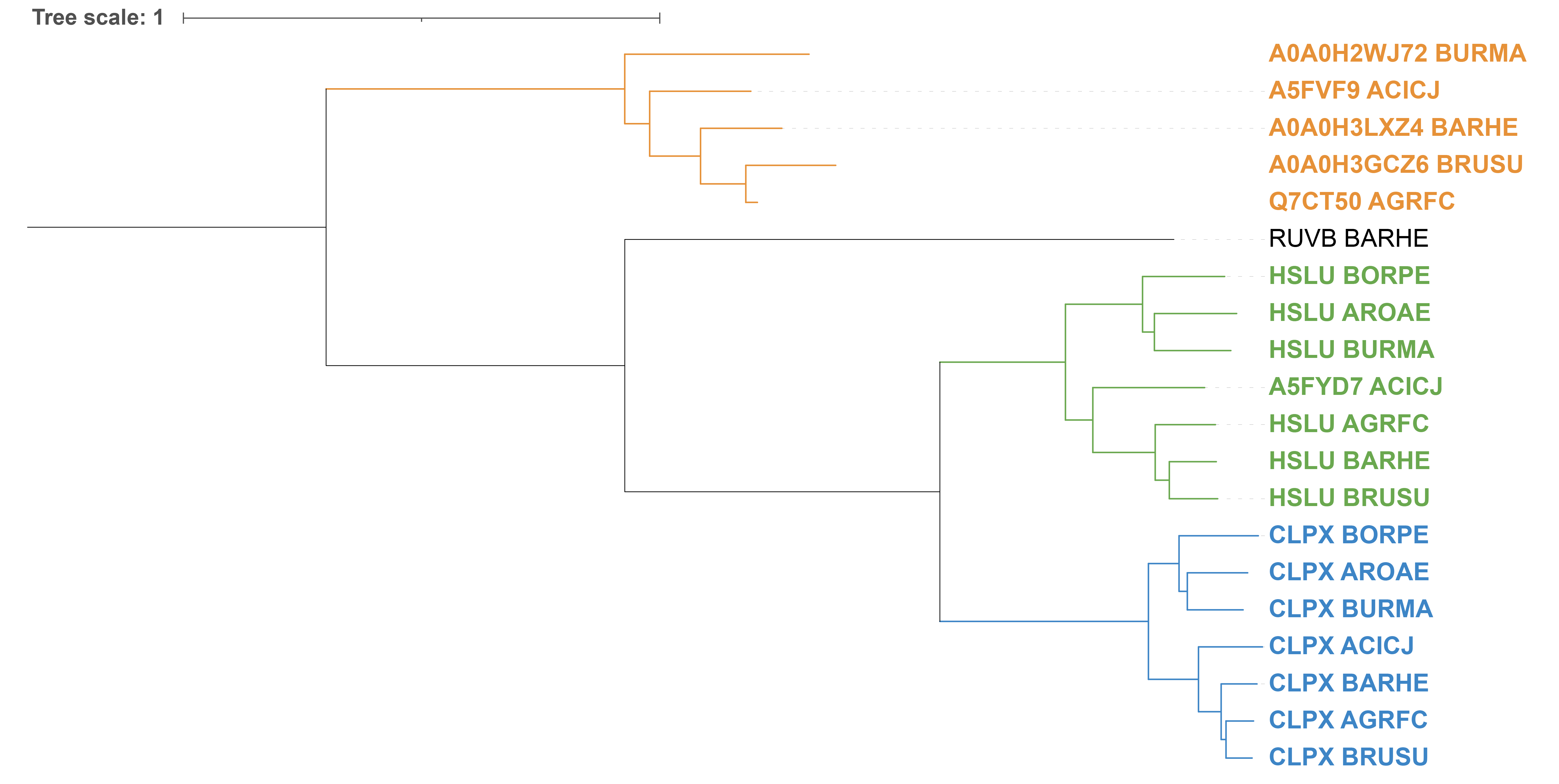
Также было решено построить дерево, на котором группы ортологичных белков были бы «схлопнуты» (рис. 3). Таким образом получится филогенетическое древо групп ортологичных белков. Классификация внутри этих групп примерно соответствует истинной (с точностью до перестановок внутри клады размером в три вида).
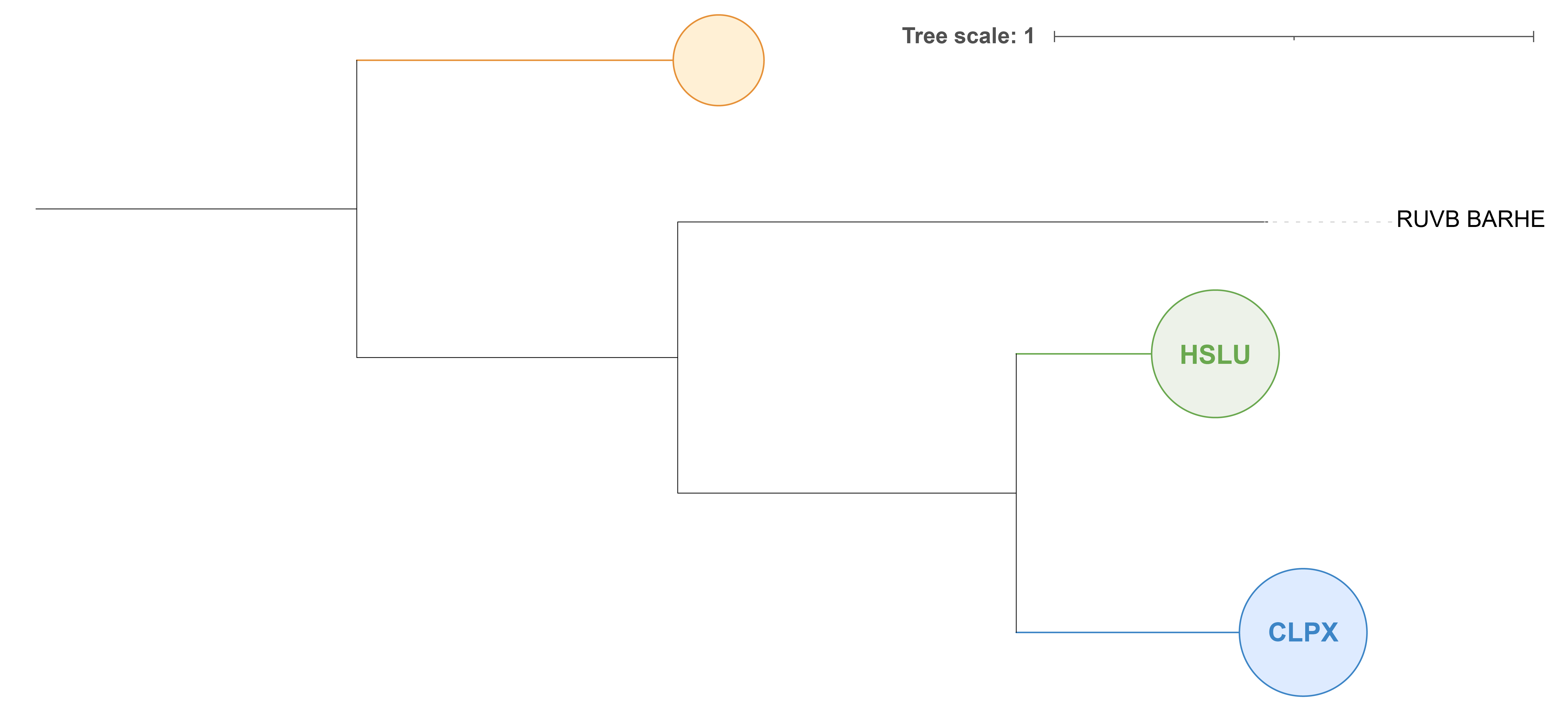
На рис. 3 можно заметить три основные группы белков, из которых только у двух можно выделить общие названия и функции: это HSLU и CLPX, на дереве они найдены для всех выбранных бактерий.
- HSLU — это АТФазный домен протеасомо-подобного белка. Активность домена аналогична шаперонной: Связывание АТФ и его последующий гидролиз HslU необходимы для разворачивания белков, впоследствии гидролизуемых HslV. HslU распознает N-концевую часть этих белков и разворачивает их, прежде чем они будут направлены в HslV для гидролиза.
- CLPX — это тоже АТФазный домен протеасомо-подобного комплекса (Clp протеазы) и его тоже сравнивают с шапперонами по функционалу.
Третья группа белков не имеет одного названия, так что я решил изучить их функционал по-отдельности. Это оказались АТФ-зависимые цинковые металлопептидазы для цитоплазматических и мембранных белков, так что их функционал схож с HSLU и CLPX — все три группы являются АТФ-зависимыми и входят в состав тех или иных пептидаз.