Трансмембранные белки
Сравнение предсказаний трансмембранных участков в бета-листовом белке
Я нашел бесподобный белок иммунной системы человека — мембраноатакующий комплекс (Membrane attack complex, PDB: 6H04; единого UniProtID нет, у белка много доменов, расскажу про них ниже: CO5_HUMAN, CO6_HUMAN, CO7_HUMAN, CO8A_HUMAN, CO8B_HUMAN, CO8G_HUMAN, CO9_HUMAN).
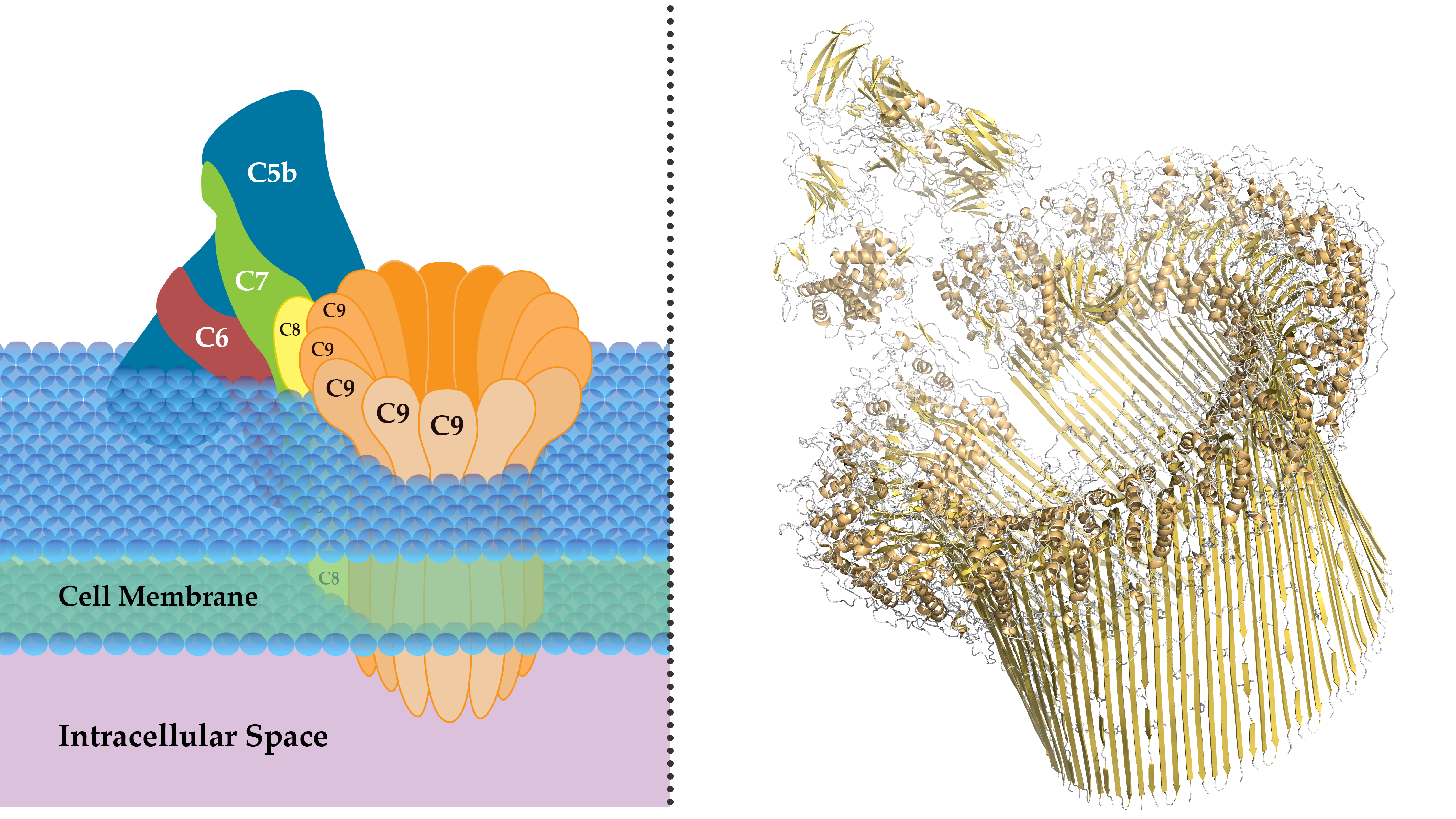
Почему он крут? Потому что он входит в систему комплемента человека (отдел иммунной системы, сам белок локазлизован в цитоплазматичексой мембране) и способен формировать поры в клетках-мишенях, что приводит к их лизису и быстрой гибели. Он состоит из комплекса четыре комплементарных белка (C5b, C6, C7 и C8, образуют «горб» на основной структуре) и множества копий белка С9, который и образует эту мега-пору. Ссылки на эти домены и приведены в списке из UniProt ID. Список трансмембранных доменов приведен в таблице ниже (табл. 1).
| Tilt | TM Segment 1 | TM Segment 2 | TM Segment 3 | TM Segment 4 | |
|---|---|---|---|---|---|
| B | 54 | 406-409 | - | - | - |
| C | 17 | 202-207 | 210-214 | 329-336 | 339-344 |
| F | 14 | 222-228 | 231-235 | 350-356 | 359-363 |
| G | 10 | 217-224 | 244-250 | 363-370 | 374-380 |
| H | 15 | 214-222 | 245-251 | 362-369 | 374-381 |
| I | 18 | 214-221 | 245-252 | 361-369 | 374-381 |
| J | 20 | 214-221 | 245-252 | 361-369 | 374-381 |
| K | 22 | 214-221 | 245-252 | 361-369 | 374-381 |
| L | 23 | 214-221 | 245-252 | 361-369 | 374-381 |
| M | 25 | 214-222 | 245-252 | 361-370 | 374-381 |
| N | 24 | 214-222 | 244-251 | 362-370 | 374-380 |
| O | 25 | 215-223 | 244-251 | 362-370 | 374-380 |
| P | 12 | 216-223 | 244-251 | 362-370 | 374-380 |
| Q | 24 | 215-223 | 244-251 | 362-370 | 374-380 |
| R | 23 | 215-223 | 244-251 | 362-370 | 374-380 |
| S | 21 | 215-223 | 244-250 | 362-370 | 374-380 |
| T | 20 | 215-223 | 244-251 | 362-370 | 374-380 |
| U | 18 | 215-223 | 244-251 | 362-370 | 374-380 |
| V | 17 | 215-222 | 245-251 | 362-370 | 374-380 |
| W | 16 | 214-221 | 245-252 | 361-369 | 374-381 |
| X | 17 | 214-221 | 245-252 | 361-368 | 374-381 |
Далее со страницы PDB этого белка была получена его последовательность, которую я подал на вход DeepTMHMM. Грустно, конечно, но согласно предсказаниям, у этого белка нет доменов внутри мембраны, о чем однозначно говорит вывод программы. Что поделать :( Но это можно объяснить тем, что β-листы образуют пору довольно большого диаметра, так что программа, очевидно, посчитала весь белок находящимся вне мембраны. К тому же, в закреплении в мембране совсем не участвуют α-спирали.
Видимо, поскольку DeepTMHMM ничего не нашел, графическую визуализацию результатов получить не удалось. Но ничего, в следующем задании точно получится!
Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Мне достался белок MDTH_BLOFL, последовательность которого была получена из UniProt (и доступна по ссылке). Это Р-шликопротеин, или белок множественной лекарственной устойчивости (Multidrug resistance protein) из семейства ABC-переносчиков. Этот белок входит в состав АТФ-зависимых мембранных транспортеров, который во многом неспецифично выкачивает лекарственные препараты из клетки. Конкретно для этого белка показано, что он отлично выкачивает противоопухолевые препараты из раковых клеток, из-за чего его ингибиторы используются в противоопухолевой терапии.
Со страницы в UniProt была получена модель белка, предсказанная AlphaFold, и подана на вход PPM 3.0. N-конец (и С-конец) белка расположены в цитоплазме (Topology (N-ter) — in, если верить структуре и описанию в Wikipedia). Сам белок расположен во внутренней мембране Грам-отрицательной бактерии (Gram-negative bacteria inner membrane, как указано на странице в UniProt), поэтому количество мембран указано как одна. Предсказание программы в формате PDB доступно по ссылке (рис. 2). На том же рис. 2 показаны предсказания DeepTMHMM.
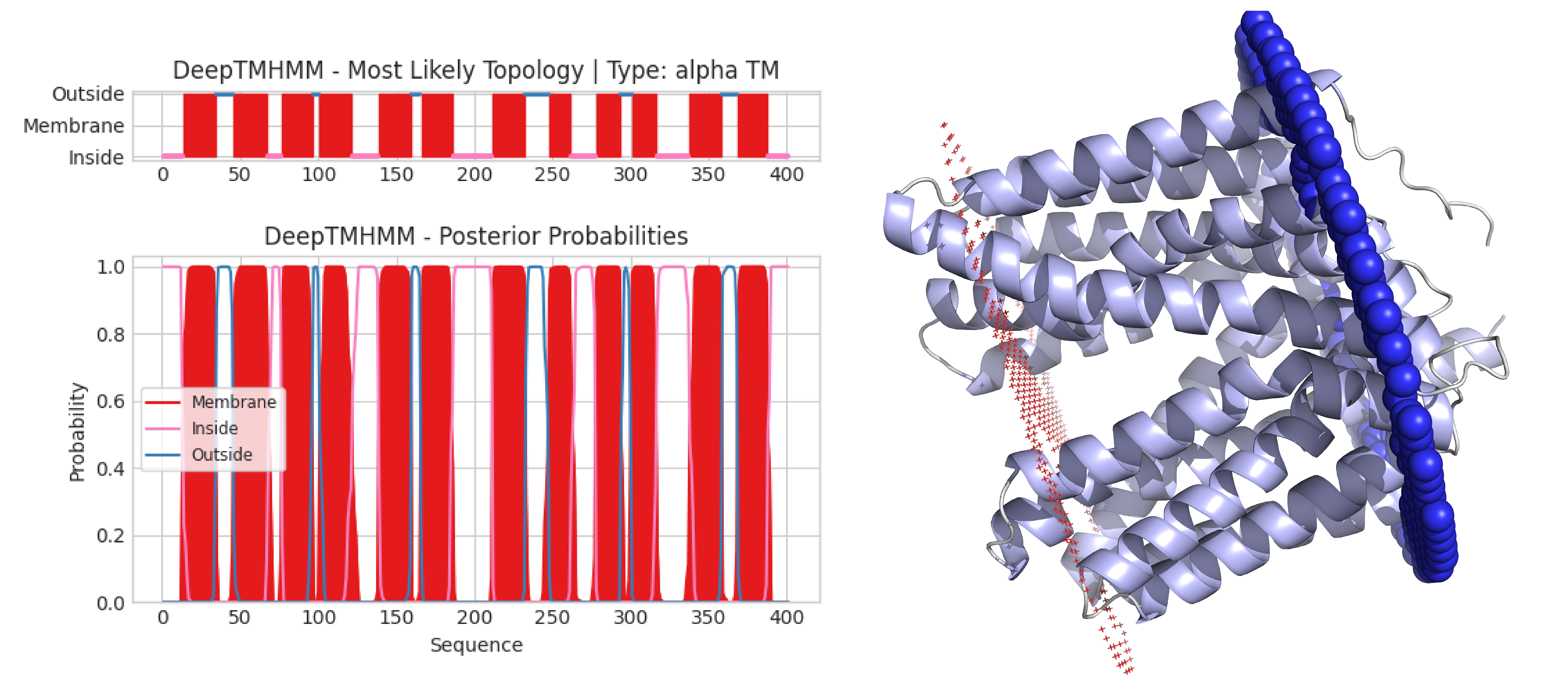
Текстовая выдача DeepTMHMM доступна по ссылке, но вкратце: найдено 12 трансмембранных α-спиралей (как и в PPM 3.0). Текстовая выдача PPM 3.0 представлена в табл. 2. Координаты α-спиралей, предсказанных двумя моделями, совпали с точностью до 2-3 аминокислот, что очень радует. Более того, предсказаниям PPM 3.0 можно верить, опираясь на практически 100% вероятность предсказания трансмембранных доменов.
| Субъединица | Tilt | Участки |
|---|---|---|
| Трансмембранные аминокислотные остатки | ||
| A | 24 | 12-34, 37, 47-70, 73, 75-96, 98-122, 124, 133-159, 162-184, 198, 202, 205-206, 209-233, 246-269, 271-295, 298-324, 333-358, 366, 368-389 |
| Трансмембранные сегменты со вторичной структурой | ||
| A | 0 | 1(12-34), 2(47-70), 3(76-95), 4(100-121), 5(134-159), 6(164-184), 7(211-233), 8(246-269), 9(274-294), 10(299-323), 11(333-358), 12(368-389) |