Базы данных Reactome, Human Protein Atlas и Human String
Выданный мне список белков было решено анализировать двумя самыми красивыми (на мой взгляд) базами данных: Reactome и Human Protein Atlas. Позже мне стало ну уж очень интересно посмотреть на граф из Human String, так что я изучил и эту базу данных тоже.
Примечание. Все картинки специально сохранены в высоком разрешении (300 dpi), так что при необходимости вы можете кликать на них, и они будут открываться на весь экран. Я старался проверять, чтобы текст везде хорошо читался.
База данных Reactome
У нее обалденный user-friendly интерфейс! Для начала работы мне было достаточно просто нажать кнопку Analysis и скопировать в нее свой список белков. Буквально через минуту я получил красивую картинку с палочками (рис. 1). Самое приятное, что эту выдачу можно было скачать в формате SVG и подгрузить к себе на сайт в высоком разрешении.
Но будем объективны: в мире куда популярнее фигуры, а не линии! Даже на постерах мы чаще не обводим текст, а выделяем его цветными блоками. База данных Reactome позволяет сделать то же самое, перейдя в базу данных Reacfoam (рис. 2). К сожалению, тут экспорт в SVG не поддерживается, так что придется довольствоваться старыми-добрыми далеко не 300 dpi.
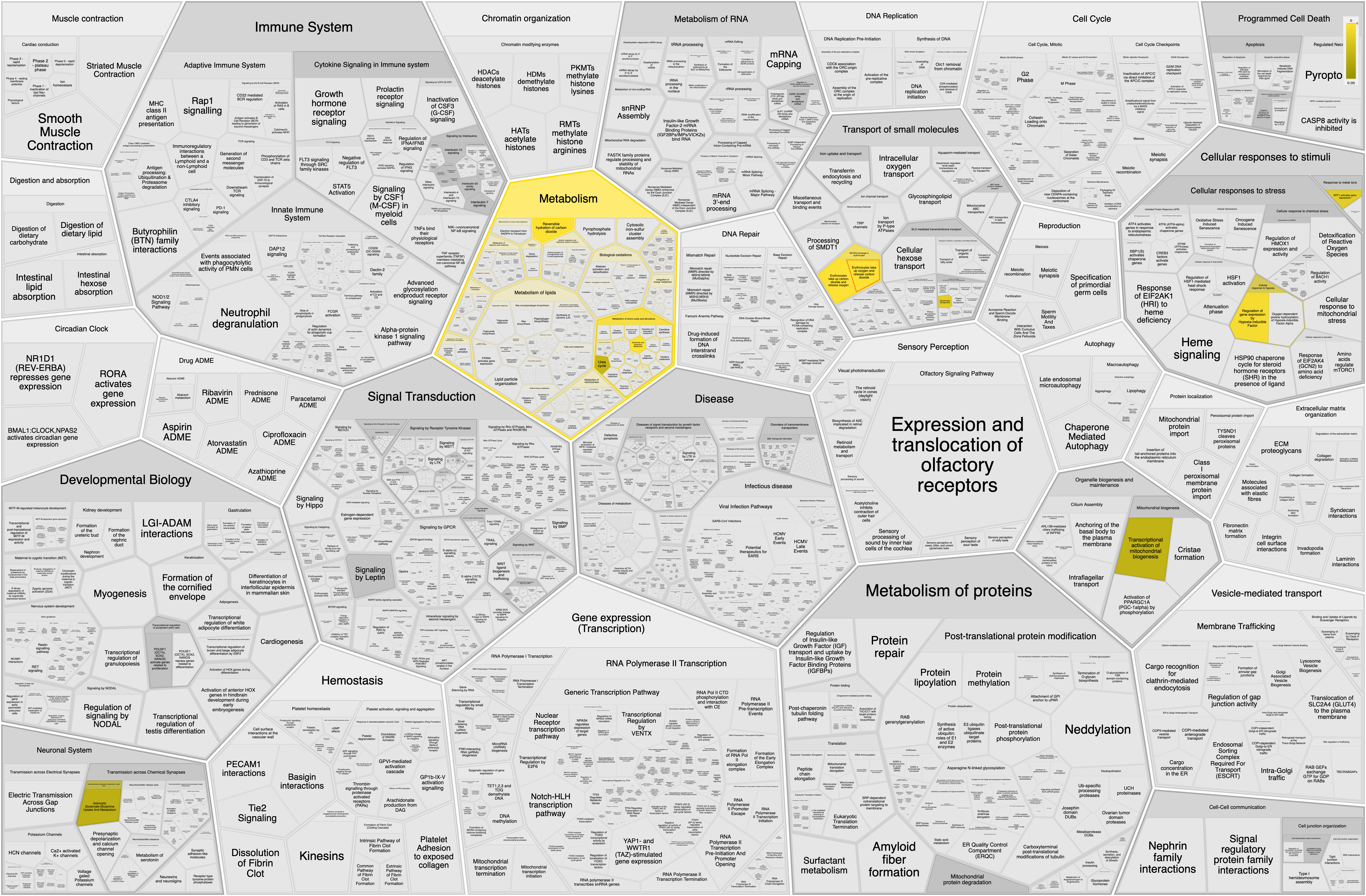
Тут уже гораздо легче увидеть, что мои белки поселились в нескольких группах (рис. 3-7). Но для удобства я экспортировал все данные в CSV формат, так с ним взаимодействовать гораздо легче.
Но как я узнаю позже, такая выдача не самая простая и информативная, поскольку не позволила узнать главного: среди белков всего две группы, карбоангидразы и связанные с метаболизмом глутамата. Но эта выдача по своему информативна, ведь позволяет хорошо ориентироваться в метаболических путях, с которыми связаны выбранные мной белки.
База данных Human Protein Atlas
К сожалению, я не нашел способа загрузить все белки сразу, поэтому пришлось по одному вбивать их в поисковую строку. Не исключаю, что был способ взаимодействия через командную строку, но мне хотелось познакомиться и с графическим интерфейсом программы. Начнем с базового анализа на примере первого белка: CA13. Выдача для него показана на рис. 8.
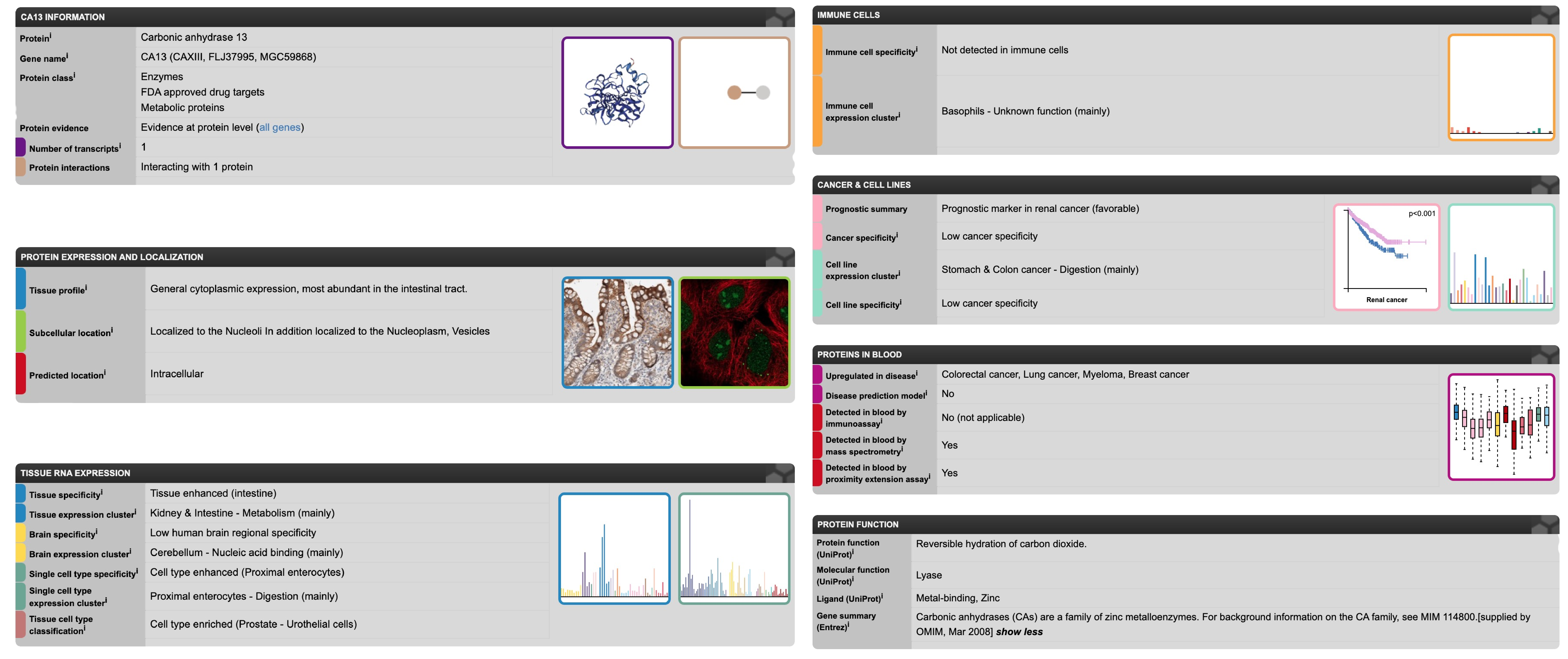
В Summary о выбранном белке сказано, что это карбоангидраза 13 — это уже упомянутый выше белок, который катализирует гидролиз СО2 до HCO3- и закодирован в гене CA13 (CAXIII, FLJ37995, MGC59868). Наиболее высокая экспрессия у человека, внезапно, в кишечнике, а сам белок локализуется в ядрышке, нуклеоплазме и реже в везикулах (рис. 9).
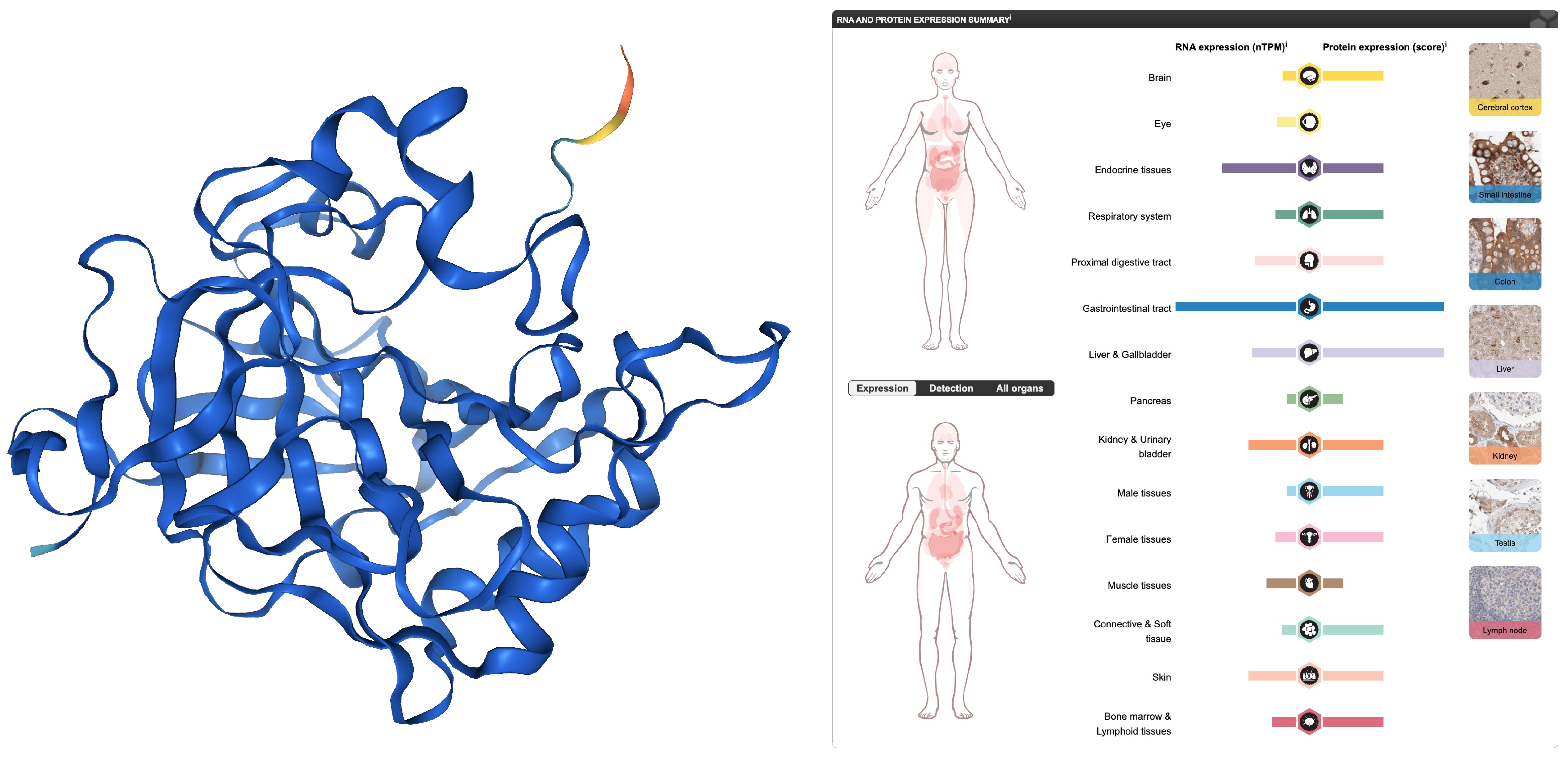
Из других выдач я узнал, что этот белок может быть ассоциирован с колоректальным раком, раком легких и молочной железы, миеломой, о чем в том числе свидетельствуют диаграммы на рис. 8.
В общем, база данных мне понравилась: картинки и правда очень красивые и информативные: можно узнать и о локализации в клетках и тканях человека, заодно почитать про ассоциированность с различными заболеваниями и все это подкреплено диаграммами.
База данных Human String
Весь интерес к этой базе данных оправдан графом, который нам также разрекламировали на лекции. Отмечу, что в этой базе данных очень удобный импорт (легко загрузил сразу весь список белков), но без указания организма (я указал Homo sapiens) граф строиться ну никак не хотел.
На выходе я получил граф (отмечу возможность скачать его в SVG, пусть и комп хотел забанить этот SVG за битость и вирусы), представленный на рис. 10.
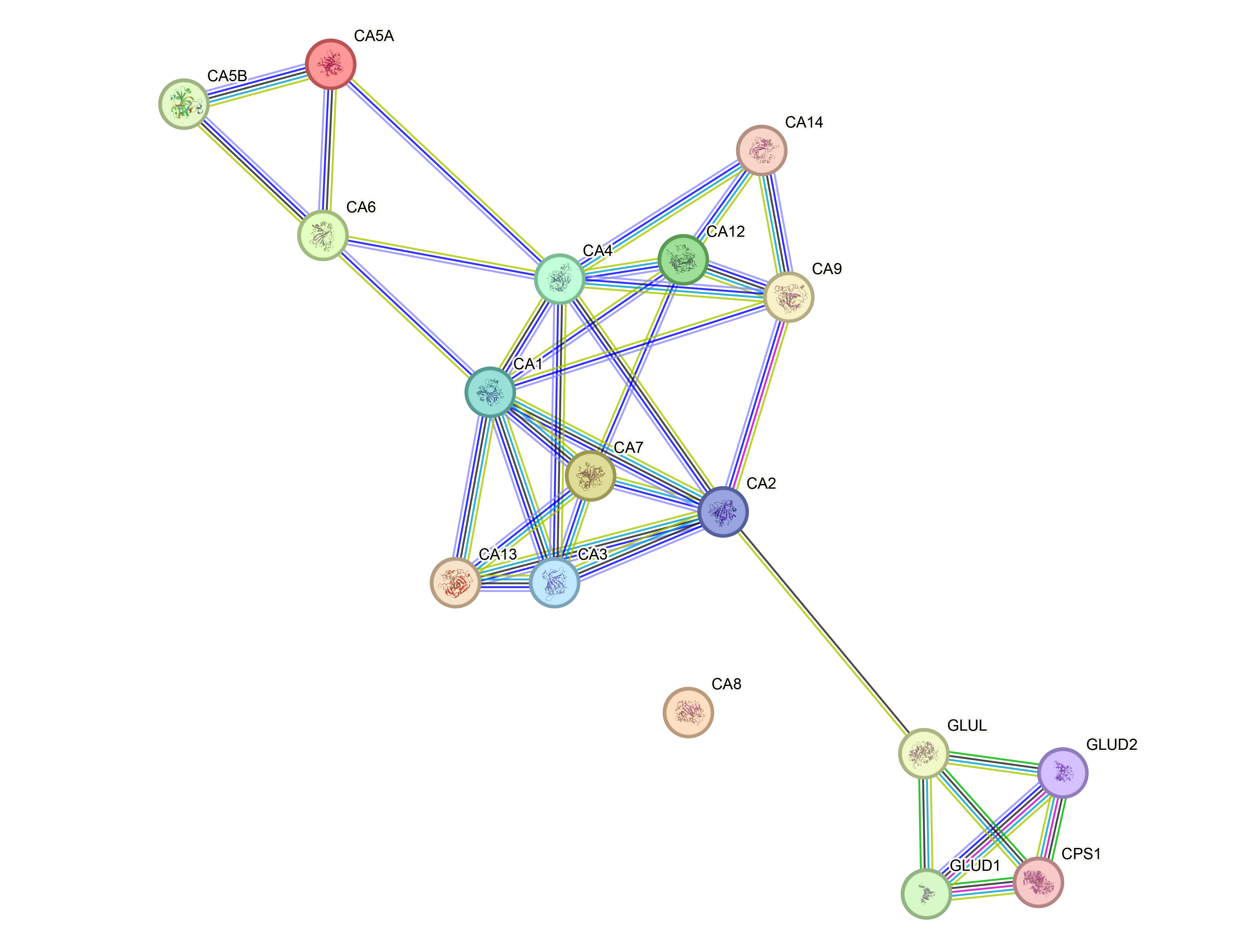
Классно! Белки кластеризовались в соответствии с выдачей базы данных Reactome: работающие с углекислым газом и кислородом в одну группу (сверху и в центре), работабщие с глутаматом и глутамином в другую (снизу). Смущает один нюанс — карбоангидраза 8 (CA8) ни с кем не кластеризовалась, что грустно и странно. Выдача базы данных не позволяет изучить этот вопрос, поэтому из интереса я залез в Human Protein Atlas.
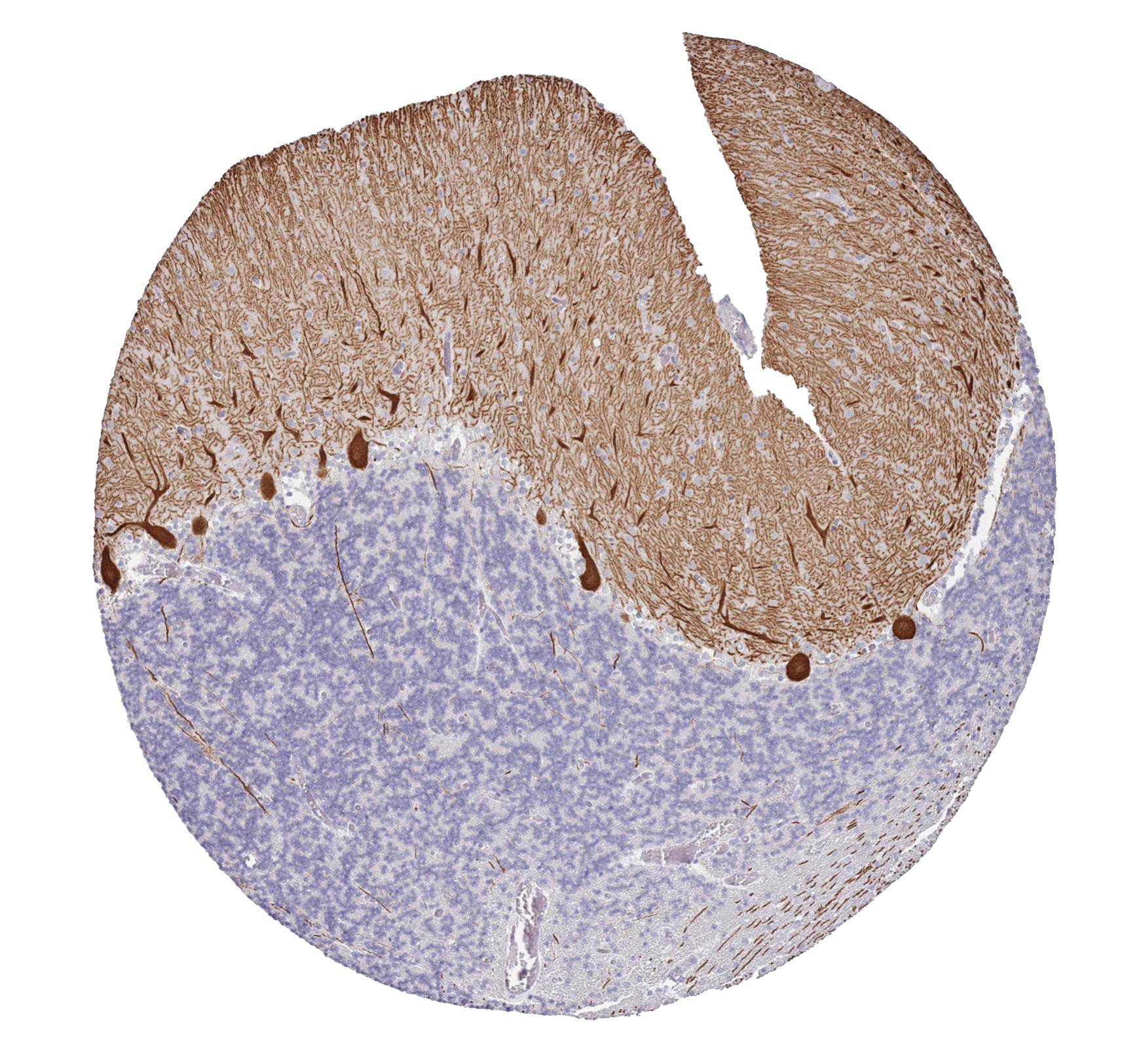
Оказалось, что это карбоангдираза клеток Пуркинье головного мозга и при этом она ассоциирована с раком поджелудочной железы! Большая часть других карбоангидраз расположены в кишечнике (реже в костном мозге, печени и слюнных железах).
Красоты ради слева приведена фотография тех самых клеток Пуркинье в формате стикера в telegram (но взят он не из telegram, а из Humam protein Atlas).

.jpg)
.jpg)
.jpg)
.jpg)
.jpg)
.webp)
.png)