Сигналы в геноме
HIV ribosomal frameshift signal
HIV ribosomal frameshift signal (Сигнал рибосомного сдвига рамки считывания ВИЧ) — это рибосомный сдвиг вируса иммунодефицита человека (ВИЧ), используемый для трансляции двух разных белков с одной молекулы РНК. Об этом свидетельствует описание сигнала из Rfam (HIV_FE, RF00480) и наличие у него двух рамок считывания (ORF, рис. 1).
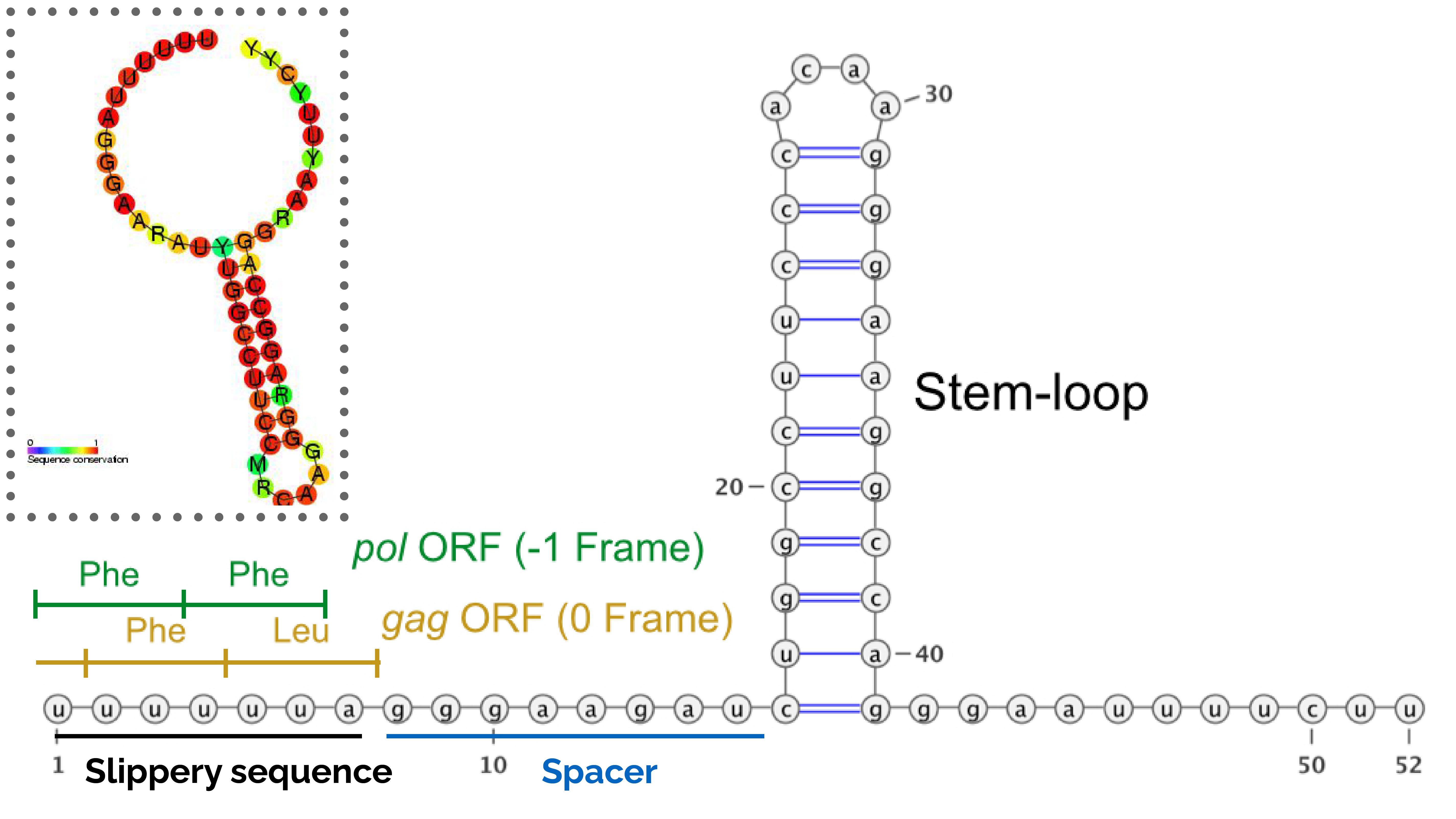
Сайт сдвига рамки считывания состоит из трех основных частей (Dinman et al., 2002; Biswas et al., 2004).
- Скользкая последовательность — семинуклеотидный участок (5' U UUU UUA 3'). Называн скользким, потому что процесс сдвига рамки считается довольно вероятным, даже если внизу не образуется петля. По данным Mouzakis et al. (2013), сдвиг происходит примерно с частотой от 0.0001% до 0.1% на каждый кодон, даже без вторичной структуры РНК.
- Спейсер — восьминуклеотидный участок, разделяющий скользкую последовательность и регуляторную петлю. Показано, что он также важен для запрограммированного сдвига рамки считывания рибосомы, поскольку делеции в области спейсера снижают стабильность нижерасположенной петли, тем самым влияя на способность сигнала вызывать сдвиг рамки на один нуклеотид назад (Mouzakis et al., 2013).
- Петля — считается, что это именно петля, но в работах некоторых авторов она описана как псевдоузловая структура и внутримолекулярный РНК-триплекс (Biswas et al., 2004; Mouzakis et al., 2013). Независимо от этого предположительная петля приводит к тому, что рибосома застревает на скользком участке.
Сдвиг рамки считывания рибосомы (programmed ribosomal frameshifting, PRF) происходит примерно в 5% случаев. Это сдвиг на один нуклеотид назад, приводящий к синтезу полипротеина Gag-Pol. Снижение эффективности сдвига может замедлять репликацию вируса. Соотношение основного продукта, Gag, к Gag-Pol оценивается примерно как 20 к 1 (Kobayashi et al., 2013).
Построение PWM для последовательности Козак Homo sapiens
Последовательность Козак в геноме человека — это сигнал старта транскрипции ДНК. Для построения ее позиционной весовой матрицы (PWM) использовался Python-скрипт, на вход которому я подал аннотированную таблицу таблицу генов человека. Код был написан на основе написанного Варварой Сафоновой в 2023 году в ходе аналогичного практикума. Матрица строилась по участкам из 7 нуклеотидов до старт-кодона и 3 нуклеотидов после него. На выходе программа создает три файла.
- Файл со 100 последовательностями для обучения.
- Файл c 300 последовательностями для теста.
- Файл с 300 случайными последовательностями аналогичной длины для негативного контроля.
В результате скрипт сгенерировал представленную ниже (табл. 1) и в файле позиционную весовую матрицу.
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0.494 | -1.187 | -0.389 | -0.34 | 0.353 | -0.017 | -0.745 | 1.218 | -5.691 | -5.691 | -0.44 | -0.126 | -0.612 |
| C | 0.347 | 0.347 | 0.414 | 0.506 | -0.623 | 0.668 | 0.911 | -5.327 | -5.327 | -5.327 | -0.187 | 0.693 | 0.275 |
| G | 0.445 | 0.851 | 0.275 | 0.347 | 0.535 | -0.248 | 0.071 | -5.327 | -5.327 | 1.582 | 0.851 | -0.076 | 0.59 |
| T | -0.34 | -0.745 | -0.293 | -0.612 | -0.899 | -0.676 | -0.819 | -5.691 | 1.218 | -5.691 | -0.612 | -0.745 | -0.389 |
Также было изучено распределение весов последовательностей в обучающей выборке, тестовой выборке и негативном контроле (рис. 2).
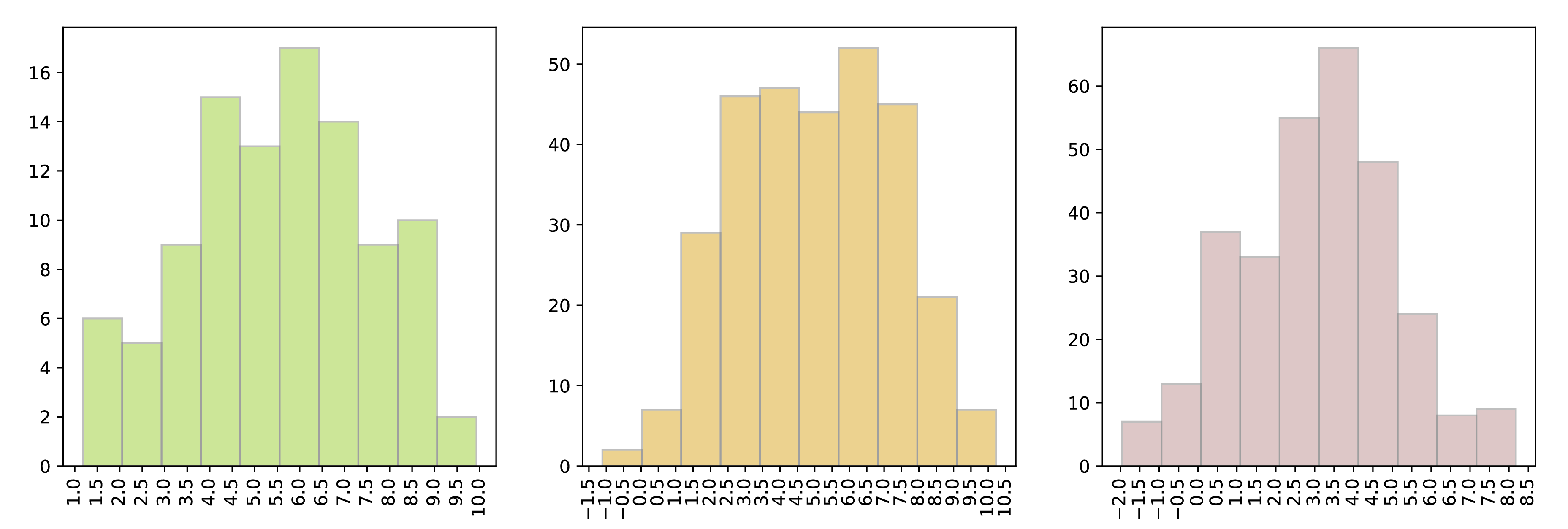
На основании диаграмм выше (рис. 2) определим пороговое значение как 4.0, так как после него число последовательностей выходит на относительное плато и дальше быстро достигает пика. Ниже приведена таблица проверки находок (табл. 2), прошедших это пороговое значение.
| Обучение | Положительный контроль | Отрицательный контроль | |
|---|---|---|---|
| Cигнал (+) | 71 (71%) | 200 (67%) | 91 (30%) |
| Cигнал (-) | 29 (29%) | 100 (33%) | 209 (70%) |
Для анализа информационного содержания (IC) этой последовательности построена матрица информационного содержания (табл. 3), для этого использовался соответсвующий Python-скрипт (на основе скрипта Варвары Сафоновой). Результат также представлен по ссылке.
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0.128 | -0.154 | -0.112 | -0.103 | 0.214 | -0.007 | -0.151 | 1.761 | 0.0 | 0.0 | -0.121 | -0.047 | -0.141 |
| C | 0.145 | 0.145 | 0.185 | 0.248 | -0.099 | 0.386 | 0.671 | 0.0 | 0.0 | 0.0 | -0.046 | 0.41 | 0.107 |
| G | 0.206 | 0.589 | 0.107 | 0.145 | 0.27 | -0.057 | 0.022 | 0.0 | 0.0 | 2.286 | 0.589 | -0.021 | 0.315 |
| T | -0.103 | -0.151 | -0.093 | -0.141 | -0.156 | -0.146 | -0.154 | 0.0 | 1.761 | 0.0 | -0.141 | -0.151 | -0.112 |
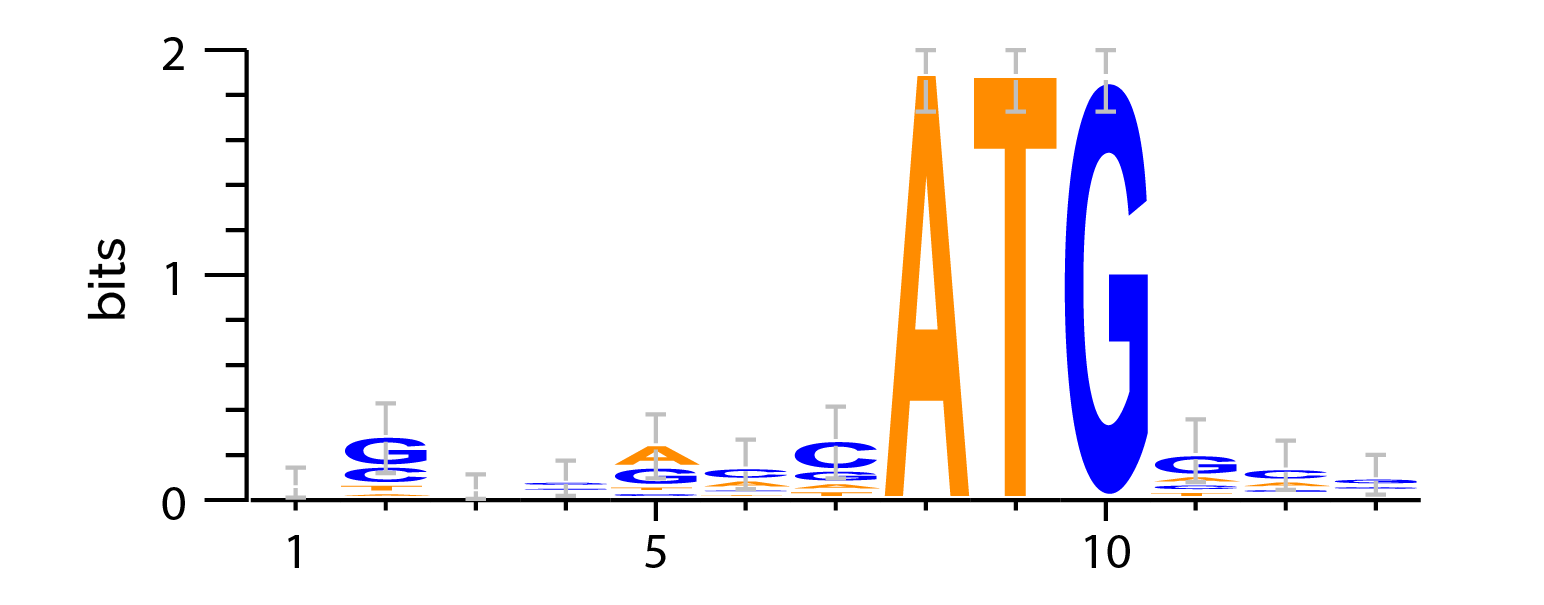
Также обучающую выборку подали на вход сервису WebLogo 3 и получили Logo (рис. 3). Из него видно, что позиции 2, 4, 5 и 7-12 (включающие старт-кодон на 8-10) имеют значимый информационный вес. На его основе у нас есть основания считать, что выравненные последовательности обладают специфической функцией.
Источники
Biswas P., Jiang X., et al. (2004). The human immunodeficiency virus type 1 ribosomal frameshifting site is an invariant sequence determinant and an important target for antiviral therapy. Journal of Virology. 78(4): 2082–2087.
Dinman J.D., Richter S., et al. (2002). The frameshift signal of HIV-1 involves a potential intramolecular triplex RNA structure. Proceedings of the National Academy of Sciences of the United States of America. 99(8): 5331–5336.
Kobayashi Y., Zhuang J., Peltz S., et al. (2010). Identification of a cellular factor that modulates HIV-1 programmed ribosomal frameshifting. The Journal of Biological Chemistry. 285(26): 19776–19784.
Mouzakis K.D., Lang A.L., Vander Meulen K.A., et al. (2013). HIV-1 frameshift efficiency is primarily determined by the stability of base pairs positioned at the mRNA entrance channel of the ribosome. Nucleic Acids Research. 41(3): 1901–1913.