Построение PWM с помощью MEME
Я взял бактерию Methylophaga nitratireducenticrescens, обзор которой у меня был в первом семестре. Для нее был скачан геном и feature table из NCBI.
Затем я воспользовался скриптом, который предоставил мне Владислав Мурзин, за что выражаю ему большую благодарность. Далее вырезал участки с -40 по -1 от начала CDS, так как у бактерии присутствуют -35 и -10 участки для посадки сигма-субъединицы, поэтому ожидается, что промотор будет в этом регионе. Из полученного файла я отобрал случайные 30 последовательностей и перевел файл в fasta-формат. После этих процедур был запущен MEME со стандартными параметрами.
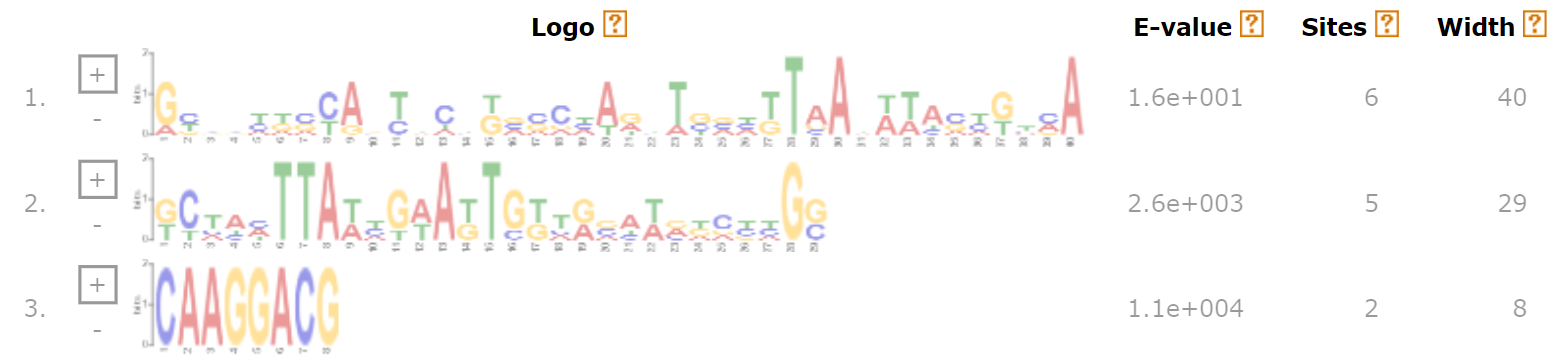
Выдачу MEME можно посмотреть здесь. Мотив GYNNYKSYANYNCNKVSCHARNTSVHKTAANWWASYKTMA (e-value 1.6e+001, PSSM), мотив KCTAHTTAWHGWAKTGKTRVAWVDCBHGS (e-value 2.6e+003, PSSM), мотив 3 CAAGGACG (e-value 1.1e+004, PSSM).
Проверка PWM с помощью FIMO
Для проверки через FIMO я загрузил все полученные ранее участки -40 и -1, а затем полный геном бактерии. P-value был установлен по умолчанию равным 0.0001.
Для первого мотива:
825 находок во всей хромосоме, 6 в промоторах.
Для второго мотива:
684 находок во всей хромосоме, 13 в промоторах.
Для третьго мотива:
29 находок во всей хромосоме, 3 в промоторах. Если нормировать на количество нуклеотидов, то вероятность нахождения сигнала в промоторе выше, чем во всей хромосомы.
Результаты
Таким образом, для первых двух мотивов было показано, что вероятность нахождения такого сигнала была сильно меньше чем по всей хромосоме, тогда как для 3 мотива она была больше. Это косвенно может говорить о его важности в промоторе. Малое количество находок может быть обусловлено тем, что этот мотив является консервативным для малого количества генов, которые регулируются определенным типом сигма-субъединицы (коих достаточно много).