Blastp
Мои бактерии:
| Corynebacterium diphtheriae | CORDI |
| Corynebacterium efficiens | COREF |
| Bifidobacterium longum | BIFLO |
| Mycolicibacterium vanbaalenii | MYCVP |
| Rhodococcus jostii | RHOJR |
| Streptomyces avermitilis | STRAW |
| Thermobifida fusca | THEFY |
Сперва я получил список гомологов и затем отобрал нужные с e-value < 0.001. Список находок приведен ниже. Содержит как находки из SwissProt (sp), так и из TrEMBL (tr). В дальнейшем буду использовавать находки SwissProt. Список находок.
Как видно из списка, самым низким e-value обладают АТФ-зависимые Clp протеазы. Среди находок велико количество иных протеаз. Также можно заметить, что все белки являются АТФазами.
Далее я составил из списка с AC идентификаторами всех белков файл с последовательностями, который можно посмотреть по ссылке. Используемый скрипт.
Построение дерева
Дерево я решил построить методом UPGMA в программе Mega X. Для этого я использовал вышеназванный FASTA-файл и построил выравнивание с помощью Muscle.
Скобочная формула полученного дерева представлена здесь.
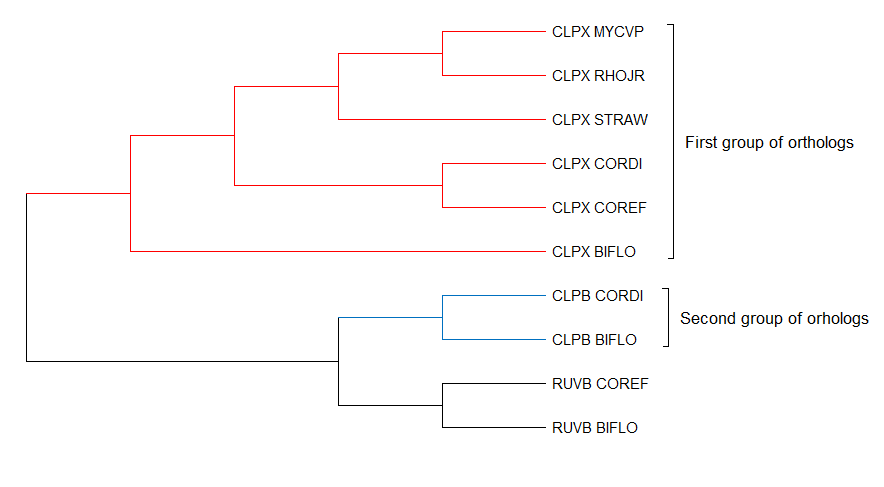
В качестве примера три пары ортологов:
- CLPX MYCVP и CLPX RHOJR
- RUVB COREF и RUVB BIFLO
- CLPB CORDI и CLPX COREF
Три пары паралогов:
- RUVB BIFLO и CLPB BIFLO
- CLPX CORDI и CLPB CORDI
- CLPX COREF и RUVB COREF
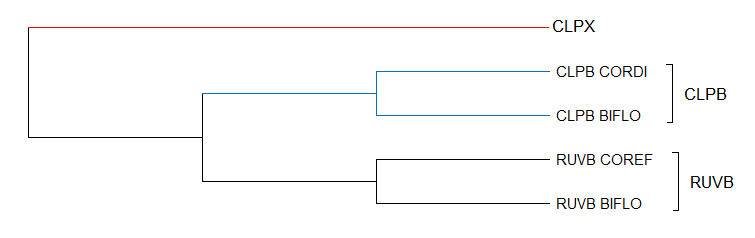
Основная часть дерева представлена АТФ-связывающими субъединицами протеазы ClpX. Туда вошли 6 различных бактерий, но дерево не показывает точную реконструкцию эволюционных взаимоотношений (в частности, STRAW ближе к BIFLO, нежели к RHOJR и MYCVP). Не вошла бактерия Thermobifida fusca.
Две остальные группы были представлены двумя гомологами белков бактерий. Первая группа шапероном ClpB (CORDI и BIFLO), а вторая субъединицей RuvB ДНК-хеликазы (COREF и BIFLO).