Подготовка чтений
| Для анализа использовались команды: | fastqc chr10.fastq
fastqc chr10_out.fastq |
| Для удаления концов с качеством ниже 20: | java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr10.fastq chr10_out1.fastq TRAILING:20 |
| Для удаления чтений длиной меньше 50: | java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr10_out1.fastq chr10_out.fastq MINLEN:50 |
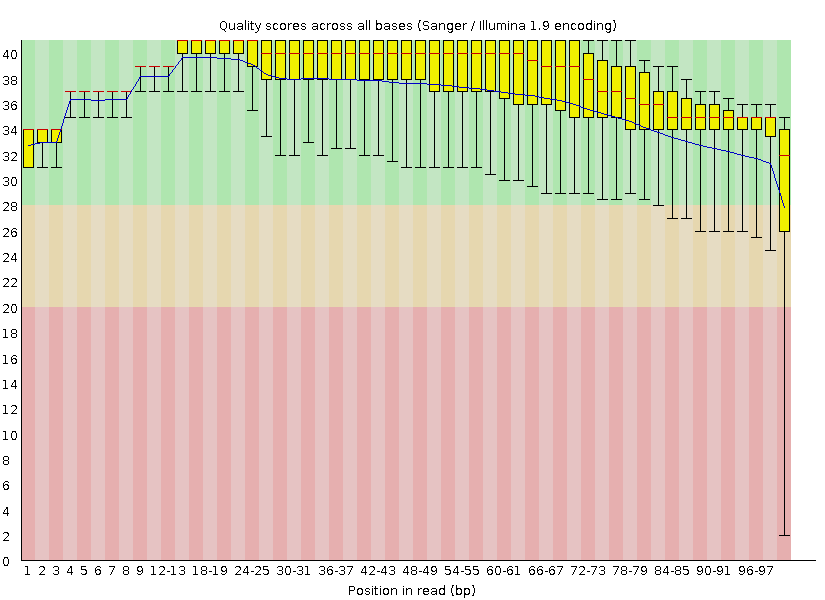
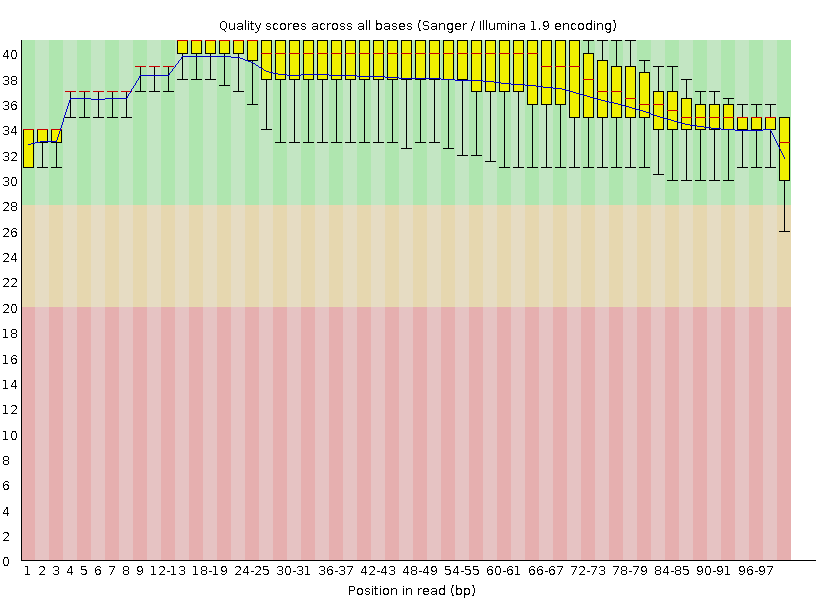
Колличество ридов упало с 10666 до 10526. Как видно, общее качество ридов улучшилось. Особенно на поледних 10 позициях, где качество обычно начиниет падать.
Картирование чтений
| Картирование: | hisat2-build chr10.fasta chr10 |
| Выравнивание: | hisat2 --no-softclip --no-spliced-alignment -x chr10 -U chr10_out.fastq -S chr10_ali.sam |
| Перевод в бинарный формат: | samtools view chr10_ali.sam -b -o chr10_ali.bam |
| Сортировка: | samtools sort -T /tmp/chr10_sorted -o chr10_sorted.bam chr10_ali.bam |
| Индексирование: | samtools index chr10_sorted.bam |
| Информация о картировании: | samtools idxstats chr10_sorted.bam |
| В результате оказалось 10398 картированых ридов и 128 некартированых. | |
Анализ SNP
| Создание файла с полиморфизмами: | samtools mpileup -uf chr10.fasta -o poly.bcf chr10_sorted.bam |
| Создание файла со списком отличий: | bcftools call -cv poly.bcf -o poly.vcf |
| Перевод в формат annovar: (Удаление Инделей) | convert2annovar.pl -format vcf4 poly.vcf > snp.avinput |
Всего 57 SNP и 9 Инделей.
Как качество, так и покрытие сильно колеблются.
Примеры Полиморфизмов:
Как качество, так и покрытие сильно колеблются.
Примеры Полиморфизмов:
- 1) 5.804.633 - Вставка; Покрытие - 119; Качество - 217.468
Ref: -
Alt: TC - 2) 5.805.087 - Делеция; Покрытие - 37; Качество - 217.468
Ref: CA
Alt: - - 3) 63.958.112 - Замена или SNP; Покрытие - 75; Качество - 225.009
Ref: T
Alt: C
| Аннотация RefgGene. Группа полиморфизма. Ген. | annotate_variation.pl -out snp.refgene -build hg19 snp.avinput /nfs/srv/databases/annovar/humandb.old/ |
| Аннотация DBSnp. Разделение по наличию rs. | annotate_variation.pl -filter -out snp.dbsnp -build hg19 -dbtype snp138 snp.avinput /nfs/srv/databases/annovar/humandb.old/ |
| Аннотация 1000 genomes. Частоты аллелей. | annotate_variation.pl -filter -out snp.1000g -build hg19 -dbtype 1000g2014oct_all snp.avinput /nfs/srv/databases/annovar/humandb.old/ |
| Аннотация GWAS. Ассоциации с болезнями. | annotate_variation.pl -regionanno -out snp.gwas -build hg19 -dbtype gwasCatalog snp.avinput /nfs/srv/databases/annovar/humandb.old/ |
| Аннотация ClinVar. | annotate_variation.pl -filter -out snp.clinvar -build hg19 -dbtype clinvar_20140211 snp.avinput /nfs/srv/databases/annovar/humandb.old/ |
Результаты Аннотаций:
- RefgGene:
SNP поделились на группы: intronic - 45; exonic - 10; URT3 - 2.
SNP попали в гены: FAM208B, RTKN2, CASP7 - DBSnp:
54 имеют rs, 3 нет. - 1000 genomes:
Среди SNP встретились самые разные частоты от 0.997604 до 0.000798722 - GWAS:
Было проаннотированно 3 SNP, связанные с болезнями: Остеосаркома, Ревматоидный артрит, Витилиго. - ClinVar:
Не дала результата