Определите таксономию и функцию прочтённой вами нуклеотидной последовательности (из практикума 6)
Исходная последовательность
 Если ориентироваться на число гомологов, то с большой вероятностью можно говорить о
семействе Terebellidae.
Если ориентироваться на число гомологов, то с большой вероятностью можно говорить о
семействе Terebellidae.
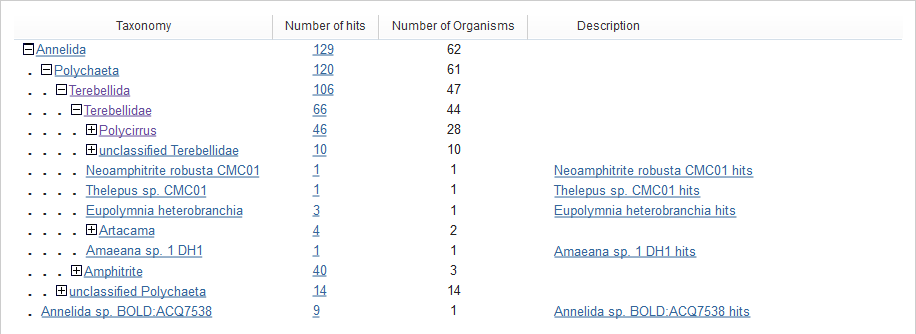 И с достаточно большой о роде Polycirrus. Этот же вывод можно сделать, взглянув на
список самых схожих последовательностей.
И с достаточно большой о роде Polycirrus. Этот же вывод можно сделать, взглянув на
список самых схожих последовательностей.
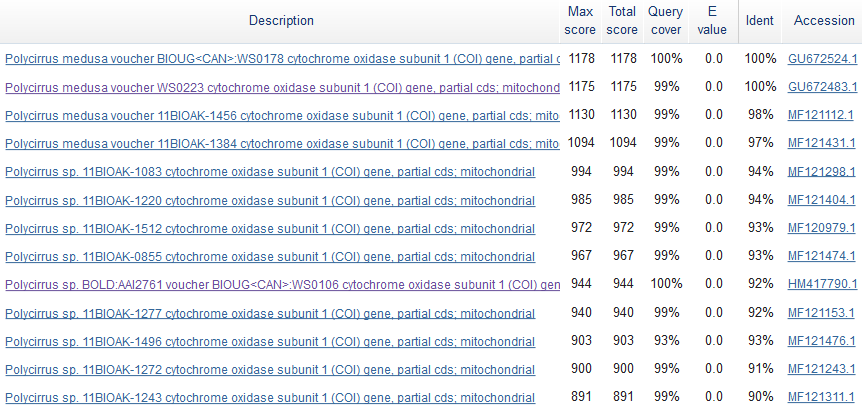
Как видно, все эти последовательности кодируют ген Сytochrome oxidase subunit 1 (COI), кодирующий белок Цитохром c-оксидазу.



Как видно, все эти последовательности кодируют ген Сytochrome oxidase subunit 1 (COI), кодирующий белок Цитохром c-оксидазу.
Сравните списки находок нуклеотидных последовательностей тремя разными вариантами blast
Megablast

Blastn (по умолчанию)

Blastn (Word size - 7, Match/Missmatch scores - 4, -5)
 Первое отличие появляется в списке Megablast двадцать первым номером. Это происходит в
тот момент, когда у находок резко падает Query cover и Ident.
Первое отличие появляется в списке Megablast двадцать первым номером. Это происходит в
тот момент, когда у находок резко падает Query cover и Ident.


Это находки под номерами с 34 по 37 двух вариаций Blastn. Тут различие появляется в тот же момент, что и в случае с Megablast.

Blastn (по умолчанию)

Blastn (Word size - 7, Match/Missmatch scores - 4, -5)



Это находки под номерами с 34 по 37 двух вариаций Blastn. Тут различие появляется в тот же момент, что и в случае с Megablast.
Проверьте наличие гомологов трех белков в неаннотированном геноме
TERT_SCHPO
Использовалась программа tblastn. В геноме Amoeboaphelidium protococcarum было найдено 5 похожих участков, из которых приемлемый E-value и Bit score у двух. Однако Identity у них порядка 25% и Positives около 47%. Этот результат я бы назвал условно положительным.
PRPC_EMENI
В этот раз было найдено 6 похожих участков, у всех хороший E-value, но Bit Score высокий у двух. В первом случае 56% Identity и 72% Positives, а во втором - 56% и 71%. Оба результата с натяжкой можно назвать положительными.
TBB_NEUCR
11 находок, все с хорошим E-value, 6 с неплохим Bit score, однако из них хочется выделить две с E-value близким к 0 и Bit Score большим остальных более чем вдвое. Первая с 82% Identity и 88% Positives и вторая с 87% и 94%. Это определённо гомологи.
Использовалась программа tblastn. В геноме Amoeboaphelidium protococcarum было найдено 5 похожих участков, из которых приемлемый E-value и Bit score у двух. Однако Identity у них порядка 25% и Positives около 47%. Этот результат я бы назвал условно положительным.
PRPC_EMENI
В этот раз было найдено 6 похожих участков, у всех хороший E-value, но Bit Score высокий у двух. В первом случае 56% Identity и 72% Positives, а во втором - 56% и 71%. Оба результата с натяжкой можно назвать положительными.
TBB_NEUCR
11 находок, все с хорошим E-value, 6 с неплохим Bit score, однако из них хочется выделить две с E-value близким к 0 и Bit Score большим остальных более чем вдвое. Первая с 82% Identity и 88% Positives и вторая с 87% и 94%. Это определённо гомологи.
Найдите какой-нибудь ген белка в одном из контигов
Был выбран контиг длины 12763 с названием unplaced-5.
Blast выдал множество совпадений промежутка 3254-4608 с геном, кодирующим альфа тубулин.
Blast выдал множество совпадений промежутка 3254-4608 с геном, кодирующим альфа тубулин.
Карта локального сходства геномов двух бактерий
Карта локального сходства геномов Chlamydia trachomatis и Chlamydia avium.
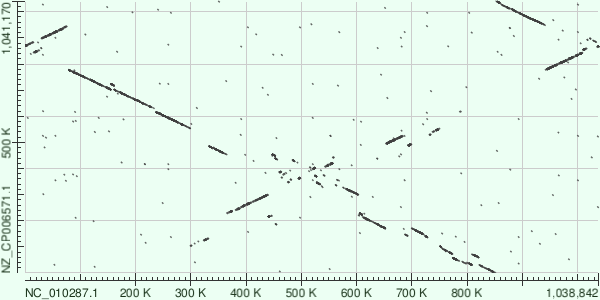 Как видно в геномах есть множество длинных похожих участков, но они расположены
нелинейно, а некоторые ещё и инвертированы.
Как видно в геномах есть множество длинных похожих участков, но они расположены
нелинейно, а некоторые ещё и инвертированы.
