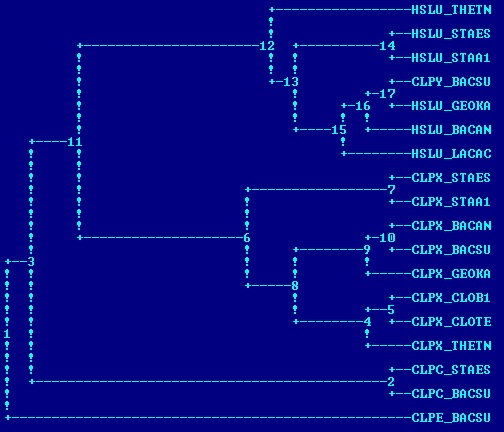
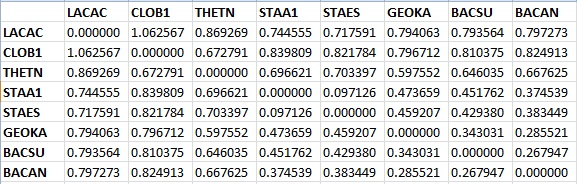
как часть пакета EMBOSS (неукорененное!):
| Название | Мнемоника |
| Clostridium botulinum | CLOB1 |
| Thermoanaerobacter tengcongensis | THETN |
| Lactobacillus acidophilus | LACAC |
| Bacillus anthracis | BACAN |
| Bacillus subtilis | BACSU |
| Geobacillus kaustophilus | GEOKA |
| Staphylococcus aureus | STAA1 |
| Staphylococcus epidermidis | STAES |
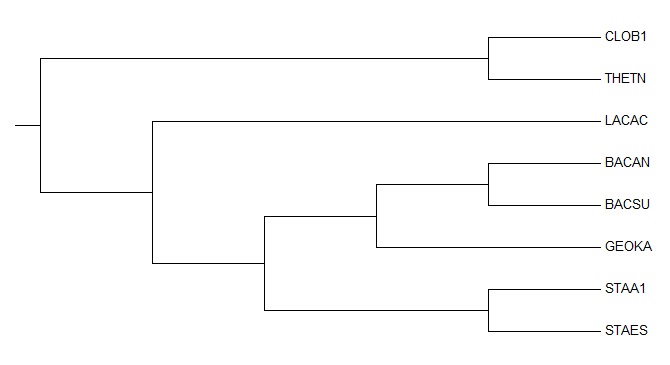
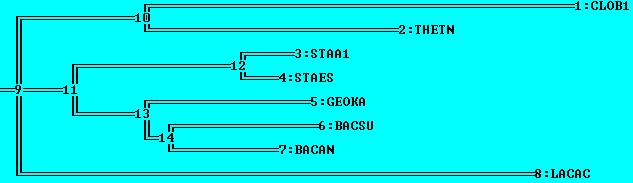
((((STAA1,STAES),((BACAN,BACSU),GEOKA)),LACAC),(CLOB1,THETN));

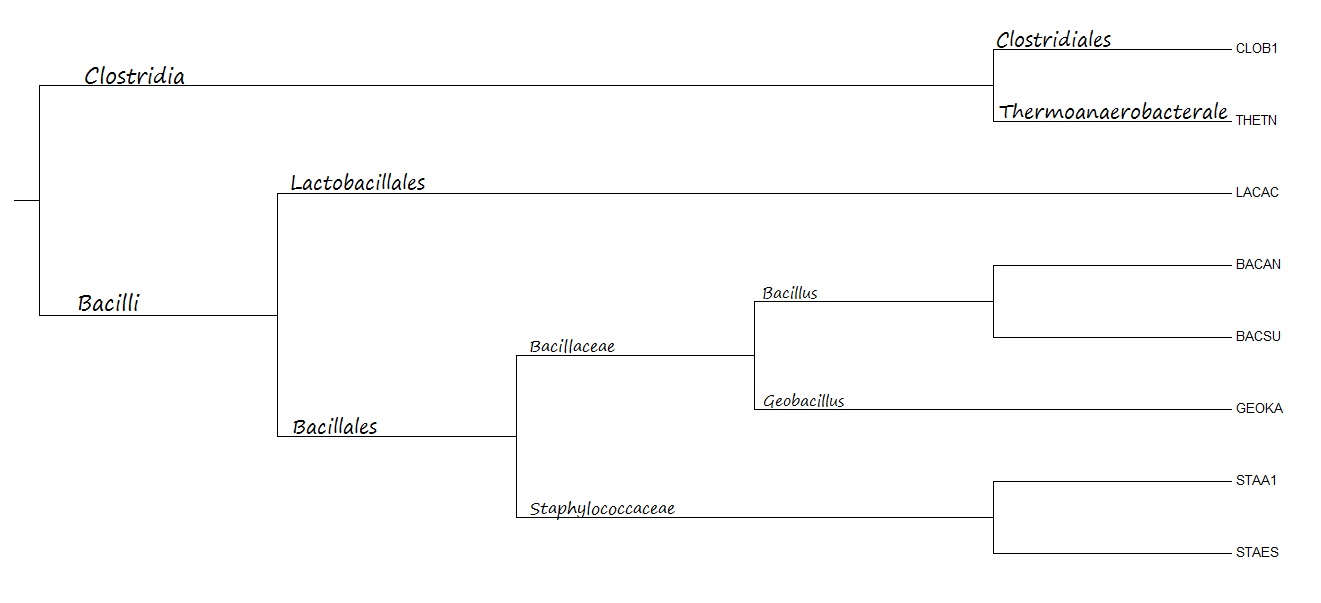
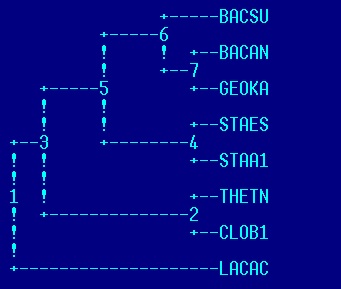
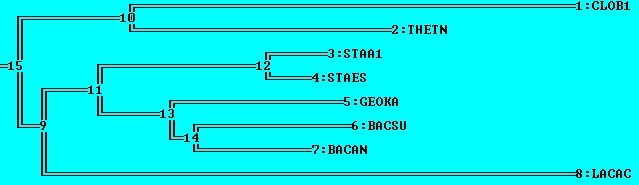
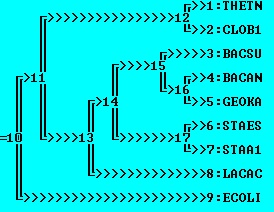
| Дерево, построенное программой fprotpars, установленой на сервере kodomo как часть пакета EMBOSS (неукорененное!): | Правильное дерево: |
 |  |
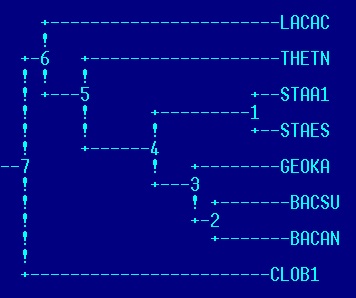
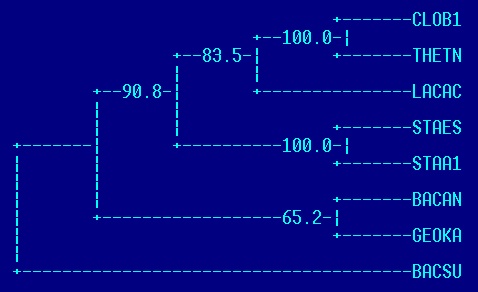
| алгоритм UPGMA | алгоритм Neighbor-Joining |
 | 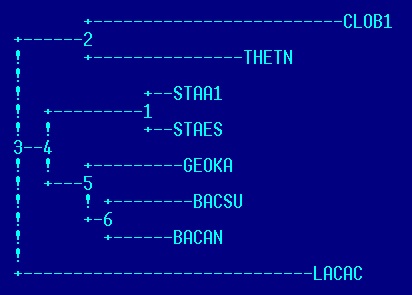 |
| в отличие от правильного дерева, отсутствует ветвь {CLOB1, THETN} vs {LACAC, BACAN, BACSU, GEOKA, STAA1, STAES} (та, в которую должно быть укоренено дерево), но присутствует ветвь {CLOB1, LACAC} vs {THETN, BACAN, BACSU, GEOKA, STAA1, STAES} | неукорененное дерево; все ветви совпадают с ветвями правильного дерева |
| алгоритм предполагает молекулярные часы; длины ветвей можно интерпритировать как время; | алгоритм не предполагает молекулярные часы; длины ветвей отражают число мутаций, произошедших на пути от общего предка; |
 .
.
 .
.
| Бактерия | AC записи EMBL, описывающей полный геном бактерии | Фрагмент генома бактерии, кодирующий 16S rRNA (все фрагменты лежат на цепях, выбранных для записи) |
| CLOB1 | CP000726 | 9282..10783 |
| THETN | AE008691 | 53858..55384 |
| LACAC | CP000033 | 59255..60826 |
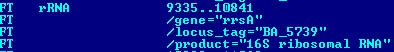
| BACAN | AE016879 | 9335..10841 |
| BACSU | AL009126 | 9810..11364 |
| GEOKA | BA000043 | 10421..11973 |
| STAA1 | AP009324 | 531922..533476 |
| STAES | AE015929 | 1598006..1599559 |