В файле 1ffy.pdb описана тРНК из Staphylococcus aureus с идентификатором цепи T.
Всего в файле 1ffy.pdb описаны 2 молекулы:
- Изолейцин-тРНК синтетаза (SYNONYM: ISOLEUCINE--TRNA LIGASE, ILERS), цепь A, идентификатор EC: 6.1.1.5 (белок был синтезирован);
- Изолейциновая тРНК, цепь T (была синтезирована)
G G G C U U G U A G C U C A G G U G G U U A G A G C G C A C C C C U G A U A A G G G U G A G G U C G G U G G U U C A A G U C C A C U C A G G C C C A CМодифицированных оснований нет. Имеется вставка: 21A, вставлен дезоксиробоаденозинмонофосфат. Нумерация нуклетидов начинается с 7409 и заканчивается номераом 9011.
find_pair -t 1ffy1.pdb stdout | analyzeСреди выданных программой файлов находился скрипт col_helices.scr для выделения различных α-спирлей структуры.
В итоге в структуре оказалось 4 спирали.
|
Участки, входящие в разные спирали.
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 G G G C U U G U A G C U C A G G U G G U 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 U A G A G C G C A C C C C U G A U A A G 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 G G U G A G G U C G G U G G U U C A A G 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 U C C A C U C A G G C C C A CКрасным покаршена спираль I, зеленым - II, синим - III и желтым - IV. |
Изображение построено на основе файла 1ffy1.pdb, содержащего только структуру тРНК.
Последовательность команд, использованная для создания картинки
select all backbone 150 wireframe off select 2:T and *.P label G 2 chain T/5' konech select 74:T and *.P label C 74 chain T/3' konech define helix1 1-7:T, 49-55:T, 18:T, 58:T, 61-72:T define helix2 36:T, 38-44:T, 10-15:T, 48:T, 8:T, 22-33:T define helix3 16-17:T define helix4 19:T, 56:T select helix1 color red select helix2 color green select helix3 color blue select helix4 color yellow |
Изображение цепи тРНК
|
Красным покаршена спираль I, зеленым - II, синим - III и желтым - IV |
- Пример внеспирального стекинг-взаимодействия между основаниями

Взаимодействующие основания подписаны. На изображении выделено 2 остатка U 21 chain T, один из них участвует во внеспиральном стекинг-взаимодействии, второй нет, но обратиться к ним отдельно не получилось.
- Пример водородных связей между основаниями, не сводящийся к Уотсон-Криковскому спариванию комплементарных оснований.
Данные из файла 1ffy.out. '*' обозначены не сводящийся к Уотсон-Криковскому спариванию комплементарных оснований.
Strand I Strand II 5 (0.007) T:...5_:[..U]U-*---G[..G]:..68_:T (0.009) ... 8 (0.006) T:..49_:[..G]G-*---U[..U]:..65_:T (0.005) ... 14 (0.008) T:..55_:[..U]Ux**+xG[..G]:..18_:T (0.011) 15 (0.011) T:..36_:[..U]Ux**+-U[..U]:..33_:T (0.005) 16 (0.013) T:..38_:[..A]A-*---C[..C]:..32_:T (0.003) ... 22 (0.009) T:..44_:[..A]Ax*---G[..G]:..26_:T (0.011) ... 29 (0.005) T:..16_:[..G]Gx**-xU[..U]:..17_:T (0.003)

Пример не Вотсон-Криковского взаимодействия между остатками U 5 chain T и G 68 chain T.
| Спираль I | Вид сверху

|
Длины бороздок:
Длина большой бороздки 11.446 (C64T.P-G2T.P) Длина малой бороздки 16.301 (U54T.P-C64T.P) |
| Спираль II | Вид сверху

|
Длины бороздок:
Длина большой бороздки 9.080 (C25T.P-G40T.P) Длина малой бороздки 17.946 (A14T.P-C25T.P) |
Как визуально, так и по данным длин малой и большой бороздок двух спиралей тРНК видно, что обе спирали похожи на А-форму ДНК.
Программа запускалась с параметром Minimum score threshold 10, так как при значениях параметра 50, 40 и 30, программа не давала никакого результата. По выданным прграммой выравниваниям можно предположить возможные двуспиральные участки в последовательности исследуемой РНК. Было получено 2 выранивания между 23-32м и 49-40м основаними и 1-7м и 62-57м основаними.
seq: Score 16: 8/10 ( 80%) matches, 0 gaps
23 gagcgcaccc 32
|| | |||||
49 ctggagtggg 40
seq: Score 14: 6/7 ( 85%) matches, 0 gaps
1 gggcttg 7
|| ||||
63 cctgaac 57
Участки 23-32 и 1-7 целиком входят во вторую и первую спирали соответсвенно.
Участок 40-49 входит в спираль 2 (40-44, 48) и спираль 1 (49).
Поэтому лишь часть первого выравнивания соответсвует полученным ранее данным.
Участок 57-63 входит в спираль 1 (61-63), поэтому часть второго выравнивания
соответсвует данным о спиралях исследуемой тРНК.
В общем можно сказать, что программа не очень хорошо справилась с задачей
по поиску спиралей с заданной тРНК, но и сказать, что результатов никаких нет нельзя.
Скорее всего это обусловлено в первую очередь сложностью структуры.
Так же на результативность выравнивания повлияли внеспиральные стекинг-взаимодействия между основаниями.
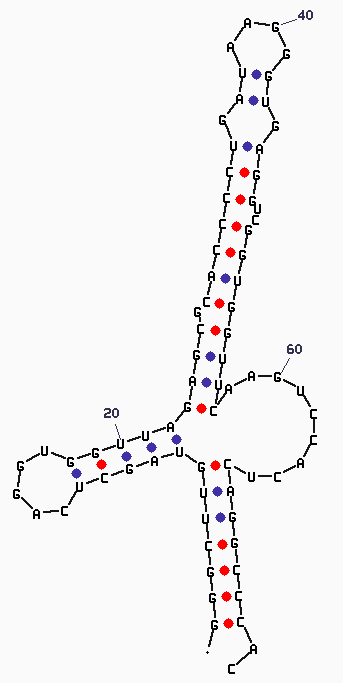
mfold SEQ=1ffy.fasta P=10При изменениях параметра P количество изображений на выходе не менялось. Всего получено 7 различных вторичны структур. Максимально близкое к реальности, то есть к виду клеверного листа, оказалось 6е изображение.