Практикум 8
Поиск в геноме Caenorhabditis elegans гена, кодирующий δ-субъединицу АТФ-синтазы
В практикуме использовалась геномная сборка Caenorhabditis elegans. С помощью текстового поиска была найдена δ-субъединица АТФ-синтазы (идентификатор: CCD72069.1) в файле с последовательностями белков, который был скачан ранее в практикуме 7.
Ссылка на последовательность белкаИдентификатор нуклеотидной записи гена, кодирующий данный белок: F58F12.1
Ссылка на файл с последовательностью кодирующей белок частиГен расположен на 2 хромосоме.

Варианты BLAST
В данном задании были использованы разные варианты BLAST для поиска гомологов среди последовательностей геномов семейства Собачьих (Canidae) для δ-субъединицы АТФ-синтазы из Caenorhabditis elegans (Caenorhabditis elegans относится к первичноротым, поэтому для поиска BLAST будем использовать удаленное от исходного организма семейство среди вторичноротых - Собачьи (Canidae)).
В NCBI Datasets для запроса Canidae было найдено 8 сборок геномов, включеных в RefSeq.
В качестве базы данных для поиска был выбран refseq_genomes.
Поиск гомологов с использованием blastnВсе параметры были использованы по умолчанию. В результате ничего не было найдено (также и при изменении параметра длины слова). Это связано с тем, что blastn сравнивает последовательности только на нуклеотидном уровне и не учитывает вырожденность генетического кода. Однако, в процессе эволюции между двумя гомологичными генами происходит множество синонимичных замен, которые в свою очередь снижают счет сходства в blastn.
Megablast в данном случае тоже некорректно использовать, так как он служит для поиска почти идентичных последовательностей.
Поиск гомологов с использованием tblastntblastn берет на вход последовательность белка и по ней ищет гомологи в транслированной нуклеотидной базе данных.
Как и ожидалось, было найдено 8 гомологов. Однако в поле Database - 7 сборок, использовавшихся для поиска. При уменьшении длины слова до 2, нашлось ещё 5 последовательностей, среди которых один новый вид и 4 последовательности из других хоромосом тех же видов. Значения Percentage Identity у этих гомолог ниже, e-value у трёх новых гомологов несколько выше остальных 1е-04 (по сравнению с 3е-18 и 1е-07), у других 5е-11 и 5е-05.
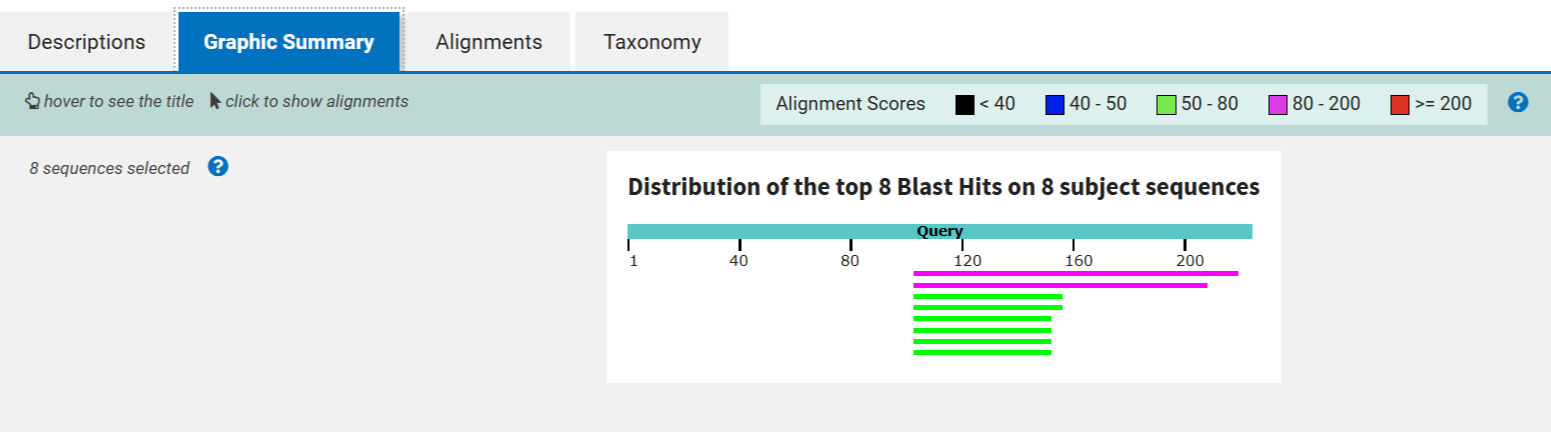
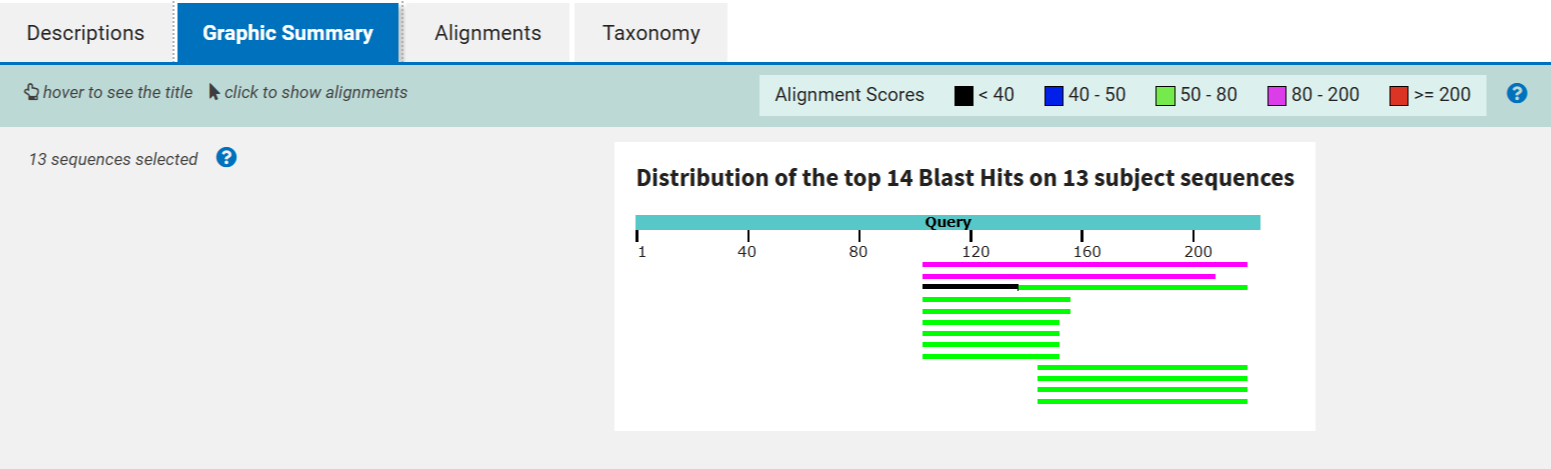
В отличие от tblastn, tblastx сравнивает все шесть возможных белковых последовательностей из нуклеотидного запроса со всеми шестью возможными белковыми последовательностями из базы данных. tblastx самый чувствительный из всех вариантов BLAST. Он способен найти гомологи, когда нуклеотидные последовательности очень сильно разошлись, или когда мы не уверены в правильной рамке считывания в запросе. Но в данном случае это не требуется, так как у нас уже есть белковая последовательность.
Поиск в геноме Caenorhabditis elegans генов основных рибосомальных РНК по далекому гомологу
Нужно найти в сборке генома Caenorhabditis elegans гены рРНК по далекому гомологу - генам рРНК из E.coli. Поиск проводился по последовательностям генов 23S и 16S рРНК E.coli. При инициации трансляции 16S рРНК способна комплементарно связываться с последовательностью Шайна-Дальгарно в мРНК прокариот, что обеспечивает начало трансляции с верного старт кодона. 23S рРНК - рибозим, катализирует пептидилтрансферазную реакцию.
Проиндексируем последовательности генома для работы локального BLAST
Команды для запуска локального поиска blastn отдельно для каждой рРНК:
В выравнивании для 16S 14 находок, для 23S -16. Только некоторые являются статистически значимыми. Для 16S e-value меньше единицы наблюдается у 9 находок, причём с учётом длины выравнивания (она должна быть не слишком маленкой) в качестве возможных гомологов могут подойти только 6 находок (BX284601.5). Если длина выравненных последовательностей крайне мала, то значения e-value будут высокие, что исключает гомологию. Для 23S e-value меньше единицы у 10 находок, но с учётом координат нам подходят только первые 8 (BX284601.5). Все последовательности генов рРНК находятся на 1 хромосоме.
Для того, чтобы определить гомологии нужно внимательно посмотреть на координаты статистически значимых находок (в случае 16S таких находок 6, для 23S их 8). Комплементарные участки должны находится на небольшом расстоянии друг от друга. Для 16S найдено 2 гомолога, для 23S их тоже 2.
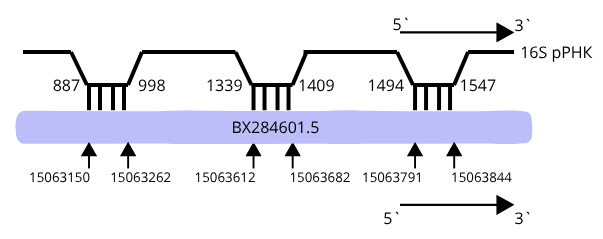
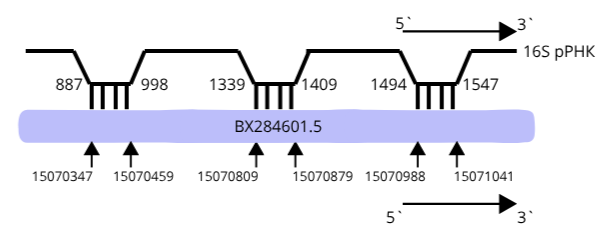
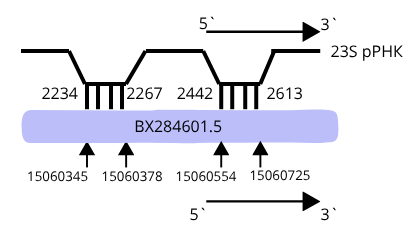
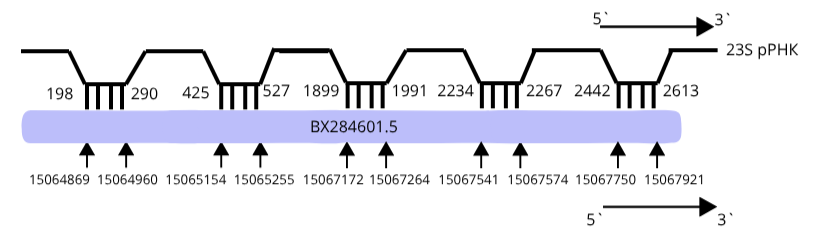
Карты локального сходства двух геномов
Для построения карты локального сходства с использованием Megablast и BLASTN были выбраны референсные геномы двух разных штаммов E.coli - Escherichia coli K-12 штамм MG1655 (NZ_CP169634.1) и Escherichia coli O157:H7 штамм EDL933 (NZ_CP028305.1).
Поисковые запросы в базе данных NCBI Nucleotides: (Escherichia coli K-12[Organism]) AND chromosome[Title]
(Escherichia coli O157:H7[Organism]) AND chromosome[Title]
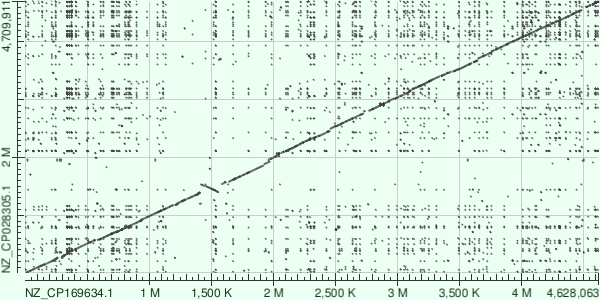
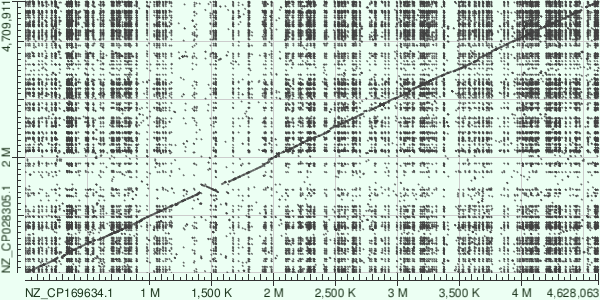
На картах видно, что в районе 1.5М нуклеотида произошла инверсия. Множество точек на карте, построенной blastn, означают наличие повторов и транспозонов в последовательностях.