Чтение последовательностей по Сэнгеру
Задание 1. Чтение последовательности ДНК на основании данных, полученных из капиллярного секвенатора по Сэнгеру.
Капиллярный секвенатор выдает файлы с хроматограммой и автоматически прочтённой последовательностью в формате .ab1. Вам дано два файла в формате .ab1, соответствующие прочтению прямой и обратной цепочки секвенируемой ДНК. См. список данных, файлы лежат на диске P в соответствующей директории: P:\y16\term3\block2\ab1_files Для просмотра хроматограмм и редактирования автоматического прочтения будем использовать программу Chromas (Lite). Ссылка на файл с результатом — последовательностью в fasta формате с выделенными строчными буквами проблемными нуклеотидами и полиморфизмами.
Ссылка на файл с прямой цепочкой секвенируемой ДНК (fasta)
Ссылка на файл с обратной цепочкой секвенируемой ДНК (fasta)
Ссылка на файл с прямой цепочкой секвенируемой ДНК в .ab1 формате
Ссылка на файл с обратной цепочкой секвенируемой ДНК в .ab1 формате
Рис.1. Выравнивание в JalView с выделенными проблемными участками

Выполненные исправления в прямой цепи:
1)№56: N=>y
2)№58: N=>a
3)№60: N=>t
4)№373: N=>g
5)№654: N=>y
6)№657: N=>y
Также стоит отметить большой пик: №54-№55 и расплывчатый пик №56.
Обоснования решений:
В позициях 55-58 образовался большой расплывчатый пик из-за которого нельзя однозначно определить нуклеотиды №56 и №58 по прямой цепи. В позиции 56 заметны 2 пика, чтобы понять какой именно нуклеотид находится в этом месте, пришлось сравнить это место с обратной последовательностью. В обратной последовательности эта позиция определяется однозначно - g. В позиции 58, если смотреть по пикам, то явно определяяется а, но так как совсем рядом идет пересечение, то лучше все равно убедиться в этом по обратному прочтению. Там тоже однозначно определяется а. С позицией 60 - аналогичная история: проверка по обратной последовательности. В позиции 373 шум выходит на уровень сигнала, поэтому лучше сделать в этом месте проверку. Находим соответствующий фррагмент на обратной последовательности и видим, что там в этом месте такой проблемы нет. Следовательно, можем однозначно определить.
Выполненные исправления в обратной цепи:
№597: N=>t
№379: N=>c
№51: N=>c
№33: N=>y
№36: N=>r
№39: N=>y
Здесь стоит упомянуть большой пик №638-№640.
(нумерация по соответствующей цепи)
Рис.2. Участок с проблемными нуклеотидами.
Рис.3. Участок с проблемными нуклеотидами.
Характеристика хроматограммы в целом: имеется всего несколько расплывчатых пиков и слабый шум,
который сильнее на обратной цепи.
2. Приведите пример нечитаемого фрагмента хроматограммы.
В задании требовалось рассмотреть нечитаемую хроматограмму.
Рис.3. Плохая хроматограмма
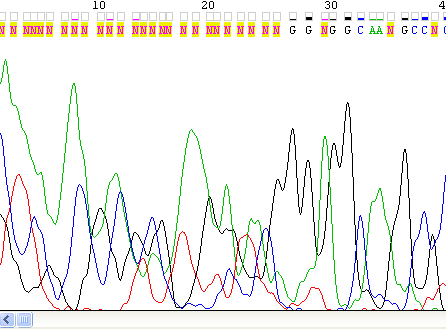
Здесь мы видим расплывчатые пики, это нехорошо. К тому же они наслаиваются друг на друга, высокий уровень шума, перекрывание - все
это не дает однозначной интерпретации хроматограммы. Возможно также попадание нескольких цепей в секвенатор. Возможно даже от разных организмов.
© Cherkashina Anastasia 2017