Занятие 12. Трансмембранные белки
1. База данных OPM
Исследуемые белки:
| 2RH1 и 5DQX |
Последовательность действий:
1. С помощью поиска по уровням классификации в базе данных OPM найдите любой белок, содержащий в трансмембранной части alpha-спирали.
Поищите какой-нибудь интересный! :)
2. Для этого белка определите:
☆ Толщину гидрофобной части белка в мембране: можно измерить в Jmol, если скачать с сайта представление белка
в мембране (правая кнопка мыши => measure => distance; в PDBTM надо фон сделать черным);
следите чтобы отрезок между атомами, выбранными для измерения, был перпендикулярен плоскости мембраны!
Можно использовать информацию о толщине гидрофобной части из БД OPM, но нужно понимать (и отвечать на коллоквиуме), откуда это число берется.
☆ Координаты трансмембранных спиралей (номера остатков, погруженных в мембрану, указаны в БД).
☆ Среднее количество остатков в одной трансмембранной спирали (или одном beta-тяже) белка.
☆ В какой мембране находится белок (например, "внешняя мембрана бактерии" и т.п.).
3. С помощью поиска по уровням классификации найдите любой белок, в трансмембранной части которого не альфа-спирали, а бета-листы.
Выполните для него действия из п. 2.
4. Полученные параметры для двух белков приведите на сайте в виде двух таблиц (в их заголовках укажите названия и идентификаторы белков).
5. Получите изображения обоих белков, где p-сторона мембраны будет направлена вверх, а вторичная структура будет хорошо видна.
Приведите изображения на странице с подписью, желательно расположить их под соответствующими таблицами, чтобы было сразу понятно, где какой белок.
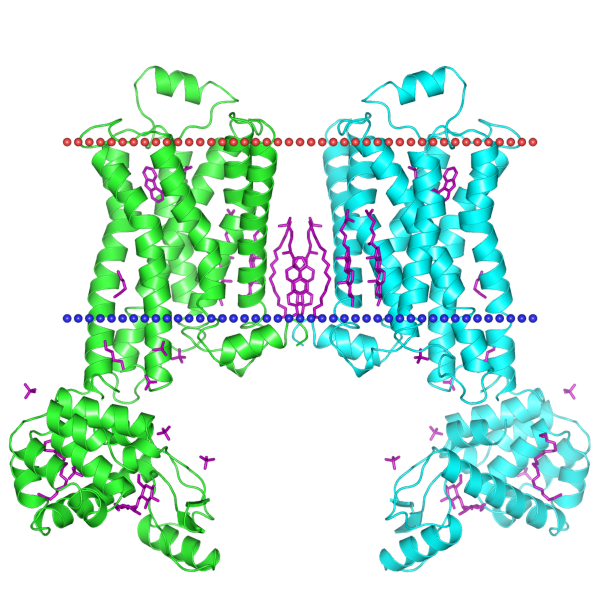
1. Трансмембранный белок с альфа-спиралями: 2rh1 (Beta-2 adrenergic receptor, inactive state, dimer) - Бета-2 адренорецептор.
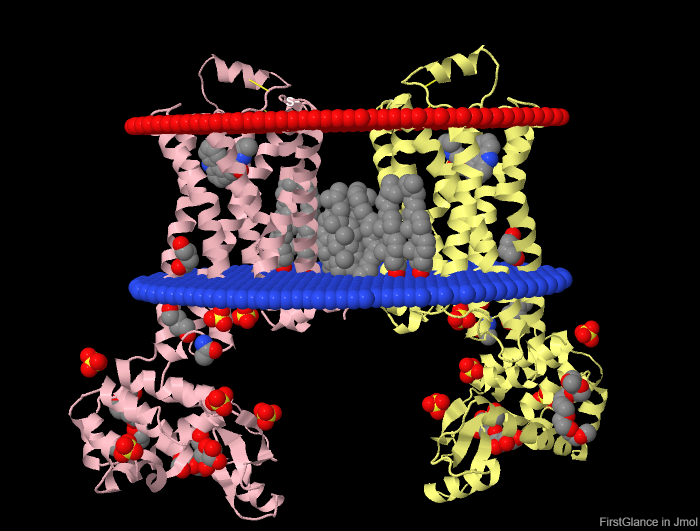
Рис.1 Изображение белка 2RH1 в Jmol
2RH1:
| Толщина гидрофобной части белка в мембране | 31.8 ± 0.9 A |
| Координаты трансмембранных спиралей | 1(31-56), 2(70-92), 3(104-129), 4(150-171), 5(198-220), 6(275-298), 7(306-327) |
| Среднее количество остатков в одной трансмембранной спирали белка | 22 |
| В какой мембране находится белок | in Eukaryotic plasma membrane |
Табл.1 Информация о белке 2RH1
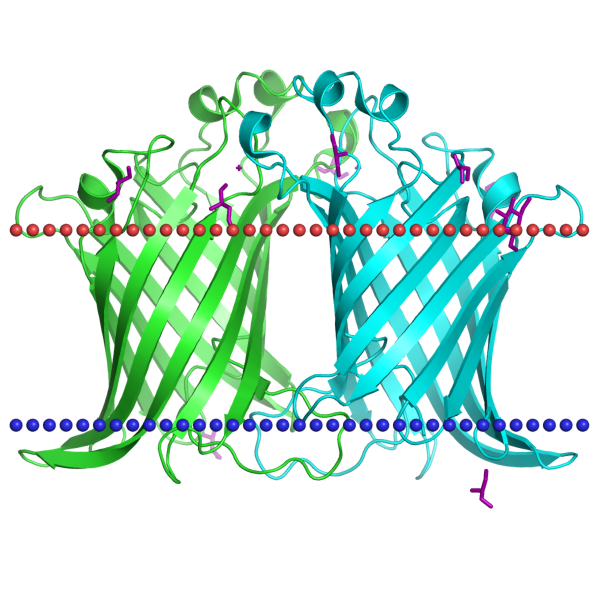
2. Трансмембранный белок с бета-тяжями: 5dqx (Outer membrane phospholipase A, dimer) - внешнемембранная фосфолипаза А.
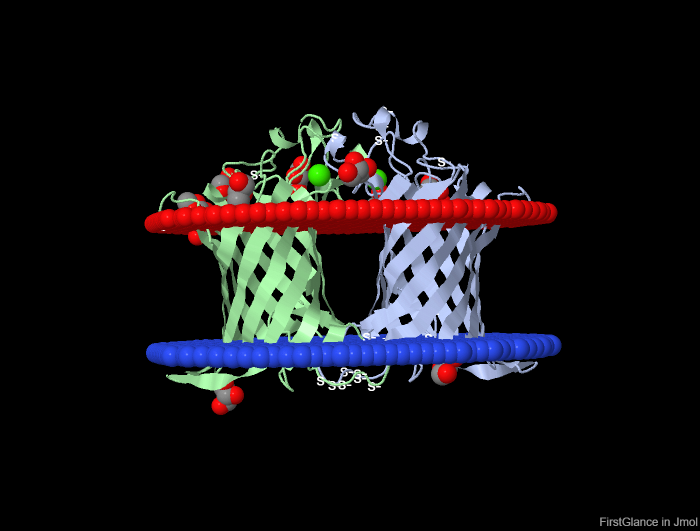
Рис.2 Изображение белка 5DQX в Jmol
5DQX:
| Толщина гидрофобной части белка в мембране | 23.4 ± 1.1 A |
| Координаты трансмембранных спиралей | 1( 53- 63), 2( 87- 98), 3( 108- 116), 4( 133- 142), 5( 155- 163), 6( 176- 184), 7( 189- 199), 8( 215- 223), 9( 225- 233), 10( 242- 249), 11( 257- 263), 12( 279- 287) |
| Среднее количество остатков в одном бета-тяже белка | 10 |
| В какой мембране находится белок | in Bacterial Gram-negative outer membrane |
Табл.2 Информация о белке 5DQX
2. Анализ предсказания трансмембранных спиралей
Цели задания: оценить корректность предсказания сервисов TMHMM и Phobius.
1. Запустите сервисы TMHMM TMHMM
и Phobius
для описанного вами в задании №1 альфа-спирального белка.
Сохраните результаты в графическом и текстовом видах для обоих программ. Приведите графики на сайте;
в подписи к рисункам опишите подробно, что показано по осям и что каким цветом окрашено.
2. На качественном уровне опишите, насколько совпадают результаты программ TMHMM и Phobius друг с другом и реальной структурной информацией из БД OPM (результаты задания №1). Есть ли спирали, полностью "не замеченные" предсказывающими программами или, наоборот, лишние предсказания?
3. (*, не обязательно) Опишите, в чем состоит различие алгоритмов TMHMM и Phobius. Пользуетесь обзорными статьями или статьями по данным программам.
Для определения альфа-спиралей в первом случае используем ТМНММ. При запуске программа выдала следующее:
Далее приведены таблица и гарфик ТМРММ:
Табл.3 Таблица, выданная программой ТМРММ
1-я строчка: длина белка
2-я строчка: число предсказанных альфа-спиралей
3-я строчка: ожидаемое число аминокислотных остатков, котрые входят в альфа-спираль
4-я строчка: ожидаемое число аминокислотных остатков, котрые входят в альфа-спираль, из первых 60-ти
5-я строчка: вероятность того, что N-конец уходит в цитоплазму
6-я строчка: возможность того, что N-конец является сигнальной последовательностью
Последующие строчки соотносят координаты белковой последвательности с принадлежностью (или непринадлежностью) к трансмембранной альфа-спирали.
Рис.3 График, выданный программой ТМРММ
На графике ось ОХ - координаты аминокислотных остатков белка, а OY - вероятность. Красная линия показывает вероятность оказаться трансмембранным участком, розовая - внешним, а синяя - внутренним. В целом, предсказание является довольно точным. ТМРММ правильно определили число трансмембранных участков и пости верно определил их местоположение.
Во втором случае используем Phobius.
При запуске программа выдала следующее:
Далее приведены таблица и гарфик Phobius:
Табл.4 Таблица, выданная программой Phobius
Phobius выдает таблицу, в которой показана разметка белка на участки: цитоплазматичекий и не цитоплазматический. Та пара чисел, которые расположены между ними - координаты трансмембранных участков.
Рис.4 График, выданный программой Phobius
Оси OX и OY аналогичны тем, что были в предыдущей программе. Серая область обозначает трансмембранный участок, зеленая линия - вероятность
данного фрагмента белка оказаться цитоплазматическим, а синяя - не цитоплазматическим.
Данное предсказание так же является очень точным. Предсказаны 7 трансмембранных участков, координаты которых указаны точно, только эти фрагменты
короче действительных.

3. База данных TCDB
Поиск прошел успешно только по первому белку 2RH1 - бета-2 адренорецептору.
ссылка
Идентификатор в БД - 9.A.14.3.5
В БД все белки поделены на суперсемьи (TC-superfamilies). Бета-2 адренорецеторы относятся к суперсемейству транспортер-опсин-G белок-связанных
рецепторов (ТОГ) - Transporter-Opsin-G protein-coupled receptor (TOG) Superfamily.
Идентификатор семейства белка:
9.A.14 - The G-protein-coupled receptor (GPCR) Family
9.A.14 The G-protein-coupled receptor (GPCR) Family
|
G-белок-связанные рецепторы (GPCR), представляют собой большое семейство, вовлеченное в различные типы путей передачи сигнала, вызванные гормонами, пептидами, белками и другими типами лигандов. Семейство настолько разнообразно, что многие члены не имеют видимого сходства, хотя все они пронизывают клеточную мембрану семь раз внеклеточным N- и цитозольным С-концом. Больше узнать про семейство можно здесь |
© Cherkashina Anastasia 2018

