
Семейство доменов названо по названию рибосомы, которая вступает в взаимодействие** с GTPase* белками.
у 13 неизвестна функция (PFAM ID начинается с DUF)
| Главная | Семестры | Проекты | Обо мне |
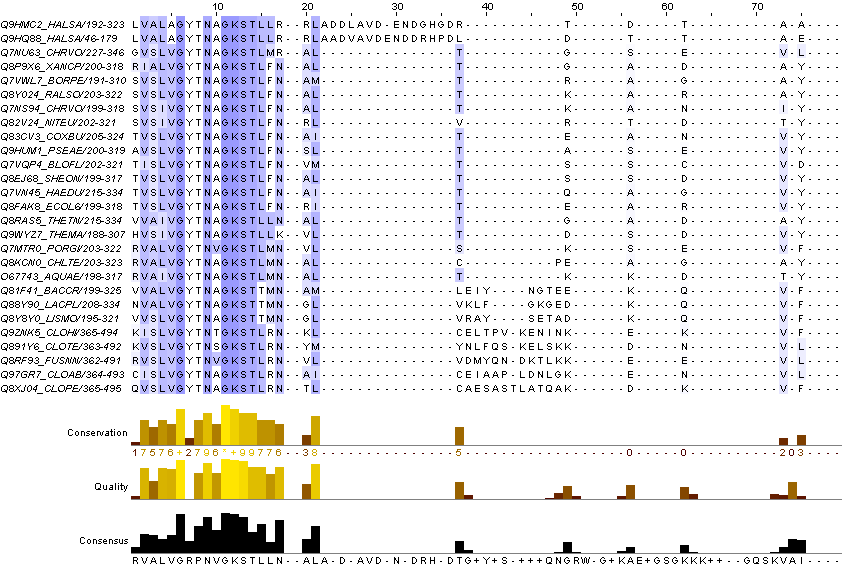
1.Опиcание доменной архитектуры белка engb_bacsu в соответствии с банком Pfam
Cхема из Pfam: |
|||||
| Пояснения к схеме |
|||||
| № | Pfam AC | Pfam ID | Полное название семейства доменов |
Положение в последовательности белка ENGB_BACSU | Клан |
| 1. | P38424 | ENGB | 50S рибсома-связка GTPase* (50S ribosome-binding GTPase*). Семейство доменов названо по названию рибосомы, которая вступает в взаимодействие** с GTPase* белками. |
25–143 | Клан P-loop_NTPase (CL0023), содержит 193 семейства,
у 13 неизвестна функция (PFAM ID начинается с DUF) |

|
Таксон
|
Количество белков с доменом PF06071.
|
Количество белков с доменом PF01926.
|
|
| Эукариоты | Зеленые растения | 59 | * |
| Грибы | 134 | * | |
| Животные | 184 | * | |
| Остальные эукариоты | 90 | * | |
| Археи | - | * | |
| Бактерии | 2893 | * | |
| Вирусы | - | * | |
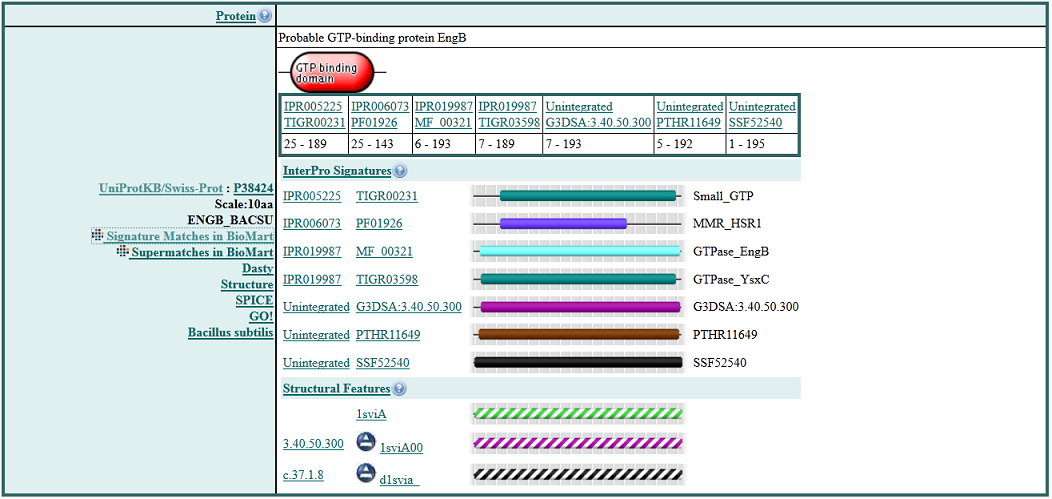
Представлены следующие структурные подписи:
1sviA PDB Chain
3.40.50.300 1sviA00 CATH Domain
c.37.1.8 d1svia_ SCOP Domain
Границы домена PF01926 не изменились по сравнению с Pfam, все так же 25 - 143.